Manuscrit Full.Pdf
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Supplemental Figure S1 Differentially Methylated Regions (Dmrs
Supplemental Figure S1 '$$#0#,2'**7+#2&7*2#"0#%'-,11 #25##,"'1#1#122#1 '!2-0'*"#.'!2'-,-$122,1'2'-,$0-+2- !"Q !"2-$%," $ 31',% 25-$-*" !&,%# ," ' 0RTRW 1 !32V-$$ !0'2#0'T - #.0#1#,22'-, -$ "'$$#0#,2'**7+#2&7*2#"%#,#11',.0#,2#1,"2&#'0 #&4'-022,1'2'-, #25##,"'$$#0#,2"'1#1#122#1T-*!)00-51',"'!2#&7.#0+#2&7*2#"%#,#1Q%0700-51 &7.-+#2&7*2#"%#,#1Q31',%25-$-*"!&,%#,"'0RTRW1!32V-$$!0'2#0'T-%#,#1 +#22&# -4#!0'2#0'22,1'2'-,$0-+$%2-$Q5#2�#$-0#*1-',!*3"#" %#,#15'2&V4*3#0RTRWT$$#!2#"%#,10#&'%&*'%&2#" 712#0'1)1#T Supplemental Figure S2 Validation of results from the HELP assay using Epityper MassarrayT #13*21 $0-+ 2&# 1$ 117 5#0# !-00#*2#" 5'2& /3,2'22'4# +#2&7*2'-, ,*78#" 7 '13*$'2#11007$-04V-,"6U-%#,#.0-+-2#00#%'-,1T11007 51.#0$-0+#"31',%**4'* *#1+.*#1T S Supplemental Fig. S1 A unique hypermethylated genes (methylation sites) 454 (481) 5693 (6747) 120 (122) NLMGUS NEWMM REL 2963 (3207) 1338 (1560) 5 (5) unique hypomethylated genes (methylation sites) B NEWMM 0 (0) MGUS 454 (481) 0 (0) NEWMM REL NL 3* (2) 2472 (3066) NEWMM 2963 REL (3207) 2* (2) MGUS 0 (0) REL 2 (2) NEWMM 0 (0) REL Supplemental Fig. S2 A B ARID4B DNMT3A Methylation by MassArray Methylation by MassArray 0 0.2 0.4 0.6 0.8 1 1.2 0.5 0.6 0.7 0.8 0.9 1 2 0 NL PC MGUS 1.5 -0.5 NEW MM 1 REL MM -1 0.5 -1.5 0 -2 -0.5 -1 -2.5 -1.5 -3 Methylation by HELP Assay Methylation by HELP Methylation by HELP Assay Methylation by HELP -2 -3.5 -2.5 -4 Supplemental tables "3..*#+#,2*6 *#"SS 9*','!*!&0!2#0'12'!1-$.2'#,21+.*#1 DZ_STAGE Age Gender Ethnicity MM isotype PCLI Cytogenetics -

Article Association of Systemic Lupus Erythematosus
Article Association of Systemic Lupus Erythematosus Susceptibility Genes with IgA Nephropathy in a Chinese Cohort Xu-Jie Zhou, Fa-Juan Cheng, Li Zhu, Ji-Cheng Lv, Yuan-Yuan Qi, Ping Hou, and Hong Zhang Abstract Background and objectives One hypothesis states that IgA nephropathy (IgAN) is a syndrome with an Renal Division, Peking University First autoimmune component. Recent studies strongly support the notion of shared genetics between immune-related Hospital; Peking diseases. This study investigated single-nucleotide polymorphisms (SNPs) reported to be associated with University Institute of systemic lupus erythematosus (SLE) in a Chinese cohort of patients with IgAN and in controls. Nephrology; Key Laboratory of Renal Disease, Ministry of Design, setting, participants, & measurements This study investigated whether SNP markers that had been Health of China; and reported to be associated with SLE were also associated with IgAN in a Chinese population. The study cohort Key Laboratory of consisted of 1194 patients with IgAN and 902 controls enrolled in Peking University First Hospital from 1997 to Chronic Kidney 2008. Disease Prevention and Treatment (Peking University), Ministry , 3 25 Results Ninety-six SNPs mapping to 60 SLE loci with reported P values 1 10 were investigated. CFH of Education, Beijing, 2 2 2 2 2 (P=8.41310 6), HLA-DRA (P=4.91310 6), HLA-DRB1 (P=9.46310 9), PXK (P=3.62310 4), BLK (P=9.32310 3), People’s Republic of and UBE2L3 (P=4.0731023) were identified as shared genes between IgAN and SLE. All associations reported China herein were corroborated by associations at neighboring SNPs. -
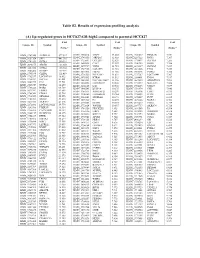
(A) Up-Regulated Genes in HCC827-GR-High2 Compared to Parental HCC827
Table S2. Results of expression profiling analysis (A) Up-regulated genes in HCC827-GR-high2 compared to parental HCC827 Fold Fold Fold Unique ID Symbol Unique ID Symbol Unique ID Symbol change* change* change* ILMN_1709348 ALDH1A1 577.587 ILMN_2310814 MAPT 13.003 ILMN_1741017 PIP4K2B 7.331 ILMN_1651354 SPP1 441.316 ILMN_1748650 MRPL45 12.988 ILMN_3237623 RNY1 7.297 ILMN_1701831 GSTA1 260.591 ILMN_1755897 UGT2B7 12.629 ILMN_1734897 SLC4A4 7.285 ILMN_1658835 CAV2 12.357 ILMN_1746359 RERG 7.280 ILMN_2094875 ABCB1 183.050 ILMN_1678939 VNN2 11.935 ILMN_1671337 SLC2A5 7.257 ILMN_3251540 GSTA2 145.982 ILMN_1729905 GAL3ST1 11.910 ILMN_1691606 LYG2 7.254 ILMN_2062468 IGFBP7 127.721 ILMN_1672536 FBLN1 11.716 ILMN_1785646 PMP22 7.246 ILMN_1795190 CLDN2 111.439 ILMN_1796339 PLEKHA2 11.631 ILMN_1737387 LOC728441 7.207 ILMN_1782937 LOC647169 98.612 ILMN_1676563 HTRA1 11.592 ILMN_1684401 FMO1 7.117 ILMN_1754247 SLC3A1 81.001 ILMN_3263423 LOC100129027 11.346 ILMN_1687035 ADAMTSL4 7.098 ILMN_1662795 CA2 79.581 ILMN_1694898 LOC653857 10.906 ILMN_2153572 MAGEA3 7.086 ILMN_2168747 GSTA2 66.250 ILMN_2404625 LAT 10.560 ILMN_1784283 USH1C 7.079 ILMN_1764228 DAB2 64.709 ILMN_1666546 DUSP14 10.375 ILMN_1731374 CPE 7.046 ILMN_1675797 EPDR1 63.605 ILMN_1764571 ARHGAP23 10.299 ILMN_1765446 EMP3 6.933 ILMN_1708341 PDZK1 59.714 ILMN_3200140 LOC645638 10.284 ILMN_1754002 IL1F8 6.863 ILMN_1713529 SEMA6A 52.575 ILMN_3244343 SNORA21 10.171 ILMN_1878007 FUT9 6.835 ILMN_1708391 NR1H4 43.218 ILMN_1671489 PC 10.075 ILMN_1699208 NAP1L1 6.763 ILMN_2412336 AKR1C2 42.826 ILMN_2404688 -

1 SUPPLEMENTAL DATA Figure S1. Poly I:C Induces IFN-Β Expression
SUPPLEMENTAL DATA Figure S1. Poly I:C induces IFN-β expression and signaling. Fibroblasts were incubated in media with or without Poly I:C for 24 h. RNA was isolated and processed for microarray analysis. Genes showing >2-fold up- or down-regulation compared to control fibroblasts were analyzed using Ingenuity Pathway Analysis Software (Red color, up-regulation; Green color, down-regulation). The transcripts with known gene identifiers (HUGO gene symbols) were entered into the Ingenuity Pathways Knowledge Base IPA 4.0. Each gene identifier mapped in the Ingenuity Pathways Knowledge Base was termed as a focus gene, which was overlaid into a global molecular network established from the information in the Ingenuity Pathways Knowledge Base. Each network contained a maximum of 35 focus genes. 1 Figure S2. The overlap of genes regulated by Poly I:C and by IFN. Bioinformatics analysis was conducted to generate a list of 2003 genes showing >2 fold up or down- regulation in fibroblasts treated with Poly I:C for 24 h. The overlap of this gene set with the 117 skin gene IFN Core Signature comprised of datasets of skin cells stimulated by IFN (Wong et al, 2012) was generated using Microsoft Excel. 2 Symbol Description polyIC 24h IFN 24h CXCL10 chemokine (C-X-C motif) ligand 10 129 7.14 CCL5 chemokine (C-C motif) ligand 5 118 1.12 CCL5 chemokine (C-C motif) ligand 5 115 1.01 OASL 2'-5'-oligoadenylate synthetase-like 83.3 9.52 CCL8 chemokine (C-C motif) ligand 8 78.5 3.25 IDO1 indoleamine 2,3-dioxygenase 1 76.3 3.5 IFI27 interferon, alpha-inducible -

A Novel Mirna Cluster Within the Circadian Clock Gene NPAS2 and the Implications of Rs1811399, an Autism Enriched Single Nucleot
A novel miRNA cluster within the circadian clock gene NPAS2 and the implications of rs1811399, an autism enriched single nucleotide polymorphism. Thesis submitted in accordance with the requirements of Bangor University for the degree of Doctor in Philosophy Dylan Wyn Jones Bangor University, UK School of Biological Sciences December 2014 ACKNOWLEDGMENTS I would like to extend my deepest thanks to Dr Thomas Caspari for taking on this difficult project and entrusting me to deliver the result. I am grateful for all his work and insight into the scientific process. I would also care to extend my gratitude to Dr Natalia Harrison who trained me during the first few months in the intricacies of tissue culture. I would also like to commend Dr Brad Nicholas and Dr Dawn Wimpory for their invaluable insights and assistance throughout the project. Without their prior work in the field I would not have had these three years. Thanks also go to the Knowledge Economy Skills Scholarship (KESS) and Autism Cymru for funding this research project. ************ Buaswn yn hoffi diolch, a cyflwyno, yr gwaith yma I Bethan Davies-Jones fy annwyl wraig. Dros yr tair blynedd ddweutha rwyt wedi bod yn gefn i mi ac yn ysbridoliaeth. Nid wyf yn deud gormod wrth ddweud na hebdda ti ni buaswn wedi cyraedd yr lle rwyf wedi. I fy nheulu am yr holl cefnogaeth drost y blynyddoedd. I Mam a Dad am fy magu ac I Endaf, fy mhrawd. Hefyd mewn côf o Nain a Taid Rhos-y-bôl a Taid Tŷ Croes. Effallai nid ydych yma i weld diwedd fy nhaith drwyr brif ysgol ond mae’r cofion melus yn ein cadw ni yn fynd. -

Transcriptomic Analysis of Patients with Tetralogy of Fallot Reveals the Effect of Chronic Hypoxia on Myocardial Gene Expression
Ghorbel et al Congenital Heart Disease Transcriptomic analysis of patients with tetralogy of Fallot reveals the effect of chronic hypoxia on myocardial gene expression Mohamed T. Ghorbel, PhD, Myriam Cherif, PhD, Emma Jenkins, PhD, Amir Mokhtari, MRCS, Damien Kenny, MRCPCH, Gianni D. Angelini, FRCS, and Massimo Caputo, MD Objectives: In cyanotic patients undergoing repair of heart defects, chronic hypoxia is thought to lead to greater susceptibility to ischemia and reoxygenation injury. We sought to find an explanation to such a hypothesis by investigating the cardiac gene expression in patients with tetralogy of Fallot undergoing cardiac surgery. CHD Methods: The myocardial gene profile was investigated in right ventricular biopsy specimens obtained from 20 patients with a diagnosis of cyanotic (n ¼ 11) or acyanotic (n ¼ 9) tetralogy of Fallot undergoing surgical repair. Oligonucleotide microarray analyses were performed on the samples, and the array results were validated with Western blotting and enzyme-linked immunosorbent assay. Results: Data revealed 795 differentially expressed genes in cyanotic versus acyanotic hearts, with 198 upregu- lated and 597 downregulated. Growth/morphogenesis, remodeling, and apoptosis emerged as dominant func- tional themes for the upregulated genes and included the apoptotic gene TRAIL (tumor necrosis factor–related apoptosis-inducing ligand), the remodeling factor OPN (osteopontin), and the mitochondrial function gene COX11 (cytochrome-c oxidase 11). In contrast, transcription, mitogen-activated protein kinase signaling, and contractile machinery were the dominant functional classes for the downregulated genes, which included the calcium-handling gene NCX1 (sodium-calcium exchanger). Protein levels of COX11, NCX1, OPN, and LYZ (ly- sozyme) in the myocardium followed the same pattern obtained by means of transcriptomics. -
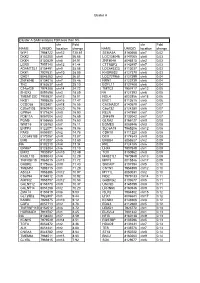
Copy of Supplementary Table 2
Cluster A Cluster A SAM analysis FDR less than 5% chr Fold chr Fold NAME UNIQID location change NAME UNIQID location change WIF1 7964722 chr12 130.61 SEMA3A 8140668 chr7 0.02 DKK2 8102200 chr4 56.66 LOC138046 8147065 chr8 0.03 DKK4 8150529 chr8 54.91 ZNF804A 8046815 chr2 0.03 LGR5 7957140 chr12 41.44 CTTNBP2 8142497 chr7 0.03 ADAMTSL1 8154491 chr9 35.08 LOC645323 8113037 chr5 0.03 DKK1 7927631 chr10 28.09 KHDRBS2 8127370 chr6 0.03 GAD1 8046283 chr2 26.81 LOC727966 8127399 chr6 0.04 ZNF804B 8134018 chr7 25.46 NRN1 8123739 chr6 0.04 TNC 8163637 chr9 25.10 EGFL11 8127408 chr6 0.04 C14orf29 7974288 chr14 24.72 TMTC2 7957417 chr12 0.05 SHOX2 8091698 chr3 18.89 NA 8127393 chr6 0.05 TMEM132C 7959827 chr12 18.01 NOL4 8022856 chr18 0.06 NKD1 7995525 chr16 17.47 ENC1 8112615 chr5 0.06 CCDC68 8023401 chr18 16.55 CACNA2D1 8140579 chr7 0.07 C20orf103 8060940 chr20 16.06 C6orf32 8124280 chr6 0.07 GPR64 8171624 chrX 15.90 RELN 8141950 chr7 0.07 PDE11A 8057004 chr2 15.69 ZNF679 8133042 chr7 0.07 PGM5 8155665 chr9 15.60 GLRA2 8166127 chrX 0.08 WNT16 8135763 chr7 15.11 EOMES 8085946 chr3 0.08 ENPP3 8122071 chr6 15.06 SLC6A15 7965206 chr12 0.08 PAX3 8059301 chr2 14.78 CDH18 8111220 chr5 0.08 LOC645188 8170257 chrX 13.87 DCX 8174543 chrX 0.08 EMB 8112007 chr5 13.60 ERBB4 8058627 chr2 0.09 NA 8102210 chr4 13.34 PRL 8124185 chr6 0.09 EPHA7 8128284 chr6 13.10 LHX4 7907849 chr1 0.09 EMX2 7930857 chr10 12.49 TOX 8150962 chr8 0.09 RASL11B 8095043 chr4 12.29 MAB21L1 7970949 chr13 0.09 TNFRSF19 7968015 chr13 11.72 MPP3 8015846 chr17 0.09 GABRE 8175666 chrX 11.40 -

STAT3 Targets Suggest Mechanisms of Aggressive Tumorigenesis in Diffuse Large B Cell Lymphoma
STAT3 Targets Suggest Mechanisms of Aggressive Tumorigenesis in Diffuse Large B Cell Lymphoma Jennifer Hardee*,§, Zhengqing Ouyang*,1,2,3, Yuping Zhang*,4 , Anshul Kundaje*,†, Philippe Lacroute*, Michael Snyder*,5 *Department of Genetics, Stanford University School of Medicine, Stanford, CA 94305; §Department of Molecular, Cellular, and Developmental Biology, Yale University, New Haven, CT 06520; and †Department of Computer Science, Stanford University School of Engineering, Stanford, CA 94305 1The Jackson Laboratory for Genomic Medicine, Farmington, CT 06030 2Department of Biomedical Engineering, University of Connecticut, Storrs, CT 06269 3Department of Genetics and Developmental Biology, University of Connecticut Health Center, Farmington, CT 06030 4Department of Biostatistics, Yale School of Public Health, Yale University, New Haven, CT 06520 5Corresponding author: Department of Genetics, Stanford University School of Medicine, Stanford, CA 94305. Email: [email protected] DOI: 10.1534/g3.113.007674 Figure S1 STAT3 immunoblotting and immunoprecipitation with sc-482. Western blot and IPs show a band consistent with expected size (88 kDa) of STAT3. (A) Western blot using antibody sc-482 versus nuclear lysates. Lanes contain (from left to right) lysate from K562 cells, GM12878 cells, HeLa S3 cells, and HepG2 cells. (B) IP of STAT3 using sc-482 in HeLa S3 cells. Lane 1: input nuclear lysate; lane 2: unbound material from IP with sc-482; lane 3: material IP’d with sc-482; lane 4: material IP’d using control rabbit IgG. Arrow indicates the band of interest. (C) IP of STAT3 using sc-482 in K562 cells. Lane 1: input nuclear lysate; lane 2: material IP’d using control rabbit IgG; lane 3: material IP’d with sc-482. -

The Characterization of Varicella Zoster Virus Specific T Cells in Skin and Blood During
The Characterization of Varicella Zoster Virus Specific T Cells In Skin and Blood During Ageing Milica Vukmanovic-Stejic1, Daisy Sandhu 1,2, Judith A. Seidel1, Neil Patel1,2, Toni O. Sobande 1, Elaine Agius1,2, Sarah E. Jackson1, Judilyn Fuentes-Duculan3, Mayte Suarez- Farinas 3, Neil A. Mabbott4, Katie E. Lacy5, Graham Ogg6 , Frank O Nestle5, James G. Krueger 3, Malcolm H.A. Rustin 2, Arne N. Akbar 1 1Division of Infection and Immunity, University College London, London, W1T 4JF, England, United Kingdom. 2 Department of Dermatology, Royal Free Hospital, London, NW3 2QG, England, United Kingdom. 3Laboratory for Investigative Dermatology, Rockefeller University, New York, NY 10021, USA 4The Roslin Institute and Royal (Dick) School of Veterinary Studies, University of Edinburgh, Easter Bush, Midlothian, EH25 9RG, UK 5 St. Johns Institute of Dermatology, Guys and St. Thomas' Hospital, London. 6 MRC Human Immunology Unit, University of Oxford, NIHR Biomedical Research Centre, Oxford, UK Corresponding author: Professor Arne N. Akbar, or Dr M Vukmanovic-Stejic, tel: +44-20-31082172/ 02031082173 E-mail: [email protected] or [email protected] 1 This work was funded by grants from the Medical Research Council, the Biotechnology and Biological Sciences Research Council, The British Skin Foundation and Dermatrust. Key words: T cell, memory, skin resident, antigen-specific, ageing Abbreviations: VZV (varicella zoster virus), Treg (regulatory T cells), CMV (cytomegalovirus), HSV (herpes simplex virus), Running title: Effects of age on VZV specific T cells in blood and skin 2 ABSTRACT The varicella-zoster virus (VZV) re-activation increases during ageing. Although the effects of VZV re-activation are observed in the skin (shingles) the number or functional capacity of cutaneous VZV specific T cells have not been investigated. -

Gene Mapping of Monogenic Disorders and Complex Diseases Via Genome Wide Association Studies Xia Zhao Iowa State University
Iowa State University Capstones, Theses and Graduate Theses and Dissertations Dissertations 2012 Gene mapping of monogenic disorders and complex diseases via genome wide association studies Xia Zhao Iowa State University Follow this and additional works at: https://lib.dr.iastate.edu/etd Part of the Animal Diseases Commons, and the Molecular Biology Commons Recommended Citation Zhao, Xia, "Gene mapping of monogenic disorders and complex diseases via genome wide association studies" (2012). Graduate Theses and Dissertations. 12878. https://lib.dr.iastate.edu/etd/12878 This Dissertation is brought to you for free and open access by the Iowa State University Capstones, Theses and Dissertations at Iowa State University Digital Repository. It has been accepted for inclusion in Graduate Theses and Dissertations by an authorized administrator of Iowa State University Digital Repository. For more information, please contact [email protected]. Gene mapping of monogenic disorders and complex diseases via genome wide association studies by Xia Zhao A dissertation submitted to the graduate faculty in partial fulfillment of the requirements for the degree of DOCTOR OF PHILOSOPHY Major: Genetics Program of Study Committee: Max Rothschild, Co-major Professor Dorian Garrick, Co-major Professor Susan Lamont N. Matthew Ellinwood Peng Liu Iowa State University Ames, Iowa 2012 Copyright © Xia Zhao, 2012. All rights reserved. ii TABLE OF CONTENTS LIST OF FIGURES iv LIST OF TABLES vi ABSTRACT vii CHAPTER1. GENERAL INTRODUCTION 1 INTRODUCTION 1 RESEARCH OBJECTIVES 2 DISSERTATION ORGANIZATION 2 LITERATURE REVIEW 4 REFERENCES 27 CHAPTER 2. A NOVEL NONSENSE MUTATION IN THE DMP1 GENE IDENTIFIED BY A GENOME-WIDE ASSOCIATION STUDY IS RESPONSIBLE FOR INHERITED RICKETS IN CORRIEDALE SHEEP 38 ABSTRACT 38 INTRODUCTION 39 RESULTS 41 DISCUSSION 44 MATERIALS and METHODS 47 REFERENCES 51 CHAPTER 3. -

MOL #82305 TITLE PAGE Title: Induced CYP3A4 Expression In
Downloaded from molpharm.aspetjournals.org at ASPET Journals on September 28, 2021 1 This article has not been copyedited and formatted. The final version may differ from this version. This article has not been copyedited and formatted. The final version may differ from this version. This article has not been copyedited and formatted. The final version may differ from this version. This article has not been copyedited and formatted. The final version may differ from this version. This article has not been copyedited and formatted. The final version may differ from this version. This article has not been copyedited and formatted. The final version may differ from this version. This article has not been copyedited and formatted. The final version may differ from this version. This article has not been copyedited and formatted. The final version may differ from this version. This article has not been copyedited and formatted. The final version may differ from this version. This article has not been copyedited and formatted. The final version may differ from this version. This article has not been copyedited and formatted. The final version may differ from this version. This article has not been copyedited and formatted. The final version may differ from this version. This article has not been copyedited and formatted. The final version may differ from this version. This article has not been copyedited and formatted. The final version may differ from this version. This article has not been copyedited and formatted. The final version may differ from this version. This article has not been copyedited and formatted. -

The Analysis of the Mcf7 Cancer Model System And
THE ANALYSIS OF THE MCF7 CANCER MODEL SYSTEM AND THE EFFECTS OF 5-AZA-2’-DEOXYCYTIDINE TREATMENT ON THE CHROMATIN STATE USING A NOVEL MICROARRAY-BASED TECHNOLOGY FOR HIGH RESOLUTION GLOBAL CHROMATIN STATE MEASUREMENT APPROVED BY SUPERVISORY COMMITTEE Harold Garner, Ph.D. Elliott Ross, Ph.D. Thomas Kodadek, Ph.D. John Minna, M.D. Keith Wharton, M.D., Ph.D. DEDICATION Omnibus qui adiuverunt THE ANALYSIS OF THE MCF7 CANCER MODEL SYSTEM AND THE EFFECTS OF 5-AZA-2’-DEOXYCYTIDINE TREATMENT ON THE CHROMATIN STATE USING A NOVEL MICROARRAY-BASED TECHNOLOGY FOR HIGH RESOLUTION GLOBAL CHROMATIN STATE MEASUREMENT by MICHAEL RYAN WEIL DISSERTATION Presented to the Faculty of the Graduate School of Biomedical Sciences The University of Texas Southwestern Medical Center at Dallas In Partial Fulfillment of the Requirements For the Degree of DOCTOR OF PHILOSOPHY The University of Texas Southwestern Medical Center at Dallas Dallas, Texas March, 2006 Copyright by Michael Ryan Weil All Rights Reserved THE ANALYSIS OF THE MCF7 CANCER MODEL SYSTEM AND THE EFFECTS OF 5-AZA-2’-DEOXYCYTIDINE TREATMENT ON THE CHROMATIN STATE USING A NOVEL MICROARRAY-BASED TECHNOLOGY FOR HIGH RESOLUTION GLOBAL CHROMATIN STATE MEASUREMENT Publication No. MICHAEL RYAN WEIL, B.S. The University of Texas Southwestern Medical Center at Dallas, 2006 Supervising Professor: Harold Ray (Skip) Garner, Ph.D. A microarray method to measure the global chromatin state of the human genome was developed in order to provide a novel view of gene regulation. The 'chromatin array' employs traditional methods of chromatin isolation, microarray technology, and advanced data analysis, and was applied to a cancer model system.