國立中山大學海洋生物研究所碩士論文指導教授︰劉莉蓮博士台灣西海岸蚵岩螺(Thais Clavigera)
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

E Urban Sanctuary Algae and Marine Invertebrates of Ricketts Point Marine Sanctuary
!e Urban Sanctuary Algae and Marine Invertebrates of Ricketts Point Marine Sanctuary Jessica Reeves & John Buckeridge Published by: Greypath Productions Marine Care Ricketts Point PO Box 7356, Beaumaris 3193 Copyright © 2012 Marine Care Ricketts Point !is work is copyright. Apart from any use permitted under the Copyright Act 1968, no part may be reproduced by any process without prior written permission of the publisher. Photographs remain copyright of the individual photographers listed. ISBN 978-0-9804483-5-1 Designed and typeset by Anthony Bright Edited by Alison Vaughan Printed by Hawker Brownlow Education Cheltenham, Victoria Cover photo: Rocky reef habitat at Ricketts Point Marine Sanctuary, David Reinhard Contents Introduction v Visiting the Sanctuary vii How to use this book viii Warning viii Habitat ix Depth x Distribution x Abundance xi Reference xi A note on nomenclature xii Acknowledgements xii Species descriptions 1 Algal key 116 Marine invertebrate key 116 Glossary 118 Further reading 120 Index 122 iii Figure 1: Ricketts Point Marine Sanctuary. !e intertidal zone rocky shore platform dominated by the brown alga Hormosira banksii. Photograph: John Buckeridge. iv Introduction Most Australians live near the sea – it is part of our national psyche. We exercise in it, explore it, relax by it, "sh in it – some even paint it – but most of us simply enjoy its changing modes and its fascinating beauty. Ricketts Point Marine Sanctuary comprises 115 hectares of protected marine environment, located o# Beaumaris in Melbourne’s southeast ("gs 1–2). !e sanctuary includes the coastal waters from Table Rock Point to Quiet Corner, from the high tide mark to approximately 400 metres o#shore. -

The Influence of Canopy Cover on the Ecological Function of a Key Autogenic Ecosystem Engineer
diversity Article The Influence of Canopy Cover on the Ecological Function of A Key Autogenic Ecosystem Engineer Jacqueline B. Pocklington 1,2,*, Michael J. Keough 2, Tim D. O’Hara 1 and Alecia Bellgrove 3 1 Department of Marine Invertebrates, Museum Victoria, Carlton, VIC 3053, Australia; [email protected] 2 School of BioSciences, University of Melbourne, Parkville, VIC 3010, Australia; [email protected] 3 School of Life and Environmental Sciences, Centre for Integrative Ecology, Deakin University, Warrnambool Campus, PO Box 423, Warrnambool, VIC 3280, Australia; [email protected] * Correspondence: [email protected] Received: 8 April 2019; Accepted: 15 May 2019; Published: 17 May 2019 Abstract: Intertidal fucoid algae can function as ecosystem engineers across temperate marine regions. In this investigation, we assessed the function of the alga dominating rocky reefs in temperate Australia and New Zealand, Hormosira banksii. Invertebrate and algal species assemblages were examined within areas of full H. banksii canopy, areas where it was naturally patchy or absent (within its potential range on the shore) and areas where the intact canopy was experimentally disturbed. Differences in species assemblages were detected between areas with natural variation in H. banksii cover (full, patchy, negligible), with defined species associated with areas of full cover. Differences were also detected between experimentally manipulated and naturally patchy areas of canopy cover. Species assemblages altered in response to canopy manipulations and did not recover even twelve months after initial sampling. Both light intensity and temperature were buffered by full canopies compared to patchy canopies and exposed rock. This study allows us to predict the consequences to the intertidal community due to the loss of canopy cover, which may result from a range of disturbances such as trampling, storm damage, sand burial and prolonged exposure to extreme temperature, and further allow for improved management of this key autogenic ecosystem engineer. -

2006-2007 Intertidal Reef Biodiversity on Kangaroo
2006-2007 Kangaroo Island Natural Resources Management Board INTERTIDAL REEF BIODIVERSITY Intertidal Reef Biodiversity on Kangaroo Island – 2007 ON KANGAROO ISLAND 1 INTERTIDAL REEF BIODIVERSITY ON KANGAROO ISLAND Oceans of Blue: Coast, Estuarine and Marine Monitoring Program A report prepared for the Kangaroo Island Natural Resources Management Board by Kirsten Benkendorff Martine Kinloch Daniel Brock June 2007 2006-2007 Kangaroo Island Natural Resources Management Board Intertidal Reef Biodiversity on Kangaroo Island – 2007 2 Oceans of Blue The views expressed and the conclusions reached in this report are those of the author and not necessarily those of persons consulted. The Kangaroo Island Natural Resources Management Board shall not be responsible in any way whatsoever to any person who relies in whole or in part on the contents of this report. Project Officer Contact Details Martine Kinloch Coast and Marine Program Manager Kangaroo Island Natural Resources Management Board PO Box 665 Kingscote SA 5223 Phone: (08) 8553 4980 Fax: (08) 8553 0122 Email: [email protected] Kangaroo Island Natural Resources Management Board Contact Details Jeanette Gellard General Manager PO Box 665 Kingscote SA 5223 Phone: (08) 8553 0111 Fax: (08) 8553 0122 Email: [email protected] © Kangaroo Island Natural Resources Management Board This document may be reproduced in whole or part for the purpose of study or training, subject to the inclusion of an acknowledgment of the source and to its not being used for commercial purposes or sale. Reproduction for purposes other than those given above requires the prior written permission of the Kangaroo Island Natural Resources Management Board. -

Download Full Article 2.0MB .Pdf File
Memoirs of the National Museum of Victoria 12 April 1971 Port Phillip Bay Survey 2 https://doi.org/10.24199/j.mmv.1971.32.08 8 INTERTIDAL ECOLOGY OF PORT PHILLIP BAY WITH SYSTEMATIC LIST OF PLANTS AND ANIMALS By R. J. KING,* J. HOPE BLACKt and SOPHIE c. DUCKER* Abstract The zonation is recorded at 14 stations within Port Phillip Bay. Any special features of a station arc di�cusscd in �elation to the adjacent stations and the whole Bay. The intertidal plants and ammals are listed systematically with references, distribution within the Bay and relevant comment. 1. INTERTIDAL ECOLOGY South-western Bay-Areas 42, 49, 50 By R. J. KING and J. HOPE BLACK Arca 42: Station 21 St. Leonards 16 Oct. 69 Introduction Arca 49: Station 4 Swan Bay Jetty, 17 Sept. 69 This account is basically coneerncd with the distribution of intertidal plants and animals of Eastern Bay-Areas 23-24, 35-36, 47-48, 55 Port Phillip Bay. The benthic flora and fauna Arca 23, Station 20, Ricketts Pt., 30 Sept. 69 have been dealt with in separate papers (Mem Area 55: Station 15 Schnapper Pt. 25 May oir 27 and present volume). 70 Following preliminary investigations, 14 Area 55: Station 13 Fossil Beach 25 May stations were selected for detailed study in such 70 a way that all regions and all major geological formations were represented. These localities Southern Bay-Areas 60-64, 67-70 are listed below and are shown in Figure 1. Arca 63: Station 24 Martha Pt. 25 May 70 For ease of comparison with Womersley Port Phillip Heads-Areas 58-59 (1966), in his paper on the subtidal algae, the Area 58: Station 10 Quecnscliff, 12 Mar. -

Science Journals
View metadata, citation and similar papers at core.ac.uk brought to you by CORE provided by NERC Open Research Archive SCIENCE ADVANCES | RESEARCH ARTICLE MARINE ECOSYSTEMS Copyright © 2017 The Authors, some Latitudinal trends in shell production cost from the rights reserved; exclusive licensee tropics to the poles American Association for the Advancement 1,2 3 3 of Science. No claim to Sue-Ann Watson, * Simon A. Morley, Lloyd S. Peck original U.S. Government Works. Distributed The proportion of body mass devoted to skeleton in marine invertebrates decreases along latitudinal gradients under a Creative — from large proportions in the tropics to small proportions in polar regions. A historical hypothesis that latitudinal Commons Attribution differences in shell production costs explain these trends—remains untested. Using field-collected specimens NonCommercial spanning a 79°N to 68°S latitudinal gradient (16,300 km), we conducted a taxonomically controlled evaluation of License 4.0 (CC BY-NC). energetic costs of shell production as a proportion of the total energy budget in mollusks. Shell production cost was fairly low across latitudes at <10% of the energy budget and predominately <5% in gastropods and <4% in bivalves. Throughout life, shell cost tended to be lower in tropical species and increased slightly toward the poles. However, shell cost also varied with life stage, with the greatest costs found in young tropical gastropods. Low shell production costs on the energy budget suggest that shell cost may play only a small role in influencing proportional skeleton size gradients across latitudes relative to other ecological factors, such as predation in present-day oceans. -

Triphoridae in Northern New South Wales – Diversity and New Records
ISSN 1834-4259 NO. 162 JULY, 2017 Triphoridae in northern New South Wales – diversity and new records. Lynton D. Stephens Email: [email protected] The coastline of northern New South Wales possesses a -tional sources of information on south-eastern Austral- high diversity of molluscs and was an apt venue for the ian species include Wilson (1993), Stephens & Vafiadis MALSOC conference in Nov-Dec 2015. The faunal (2015) and Stephens (2015). composition of the area is largely a mix of three groups: 1). temperate Australian species at or near their northern Between 2008 and 2015 I was fortunate to spend sev- end of range; 2). incursive tropical species, and 3). east- eral months on the northern NSW coast and was able ern seaboard endemics. North of Coffs Harbour the to collect and analyse some fairly extensive samples of fauna is quite different from that seen even around Syd- shellgrit. Upon the publication of Beechey (2015), I ney, with, as one might expect, a much higher diversity realised that several identified species had not been and proportion of tropical species present. previously recorded from NSW, and/or not illustrated by Beechey. A few short notes on these and other spe- Emblematic of this is the family Triphoridae, a group of cies are provided below, including 8 new records for generally sinistral gastropods which feed on sponges. NSW. The presence of unrecognised Triphoridae in Temperate Australian species were reviewed by Marshall the north of the state is no surprise; nevertheless, this (1983), who imaged most using SEM. A very useful information may be of benefit to anyone sampling or guide was published more recently by Beechey (2015), surveying in the area. -

The Fisher Island Field Stat Ion-With an Account of Its Principal Fauna and Flora
PAPEHS AND .PROCEEDINGS tW 'THE ROYAL SOCIETY OF TASMANIA, V'OLl.lME 92 THE FISHER ISLAND FIELD STAT ION-WITH AN ACCOUNT OF ITS PRINCIPAL FAUNA AND FLORA By E. R. GUILER, D. L. SERVENTY AND J. H. WILLIS (WITH 2 PLATES AND 9 TEXT FTGURESj t GENERAL DESCRIPTION OF FISHER ISLAND AND ITS MUTTON~BIRD ROOKERIES * INTRODUCTION mately 0'75 acres. The shoreline measures about Fisher Island Oat. 40° 10' S., long. 148° 16' E.) 530 yards and the greatest length, from North is among the smallest of the archipelago of islands Point to South Point, is 150 yards. Its elevation is comprising the Furneaux Group in eastern Bass about 19 feet above spring high-water mark. Strait. It lies off the southern shoreline of the Like the other islands in the Furneaux Group, major island in the group, Flinders Island, in Fisher Island is part of the basement Devonian Adelaide Bay, a portion of Franklin Sound which granite which forms the hills and mountain ridges separates Flinders Island from Cape Barren Island in the archipelago. On Flinders Island the low (fig. 1). Its convenient location to Lady Barron lying plains are covered by Tertiary alluvium and (the main port of Flinders Island, about 220 yards sands, with calcareous deposits in restricted areas. distant), its proximity to important commercial Limited Siluro-Devonian quartzites and slates also mutton-birding islands and the presence of a small, occur, and in the northern part of Adelaide Bay, at easily handled nesting colony of mutton-birds Petrifaction Bay, are exposures of Tertiary vesicu (PufJinus tenuirostris (Temminck), made it the lar basalts. -
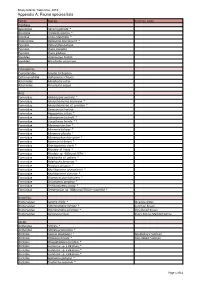
TAS-Bruny-Compiled Report-Appendix A
Bruny Island, Tasmania, 2016 Appendix A: Fauna species lists Family Species Common name Ascidians Agneziidae Agnezia glaciata * Ascididae Ascidiella aspersa ^ Cionidae Ciona intestinalis ^ Didemnidae Diplosoma listerianum? * Pyuridae Halocynthia dumosa Pyuridae Pyura elongata Pyuridae Pyura gibbosa Styelidae Asterocarpa humilis Styelidae Botrylloides perspicuus Echinoderms Comasteridae Cenolia trichoptera Ophionereididae Ophionereis schayeri Asterinidae Meridiastra calcar Asterinidae Parvulastra exigua Ants Formicidae Amblyopone australis * Formicidae Anonychomyrma biconvexa * Formicidae Anonychomyrma cf. procidua * Formicidae Camponotus hartogi Formicidae Hypoponera scitula * Formicidae Iridomyrmex bicknelli * Formicidae Linepithema humile * ^ Formicidae Monomorium leae * Formicidae Myrmecia fulvipes * Formicidae Myrmecia pilosula * Formicidae Myrmecorhynchus carteri * Formicidae Notoncus hickmani * Formicidae Orectognathus clarki * Formicidae Pheidole cf. liteae * Formicidae Pheidole sp. 'BBBrunyLF07A' * Formicidae Polyrhachis cf. patiens * Formicidae Polyrhachis femorata * Formicidae Prolasius abruptus * Formicidae Rhytidoponera tasmaniensis * Formicidae Rhytidoponera victoriae * Formicidae Stigmacros punctatissima * Formicidae Strumigenys perplexa * Formicidae Technomyrmex jocosa * Formicidae Tetramorium sp. 'BBBrunyLFSSS2nr-mayriella' * Butterflies Nymphalidae Junonia villida * Meadow Argus Nymphalidae Heteronympha merope * Common Brown Nymphalidae Heteronympha penelope * Shouldered Brown Nymphalidae Geitoneura klugii Klug's Xenica; -

Low Global Sensitivity of Metabolic Rate to Temperature in Calcified Marine Invertebrates
Oecologia (2014) 174:45–54 DOI 10.1007/s00442-013-2767-8 PHYSIOLOGICAL ECOLOGY - ORIGINAL RESEARCH Low global sensitivity of metabolic rate to temperature in calcified marine invertebrates Sue‑Ann Watson · Simon A. Morley · Amanda E. Bates · Melody S. Clark · Robert W. Day · Miles Lamare · Stephanie M. Martin · Paul C. Southgate · Koh Siang Tan · Paul A. Tyler · Lloyd S. Peck Received: 11 December 2012 / Accepted: 29 August 2013 / Published online: 14 September 2013 © The Author(s) 2013. This article is published with open access at Springerlink.com Abstract Metabolic rate is a key component of energy of calcified invertebrates (bivalves, gastropods, urchins budgets that scales with body size and varies with large- and brachiopods), using a single consistent methodology, scale environmental geographical patterns. Here we con- including 11 species whose SMR was described for the duct an analysis of standard metabolic rates (SMR) of first time. SMR of 15 out of 17 species had a mass-scaling marine ectotherms across a 70° latitudinal gradient in exponent between 2/3 and 1, with no greater support for both hemispheres that spanned collection temperatures of a 3/4 rather than a 2/3 scaling exponent. After accounting 0–30 °C. To account for latitudinal differences in the size for taxonomy and variability in parameter estimates among and skeletal composition between species, SMR was mass species using variance-weighted linear mixed effects mod- normalized to that of a standard-sized (223 mg) ash-free elling, temperature sensitivity of SMR had an activation dry mass individual. SMR was measured for 17 species energy (Ea) of 0.16 for both Northern and Southern Hemi- sphere species which was lower than predicted under the metabolic theory of ecology (Ea 0.2–1.2 eV). -

Science Journals
SCIENCE ADVANCES | RESEARCH ARTICLE MARINE ECOSYSTEMS Copyright © 2017 The Authors, some Latitudinal trends in shell production cost from the rights reserved; exclusive licensee tropics to the poles American Association for the Advancement 1,2 3 3 of Science. No claim to Sue-Ann Watson, * Simon A. Morley, Lloyd S. Peck original U.S. Government Works. Distributed The proportion of body mass devoted to skeleton in marine invertebrates decreases along latitudinal gradients under a Creative — from large proportions in the tropics to small proportions in polar regions. A historical hypothesis that latitudinal Commons Attribution differences in shell production costs explain these trends—remains untested. Using field-collected specimens NonCommercial spanning a 79°N to 68°S latitudinal gradient (16,300 km), we conducted a taxonomically controlled evaluation of License 4.0 (CC BY-NC). energetic costs of shell production as a proportion of the total energy budget in mollusks. Shell production cost was fairly low across latitudes at <10% of the energy budget and predominately <5% in gastropods and <4% in bivalves. Throughout life, shell cost tended to be lower in tropical species and increased slightly toward the poles. However, shell cost also varied with life stage, with the greatest costs found in young tropical gastropods. Low shell production costs on the energy budget suggest that shell cost may play only a small role in influencing proportional skeleton size gradients across latitudes relative to other ecological factors, such as predation in present-day oceans. However, any Downloaded from increase in the cost of calcium carbonate (CaCO3) deposition, including from ocean acidification, may lead to a projected ~50 to 70% increase in the proportion of the total energy budget required for shell production for a doubling of the CaCO3 deposition cost. -

Notes on the Intertidal Ecology of Trial Harbour, Tasmania
PAPERS AND PROCEEDINGS OF THE HOYAL SOCIETY OF TAI::3MANIA. Vo,LUME 94 NOTES ON THE INTERTIDAL ECOLOGY OF TRIAL HARBOUR, TASMANIA. By E. R. GUILER Department of Zoology, University of Tasmania. (With 3 plates) 1. Introduction. 2. DESCRIPTION OF THE TRIAL HARBOUR 2. Description of Area. AREA 3. Methods and Nomenclature. Trial Harbour, latitude 41 0 59' S., consists of 4. The Physical Environment. a reef extending a few chains out to sea in a north 5. Ecology of the Reef. westerly direction and some smaller reefs along the coast with boulder beaches. The roadstead is 6. Notes on nearby Areas. narrow. 7. Discussion. To the north of Trial Harbour the coastline 8. Acknowledgements. consists of rocky cliffs trending generally in a 9. References. north-westerly direction. The cliffs are only feebly incised by watercourses and there are few boulder ABSTRACT beaches. To the south of Trial Harbour there are three low rocky headlands interspaced by beaches. A description is given on the zonation and general The beaches are composed of yellow quartzose ecology of a reef on the exposed West Coast of rcunded grit fragments forming a rather soft gritty Tasmania. The reef shows the unusual feature sand. The hard packing of sand found on many of a Hormosira-Corallina belt on an exposed coast. ocean beaches is lacking here. There was evidence A white species of Lithothamnion which is con in November, 1956, of a very considerable removal sidered to be characteristic of cold water con by marine agencies of sand from one beach to the ditions occurs there. -

Low Global Sensitivity of Metabolic Rate to Temperature in Calcified Marine Invertebrates
Oecologia DOI 10.1007/s00442-013-2767-8 PHYSIOLOGICAL ECOLOGY - ORIGINAL RESEARCH Low global sensitivity of metabolic rate to temperature in calcified marine invertebrates Sue‑Ann Watson · Simon A. Morley · Amanda E. Bates · Melody S. Clark · Robert W. Day · Miles Lamare · Stephanie M. Martin · Paul C. Southgate · Koh Siang Tan · Paul A. Tyler · Lloyd S. Peck Received: 11 December 2012 / Accepted: 29 August 2013 © The Author(s) 2013. This article is published with open access at Springerlink.com Abstract Metabolic rate is a key component of energy of calcified invertebrates (bivalves, gastropods, urchins budgets that scales with body size and varies with large- and brachiopods), using a single consistent methodology, scale environmental geographical patterns. Here we con- including 11 species whose SMR was described for the duct an analysis of standard metabolic rates (SMR) of first time. SMR of 15 out of 17 species had a mass-scaling marine ectotherms across a 70° latitudinal gradient in exponent between 2/3 and 1, with no greater support for both hemispheres that spanned collection temperatures of a 3/4 rather than a 2/3 scaling exponent. After accounting 0–30 °C. To account for latitudinal differences in the size for taxonomy and variability in parameter estimates among and skeletal composition between species, SMR was mass species using variance-weighted linear mixed effects mod- normalized to that of a standard-sized (223 mg) ash-free elling, temperature sensitivity of SMR had an activation dry mass individual. SMR was measured for 17 species energy (Ea) of 0.16 for both Northern and Southern Hemi- sphere species which was lower than predicted under the metabolic theory of ecology (Ea 0.2–1.2 eV).