Decoding the Main Genomic Hallmark of Trichoderma
Total Page:16
File Type:pdf, Size:1020Kb

Load more
Recommended publications
-

Supplementary Information
Supplementary information (a) (b) Figure S1. Resistant (a) and sensitive (b) gene scores plotted against subsystems involved in cell regulation. The small circles represent the individual hits and the large circles represent the mean of each subsystem. Each individual score signifies the mean of 12 trials – three biological and four technical. The p-value was calculated as a two-tailed t-test and significance was determined using the Benjamini-Hochberg procedure; false discovery rate was selected to be 0.1. Plots constructed using Pathway Tools, Omics Dashboard. Figure S2. Connectivity map displaying the predicted functional associations between the silver-resistant gene hits; disconnected gene hits not shown. The thicknesses of the lines indicate the degree of confidence prediction for the given interaction, based on fusion, co-occurrence, experimental and co-expression data. Figure produced using STRING (version 10.5) and a medium confidence score (approximate probability) of 0.4. Figure S3. Connectivity map displaying the predicted functional associations between the silver-sensitive gene hits; disconnected gene hits not shown. The thicknesses of the lines indicate the degree of confidence prediction for the given interaction, based on fusion, co-occurrence, experimental and co-expression data. Figure produced using STRING (version 10.5) and a medium confidence score (approximate probability) of 0.4. Figure S4. Metabolic overview of the pathways in Escherichia coli. The pathways involved in silver-resistance are coloured according to respective normalized score. Each individual score represents the mean of 12 trials – three biological and four technical. Amino acid – upward pointing triangle, carbohydrate – square, proteins – diamond, purines – vertical ellipse, cofactor – downward pointing triangle, tRNA – tee, and other – circle. -
The Metabolic Building Blocks of a Minimal Cell Supplementary
The metabolic building blocks of a minimal cell Mariana Reyes-Prieto, Rosario Gil, Mercè Llabrés, Pere Palmer and Andrés Moya Supplementary material. Table S1. List of enzymes and reactions modified from Gabaldon et. al. (2007). n.i.: non identified. E.C. Name Reaction Gil et. al. 2004 Glass et. al. 2006 number 2.7.1.69 phosphotransferase system glc + pep → g6p + pyr PTS MG041, 069, 429 5.3.1.9 glucose-6-phosphate isomerase g6p ↔ f6p PGI MG111 2.7.1.11 6-phosphofructokinase f6p + atp → fbp + adp PFK MG215 4.1.2.13 fructose-1,6-bisphosphate aldolase fbp ↔ gdp + dhp FBA MG023 5.3.1.1 triose-phosphate isomerase gdp ↔ dhp TPI MG431 glyceraldehyde-3-phosphate gdp + nad + p ↔ bpg + 1.2.1.12 GAP MG301 dehydrogenase nadh 2.7.2.3 phosphoglycerate kinase bpg + adp ↔ 3pg + atp PGK MG300 5.4.2.1 phosphoglycerate mutase 3pg ↔ 2pg GPM MG430 4.2.1.11 enolase 2pg ↔ pep ENO MG407 2.7.1.40 pyruvate kinase pep + adp → pyr + atp PYK MG216 1.1.1.27 lactate dehydrogenase pyr + nadh ↔ lac + nad LDH MG460 1.1.1.94 sn-glycerol-3-phosphate dehydrogenase dhp + nadh → g3p + nad GPS n.i. 2.3.1.15 sn-glycerol-3-phosphate acyltransferase g3p + pal → mag PLSb n.i. 2.3.1.51 1-acyl-sn-glycerol-3-phosphate mag + pal → dag PLSc MG212 acyltransferase 2.7.7.41 phosphatidate cytidyltransferase dag + ctp → cdp-dag + pp CDS MG437 cdp-dag + ser → pser + 2.7.8.8 phosphatidylserine synthase PSS n.i. cmp 4.1.1.65 phosphatidylserine decarboxylase pser → peta PSD n.i. -
Q 297 Suppl USE
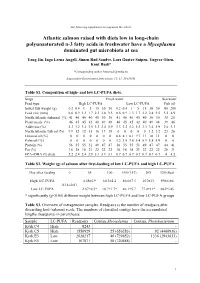
The following supplement accompanies the article Atlantic salmon raised with diets low in long-chain polyunsaturated n-3 fatty acids in freshwater have a Mycoplasma dominated gut microbiota at sea Yang Jin, Inga Leena Angell, Simen Rød Sandve, Lars Gustav Snipen, Yngvar Olsen, Knut Rudi* *Corresponding author: [email protected] Aquaculture Environment Interactions 11: 31–39 (2019) Table S1. Composition of high- and low LC-PUFA diets. Stage Fresh water Sea water Feed type High LC-PUFA Low LC-PUFA Fish oil Initial fish weight (g) 0.2 0.4 1 5 15 30 50 0.2 0.4 1 5 15 30 50 80 200 Feed size (mm) 0.6 0.9 1.3 1.7 2.2 2.8 3.5 0.6 0.9 1.3 1.7 2.2 2.8 3.5 3.5 4.9 North Atlantic fishmeal (%) 41 40 40 40 40 30 30 41 40 40 40 40 30 30 35 25 Plant meals (%) 46 45 45 42 40 49 48 46 45 45 42 40 49 48 39 46 Additives (%) 3.3 3.2 3.2 3.5 3.3 3.4 3.9 3.3 3.2 3.2 3.5 3.3 3.4 3.9 2.6 3.3 North Atlantic fish oil (%) 9.9 12 12 15 16 17 18 0 0 0 0 0 1.2 1.2 23 26 Linseed oil (%) 0 0 0 0 0 0 0 6.8 8.1 8.1 9.7 11 10 11 0 0 Palm oil (%) 0 0 0 0 0 0 0 3.2 3.8 3.8 5.4 5.9 5.8 5.9 0 0 Protein (%) 56 55 55 51 49 47 47 56 55 55 51 49 47 47 44 41 Fat (%) 16 18 18 21 22 22 22 16 18 18 21 22 22 22 28 31 EPA+DHA (% diet) 2.2 2.4 2.4 2.9 3.1 3.1 3.1 0.7 0.7 0.7 0.7 0.7 0.7 0.7 4 4.2 Table S2. -

Structural and Mechanistic Investigations on Salmonella
Chittori et al. BMC Structural Biology 2012, 12:24 http://www.biomedcentral.com/1472-6807/12/24 RESEARCH ARTICLE Open Access Structural and mechanistic investigations on Salmonella typhimurium acetate kinase (AckA): identification of a putative ligand binding pocket at the dimeric interface Sagar Chittori1,3, Handanahal S Savithri2 and Mathur RN Murthy1* Abstract Background: Bacteria such as Escherichia coli and Salmonella typhimurium can utilize acetate as the sole source of carbon and energy. Acetate kinase (AckA) and phosphotransacetylase (Pta), key enzymes of acetate utilization pathway, regulate flux of metabolites in glycolysis, gluconeogenesis, TCA cycle, glyoxylate bypass and fatty acid metabolism. Results: Here we report kinetic characterization of S. typhimurium AckA (StAckA) and structures of its unliganded (Form-I, 2.70 Å resolution) and citrate-bound (Form-II, 1.90 Å resolution) forms. The enzyme showed broad substrate specificity with kcat/Km in the order of acetate > propionate > formate. Further, the Km for acetyl-phosphate was significantly lower than for acetate and the enzyme could catalyze the reverse reaction (i.e. ATP synthesis) more efficiently. ATP and Mg2+ could be substituted by other nucleoside 50-triphosphates (GTP, UTP and CTP) and divalent cations (Mn2+ and Co2+), respectively. Form-I StAckA represents the first structural report of an unliganded AckA. StAckA protomer consists of two domains with characteristic βββαβαβα topology of ASKHA superfamily of proteins. These domains adopt an intermediate conformation compared to that of open and closed forms of ligand-bound Methanosarcina thermophila AckA (MtAckA). Spectroscopic and structural analyses of StAckA further suggested occurrence of inter-domain motion upon ligand-binding. -

Phyre 2 Results for P50456
Email [email protected] Description P50456 Thu Jan 5 12:04:43 GMT Date 2012 Unique Job d92874c542ac9979 ID Detailed template information # Template Alignment Coverage 3D Model Confidence % i.d. Template Information PDB header:transcription Chain: A: PDB Molecule:transcriptional regulator, rok family; 1 c1z05A_ 100.0 45 Alignment PDBTitle: crystal structure of the rok family transcriptional regulator, homolog2 of e.coli mlc protein. PDB header:transcription 2 c1z6rC_ Alignment 100.0 98 Chain: C: PDB Molecule:mlc protein; PDBTitle: crystal structure of mlc from escherichia coli PDB header:transferase Chain: A: PDB Molecule:n-acetylglucosamine kinase; 3 c2hoeA_ 100.0 23 Alignment PDBTitle: crystal structure of n-acetylglucosamine kinase (tm1224) from2 thermotoga maritima at 2.46 a resolution PDB header:transferase Chain: A: PDB Molecule:glucokinase; 4 c3mcpA_ 100.0 18 Alignment PDBTitle: crystal structure of glucokinase (bdi_1628) from parabacteroides2 distasonis atcc 8503 at 3.00 a resolution PDB header:transferase Chain: D: PDB Molecule:glucokinase; 5 c2qm1D_ 100.0 24 Alignment PDBTitle: crystal structure of glucokinase from enterococcus faecalis PDB header:transferase Chain: B: PDB Molecule:glucokinase; 6 c3vgkB_ 100.0 25 Alignment PDBTitle: crystal structure of a rok family glucokinase from streptomyces2 griseus PDB header:transferase Chain: A: PDB Molecule:hypothetical sugar kinase; 7 c3r8eA_ Alignment 100.0 19 PDBTitle: crystal structure of a hypothetical sugar kinase (chu_1875) from2 cytophaga hutchinsonii atcc 33406 at 1.65 -

Roles of the Methylcitrate and Methylmalonyl-COA Pathways in Mycobacterial Metabolism and Pathogenesis" (2012)
Rockefeller University Digital Commons @ RU Student Theses and Dissertations 2012 Roles of the Methylcitrate and Methylmalonyl- COA Pathways in Mycobacterial Metabolism and Pathogenesis Manisha Ulhas Lotlikar Follow this and additional works at: http://digitalcommons.rockefeller.edu/ student_theses_and_dissertations Part of the Life Sciences Commons Recommended Citation Lotlikar, Manisha Ulhas, "Roles of the Methylcitrate and Methylmalonyl-COA Pathways in Mycobacterial Metabolism and Pathogenesis" (2012). Student Theses and Dissertations. Paper 245. This Thesis is brought to you for free and open access by Digital Commons @ RU. It has been accepted for inclusion in Student Theses and Dissertations by an authorized administrator of Digital Commons @ RU. For more information, please contact [email protected]. ROLES OF THE METHYLCITRATE AND METHYLMALONYL-COA PATHWAYS IN MYCOBACTERIAL METABOLISM AND PATHOGENESIS A Thesis Presented to the Faculty of The Rockefeller University in Partial Fulfillment of the Requirements for the degree of Doctor of Philosophy by Manisha Ulhas Lotlikar June 2012 © Copyright by Manisha Ulhas Lotlikar 2012 ROLES OF THE METHYLCITRATE AND METHYLMALONYL-COA PATHWAYS IN MYCOBACTERIAL METABOLISM AND PATHOGENESIS Manisha Ulhas Lotlikar, Ph.D. The Rockefeller University 2012 Mycobacterium tuberculosis has been a human pathogen for the history of mankind, but we are only now beginning to understand how it is able to survive and persist indefinitely in the host. Understanding carbon metabolism of the pathogen during infection is key, not only as a source of potential drug targets, but also for elucidating the environment in vivo, so that drugs can be tested under relevant conditions. Studies have revealed that, during infection, M. tuberculosis relies on gluconeogenic carbon sources rather than sugars. -

Appendix C. List of Gene Up-Regulated in Cells Grown As Biofilms. Protein Category GENE NAME Function Foldchange Aveexpr T Raw.P.Val FDR.P.Val
Appendix C. List of gene up-regulated in cells grown as biofilms. Protein category GENE_NAME function FoldChange AveExpr t raw.P.val FDR.P.val Cell membrane function lmo1934 similar to non-specific DNA-binding protein HU 7.88455731 12.20228 13.74284 5.14E-16 7.69E-13 lmo2016 similar to major cold-shock protein 4.46054486 12.92885 12.4972 9.16E-15 5.06E-12 lmo1847 similar to adhesion binding proteins and lipoproteins with multiple5.10387178 specificity13.71246 for metal 11.18586cations (ABC2.32E-13 transporter)3.31E-11 lmo1548 similar to cell-shape determining protein MreB 3.12698287 13.84029 10.14071 3.58E-12 3.14E-10 lmo1364 similar to cold shock protein 3.21172011 12.68556 10.03885 4.71E-12 3.63E-10 lmo1547 similar to cell-shape determining protein MreC 3.25276936 13.32978 10.03399 4.77E-12 3.63E-10 lmo0971 DltD protein for D-alanine esterification of lipoteichoic acid and3.42366229 wall teichoic13.88932 acid 9.280949 3.77E-11 2.29E-09 lmo0973 DltB protein for D-alanine esterification of lipoteichoic acid and3.07776803 wall teichoic14.47105 acid 8.732509 1.78E-10 8.18E-09 lmo1546 similar to cell-shape determining protein MreD 3.22349266 12.61449 8.053721 1.28E-09 4.49E-08 lmo0045 highly similar to single-strand binding protein (SSB) 3.27406859 14.06565 7.792284 2.78E-09 8.59E-08 lmo1527 similar to protein-export membrane protein SecDF 2.46823503 13.93627 6.263443 2.90E-07 4.75E-06 lmo2637 conserved lipoprotein 3.67182434 14.17184 6.07871 5.14E-07 7.35E-06 lmo0927 hypothetical transmembrane protein 2.09623116 13.21212 5.573679 2.46E-06 -

E"ESNI) 379 a ROM Is A
USOO7745.391 B2 (12) United States Patent (10) Patent No.: US 7,745,391 B2 Mintz et al. (45) Date of Patent: Jun. 29, 2010 (54) HUMAN THROMBOSPONDIN POLYPEPTIDE 5,283,317. A 2/1994 Saifer et al. 5,288,514. A 2/1994 Ellman (75) Inventors: Liat Mintz, East Brunswick, NJ (US); 5,328,470 A 7, 1994 Nabel et al. Hanging Xie, Lambertville, NJ (US); 5,384,261 A 1/1995 Winkler et al. Dvir Dahary, Tel-Aviv (IL); Erez 5,459,039 A 10/1995 Modrich et al. Levanon, Petach-Tikva (IL), Shiri 5,475,092 A 12/1995 Charietal. Freilich, Herzlia (IL); Nili Beck, Kfar 5,498,531 A 3, 1996 Jarrell Saba (IL); Wei-Yong Zhu, Plainsboro, 5,510,270 A 4/1996 Fodor et al. E"ESNI)s s 379- J. A ROMomoglio is et al.a (73) Assignee: Compugen Ltd., Tel-Aviv (IL) 5,585,089 A 12/1996 Queen et al. 5,585.499 A 12/1996 Chari et al. (*) Notice: Subject to any disclaimer, the term of this 5,631, 169 A 5/1997 Lakowicz et al. patent is extended or adjusted under 35 5,693,761 A 12/1997 Queen et al. U.S.C. 154(b) by 441 days. 5,693,762. A 12/1997 Queen et al. 5,695,937 A 12/1997 Kinzler et al. (21) Appl. No.: 11/443,428 5,846,545. A 12/1998 Chari et al. 5,854,033. A 12/1998 Lizardi (22) Filed: May 31, 2006 5,876,742 A 3/1999 Cochrum et al. -

2019 Strain Engineering for Mi
Biotechnology Advances 37 (2019) 538–568 Contents lists available at ScienceDirect Biotechnology Advances journal homepage: www.elsevier.com/locate/biotechadv Research review paper Strain engineering for microbial production of value-added chemicals and T fuels from glycerol ⁎ Adam W. Westbrook , Dragan Miscevic, Shane Kilpatrick, Mark R. Bruder, Murray Moo-Young, ⁎ C. Perry Chou Department of Chemical Engineering, Waterloo, Ontario, Canada ARTICLE INFO ABSTRACT Keyword: While the widespread reliance on fossil fuels is driven by their low cost and relative abundance, this fossil-based Glycerol economy has been deemed unsustainable and, therefore, the adoption of sustainable and environmentally Metabolic engineering compatible energy sources is on the horizon. Biorefinery is an emerging approach that integrates metabolic Biorefinery engineering, synthetic biology, and systems biology principles for the development of whole-cell catalytic Biofuels platforms for biomanufacturing. Due to the high degree of reduction and low cost, glycerol, either refined or E. coli crude, has been recognized as an ideal feedstock for the production of value-added biologicals, though microbial Clostridium Klebsiella dissimilation of glycerol sometimes can be difficult particularly under anaerobic conditions. While strain de- Citrobacter velopment for glycerol biorefinery is widely reported in the literature, few, if any, commercialized bioprocesses Lactobacillus have been developed as a result, such that engineering of glycerol metabolism in microbial hosts remains an untapped opportunity in biomanufacturing. Here we review the recent progress made in engineering microbial hosts for the production of biofuels, diols, organic acids, biopolymers, and specialty chemicals from glycerol. We begin with a broad outline of the major pathways for fermentative and respiratory glycerol dissimilation and key end metabolites, and then focus our analysis on four key genera of bacteria known to naturally dissimilate glycerol, i.e. -

Product Sheet Info
Product Information Sheet for NR-8065 Francisella tularensis subsp. novicida, Packaging/Storage: “Two-Allele” Transposon Mutant Library, NR-8065 was packaged aseptically in 96-well plates. The product is provided frozen and should be stored at -60°C or Plate 31 (tnfn1_pw060510p03) colder immediately upon arrival. For long-term storage, the vapor phase of a liquid nitrogen freezer is recommended. Catalog No. NR-8065 Freeze-thaw cycles should be avoided. For research use only. Not for human use. Growth Conditions: Media: Tryptic Soy agar containing 0.1% L-cysteine and 10 µg/mL Contributor: kanamycin Colin Manoil, Ph.D., Professor of Genome Sciences, Incubation: University of Washington, Seattle, Washington, USA Temperature: 37°C Atmosphere: Aerobic with 5% CO2 Manufacturer: Propagation: BEI Resources 1. Scrape top of frozen well with a pipette tip and streak onto agar plate. Product Description: 2. Incubate the plate at 37°C for 24 to 48 hours. 1 A comprehensive 16508-member transposon mutant library of sequence-defined transposon insertion mutants of Citation: Francisella tularensis subsp. novicida, strain U112 was Acknowledgment for publications should read “The following prepared to allow the systematic identification of virulence reagent was obtained through BEI Resources, NIAID, NIH: determinants and other factors associated with Francisella Francisella tularensis subsp. novicida, “Two-Allele” pathogenesis. Genes refractory to insertional inactivation Transposon Mutant Library, Plate 31 (tnfn1_pw060510p03), helped define the genes essential for viability of the organism. NR-8065.” To facilitate genome-scale screening using the mutant Biosafety Level: 2 collection, a “two-allele” single-colony purified sublibrary, Appropriate safety procedures should always be used with this made up of approximately two purified mutants per gene, was material. -

History of Macromolecular Crystallography in India Through
FOREWORD Structural biology is concerned with the structures of and the structure- function relationship in biomolecules. Macromolecular crystallography is central to structural biology and indeed to modern biology as a whole. Although exploratory efforts in the field were initiated as early as in 1934 by J.D. Bernal and Dorothy Hodgkin (then Crowfoot), substantive results began to appear only by around 1960 with the structure solution of hemoglobin and myoglobin by the groups of Max Perutz and John Kendrew respectively. In an outstanding parallel computational/bioinformatics effort, G.N. Ramachandran and his colleagues at Madras developed what subsequently came to be known as the Ramachandran map which even now remains as the simplest descriptor and tool for validation of protein structures. However, initiation of macromolecular crystallographic studies in India had to wait two more decades. A few Indians have been involved in the early macromolecular crystallography projects abroad. From among them, I was the first to return to India. When I rejoined the Indian Institute of Science, Bangalore in 1971 after participating in the structure solution of insulin in Dorothy Hodgkin’s laboratory at Oxford, I would have liked nothing more than to straightway start macromolecular crystallography studies in the laboratory, but the resources available then were not even enough to carry out preliminary investigations in the area. I therefore concentrated on studies involving small molecules which are relevant to protein structure, and more so to chemical evolution and origin of life, while at the same time looking for opportunities to initiate macromolecular work. In the meantime, attempts to start macromolecular crystallography were initiated in the late seventies at the Bhabha Atomic Research Centre, Mumbai as well. -
Awakening a Latent Carbon Fixation Cycle in Escherichia Coli
bioRxiv preprint doi: https://doi.org/10.1101/2020.05.18.102244; this version posted May 19, 2020. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY-NC-ND 4.0 International license. Awakening a latent carbon fixation cycle in Escherichia coli Ari Satanowski1‡, Beau Dronsella1‡, Elad Noor2, Bastian Vögeli3, Hai He1, Philipp Wichmann1, Tobias J. Erb3,4, Steffen N. Lindner1*, Arren Bar-Even1* 1Max Planck Institute of Molecular Plant Physiology, Am Mühlenberg 1, 14476 Potsdam, Germany. 2Institute of Molecular Systems Biology, ETH Zürich, Otto-Stern-Weg 3, 8093 Zürich, Switzerland. 3Max Planck Institute for Terrestrial Microbiology, Karl-von-Frisch-Straße 10, 35043 Marburg, Germany. 4Center for Synthetic Microbiology (SYNMIKRO), 35043 Marburg, Germany. ‡ These authors contributed equally to this study. * Corresponding authors: Arren Bar-Even: phone: +49 331 567-8910; Email: [email protected] Steffen N. Lindner: phone: +49 331 567-8149; Email: [email protected] Keywords: metabolic engineering; synthetic biology; reductive carboxylation; NADPH biosynthesis; modular engineering approach. 1 bioRxiv preprint doi: https://doi.org/10.1101/2020.05.18.102244; this version posted May 19, 2020. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY-NC-ND 4.0 International license. Abstract Carbon fixation is one of the most important biochemical processes.