Machado Renatoassis M.Pdf
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-
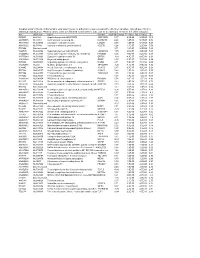
Supplementary File 2A Revised
Supplementary file 2A. Differentially expressed genes in aldosteronomas compared to all other samples, ranked according to statistical significance. Missing values were not allowed in aldosteronomas, but to a maximum of five in the other samples. Acc UGCluster Name Symbol log Fold Change P - Value Adj. P-Value B R99527 Hs.8162 Hypothetical protein MGC39372 MGC39372 2,17 6,3E-09 5,1E-05 10,2 AA398335 Hs.10414 Kelch domain containing 8A KLHDC8A 2,26 1,2E-08 5,1E-05 9,56 AA441933 Hs.519075 Leiomodin 1 (smooth muscle) LMOD1 2,33 1,3E-08 5,1E-05 9,54 AA630120 Hs.78781 Vascular endothelial growth factor B VEGFB 1,24 1,1E-07 2,9E-04 7,59 R07846 Data not found 3,71 1,2E-07 2,9E-04 7,49 W92795 Hs.434386 Hypothetical protein LOC201229 LOC201229 1,55 2,0E-07 4,0E-04 7,03 AA454564 Hs.323396 Family with sequence similarity 54, member B FAM54B 1,25 3,0E-07 5,2E-04 6,65 AA775249 Hs.513633 G protein-coupled receptor 56 GPR56 -1,63 4,3E-07 6,4E-04 6,33 AA012822 Hs.713814 Oxysterol bining protein OSBP 1,35 5,3E-07 7,1E-04 6,14 R45592 Hs.655271 Regulating synaptic membrane exocytosis 2 RIMS2 2,51 5,9E-07 7,1E-04 6,04 AA282936 Hs.240 M-phase phosphoprotein 1 MPHOSPH -1,40 8,1E-07 8,9E-04 5,74 N34945 Hs.234898 Acetyl-Coenzyme A carboxylase beta ACACB 0,87 9,7E-07 9,8E-04 5,58 R07322 Hs.464137 Acyl-Coenzyme A oxidase 1, palmitoyl ACOX1 0,82 1,3E-06 1,2E-03 5,35 R77144 Hs.488835 Transmembrane protein 120A TMEM120A 1,55 1,7E-06 1,4E-03 5,07 H68542 Hs.420009 Transcribed locus 1,07 1,7E-06 1,4E-03 5,06 AA410184 Hs.696454 PBX/knotted 1 homeobox 2 PKNOX2 1,78 2,0E-06 -

(SLC3A2) Sustains Amino Acid and Nucleotide Availability for Cell Cycle
www.nature.com/scientificreports OPEN CD98hc (SLC3A2) sustains amino acid and nucleotide availability for cell cycle progression Received: 16 April 2019 Sara Cano-Crespo1, Josep Chillarón2, Alexandra Junza3,4, Gonzalo Fernández-Miranda1, Accepted: 13 September 2019 Judit García5,6, Christine Polte7, Laura R. de la Ballina 8,9, Zoya Ignatova7, Óscar Yanes 3,4, Published: xx xx xxxx Antonio Zorzano 1,4,10, Camille Stephan-Otto Attolini 1 & Manuel Palacín1,6,10 CD98 heavy chain (CD98hc) forms heteromeric amino acid (AA) transporters by interacting with diferent light chains. Cancer cells overexpress CD98hc-transporters in order to meet their increased nutritional and antioxidant demands, since they provide branched-chain AA (BCAA) and aromatic AA (AAA) availability while protecting cells from oxidative stress. Here we show that BCAA and AAA shortage phenocopies the inhibition of mTORC1 signalling, protein synthesis and cell proliferation caused by CD98hc ablation. Furthermore, our data indicate that CD98hc sustains glucose uptake and glycolysis, and, as a consequence, the pentose phosphate pathway (PPP). Thus, loss of CD98hc triggers a dramatic reduction in the nucleotide pool, which leads to replicative stress in these cells, as evidenced by the enhanced DNA Damage Response (DDR), S-phase delay and diminished rate of mitosis, all recovered by nucleoside supplementation. In addition, proper BCAA and AAA availability sustains the expression of the enzyme ribonucleotide reductase. In this regard, BCAA and AAA shortage results in decreased content of deoxynucleotides that triggers replicative stress, also recovered by nucleoside supplementation. On the basis of our fndings, we conclude that CD98hc plays a central role in AA and glucose cellular nutrition, redox homeostasis and nucleotide availability, all key for cell proliferation. -

Lineage-Specific Programming Target Genes Defines Potential for Th1 Temporal Induction Pattern of STAT4
Downloaded from http://www.jimmunol.org/ by guest on October 1, 2021 is online at: average * The Journal of Immunology published online 26 August 2009 from submission to initial decision 4 weeks from acceptance to publication J Immunol http://www.jimmunol.org/content/early/2009/08/26/jimmuno l.0901411 Temporal Induction Pattern of STAT4 Target Genes Defines Potential for Th1 Lineage-Specific Programming Seth R. Good, Vivian T. Thieu, Anubhav N. Mathur, Qing Yu, Gretta L. Stritesky, Norman Yeh, John T. O'Malley, Narayanan B. Perumal and Mark H. Kaplan Submit online. Every submission reviewed by practicing scientists ? is published twice each month by http://jimmunol.org/subscription Submit copyright permission requests at: http://www.aai.org/About/Publications/JI/copyright.html Receive free email-alerts when new articles cite this article. Sign up at: http://jimmunol.org/alerts http://www.jimmunol.org/content/suppl/2009/08/26/jimmunol.090141 1.DC1 Information about subscribing to The JI No Triage! Fast Publication! Rapid Reviews! 30 days* • Why • • Material Permissions Email Alerts Subscription Supplementary The Journal of Immunology The American Association of Immunologists, Inc., 1451 Rockville Pike, Suite 650, Rockville, MD 20852 Copyright © 2009 by The American Association of Immunologists, Inc. All rights reserved. Print ISSN: 0022-1767 Online ISSN: 1550-6606. This information is current as of October 1, 2021. Published August 26, 2009, doi:10.4049/jimmunol.0901411 The Journal of Immunology Temporal Induction Pattern of STAT4 Target Genes Defines Potential for Th1 Lineage-Specific Programming1 Seth R. Good,2* Vivian T. Thieu,2† Anubhav N. Mathur,† Qing Yu,† Gretta L. -

Abnormal Spermatogenesis and Reduced Fertility in Transition Nuclear Protein 1-Deficient Mice
Abnormal spermatogenesis and reduced fertility in transition nuclear protein 1-deficient mice Y. Eugene Yu*†,Yun Zhang*, Emmanual Unni*‡, Cynthia R. Shirley*, Jian M. Deng§, Lonnie D. Russell¶, Michael M. Weil*, Richard R. Behringer§, and Marvin L. Meistrich*ʈ Departments of *Experimental Radiation Oncology, and §Molecular Genetics, University of Texas M. D. Anderson Cancer Center, Houston, TX 77030-4095; and ¶Department of Physiology, Southern Illinois University, School of Medicine, Carbondale, IL 62901 Edited by Richard D. Palmiter, University of Washington School of Medicine, Seattle, WA, and approved February 22, 2000 (received for review May 3, 1999) Transition nuclear proteins (TPs), the major proteins found in (15), suggesting some functional relationship between the three chromatin of condensing spermatids, are believed to be important proteins exists. Tnp1, however, is on a separate chromosome and for histone displacement and chromatin condensation during is not clearly related to the other three proteins. mammalian spermatogenesis. We generated mice lacking the ma- In vitro, TP1 decreases the melting temperature of DNA (16) jor TP, TP1, by targeted deletion of the Tnp1 gene in mouse and relaxes the DNA in nucleosomal core particles (17), which embryonic stem cells. Surprisingly, testis weights and sperm pro- led to the proposal that TP1 reduces the interaction of DNA duction were normal in the mutant mice, and only subtle abnor- with the nucleosome core. In contrast, TP2 increases the malities were observed in sperm morphology. Electron microscopy melting temperature of DNA and compacts the DNA in revealed large rod-like structures in the chromatin of mutant step nucleosomal cores, suggesting that it is a DNA-condensing 13 spermatids, in contrast to the fine chromatin fibrils observed in protein (18). -

Antibodies Products
Chapter 2 : Gentaur Products List • Human Signal peptidase complex catalytic subunit • Human Sjoegren syndrome nuclear autoantigen 1 SSNA1 • Human Small proline rich protein 2A SPRR2A ELISA kit SEC11A SEC11A ELISA kit SpeciesHuman ELISA kit SpeciesHuman SpeciesHuman • Human Signal peptidase complex catalytic subunit • Human Sjoegren syndrome scleroderma autoantigen 1 • Human Small proline rich protein 2B SPRR2B ELISA kit SEC11C SEC11C ELISA kit SpeciesHuman SSSCA1 ELISA kit SpeciesHuman SpeciesHuman • Human Signal peptidase complex subunit 1 SPCS1 ELISA • Human Ski oncogene SKI ELISA kit SpeciesHuman • Human Small proline rich protein 2D SPRR2D ELISA kit kit SpeciesHuman • Human Ski like protein SKIL ELISA kit SpeciesHuman SpeciesHuman • Human Signal peptidase complex subunit 2 SPCS2 ELISA • Human Skin specific protein 32 C1orf68 ELISA kit • Human Small proline rich protein 2E SPRR2E ELISA kit kit SpeciesHuman SpeciesHuman SpeciesHuman • Human Signal peptidase complex subunit 3 SPCS3 ELISA • Human SLAIN motif containing protein 1 SLAIN1 ELISA kit • Human Small proline rich protein 2F SPRR2F ELISA kit kit SpeciesHuman SpeciesHuman SpeciesHuman • Human Signal peptide CUB and EGF like domain • Human SLAIN motif containing protein 2 SLAIN2 ELISA kit • Human Small proline rich protein 2G SPRR2G ELISA kit containing protein 2 SCUBE2 ELISA kit SpeciesHuman SpeciesHuman SpeciesHuman • Human Signal peptide CUB and EGF like domain • Human SLAM family member 5 CD84 ELISA kit • Human Small proline rich protein 3 SPRR3 ELISA kit containing protein -

Supp Table 6.Pdf
Supplementary Table 6. Processes associated to the 2037 SCL candidate target genes ID Symbol Entrez Gene Name Process NM_178114 AMIGO2 adhesion molecule with Ig-like domain 2 adhesion NM_033474 ARVCF armadillo repeat gene deletes in velocardiofacial syndrome adhesion NM_027060 BTBD9 BTB (POZ) domain containing 9 adhesion NM_001039149 CD226 CD226 molecule adhesion NM_010581 CD47 CD47 molecule adhesion NM_023370 CDH23 cadherin-like 23 adhesion NM_207298 CERCAM cerebral endothelial cell adhesion molecule adhesion NM_021719 CLDN15 claudin 15 adhesion NM_009902 CLDN3 claudin 3 adhesion NM_008779 CNTN3 contactin 3 (plasmacytoma associated) adhesion NM_015734 COL5A1 collagen, type V, alpha 1 adhesion NM_007803 CTTN cortactin adhesion NM_009142 CX3CL1 chemokine (C-X3-C motif) ligand 1 adhesion NM_031174 DSCAM Down syndrome cell adhesion molecule adhesion NM_145158 EMILIN2 elastin microfibril interfacer 2 adhesion NM_001081286 FAT1 FAT tumor suppressor homolog 1 (Drosophila) adhesion NM_001080814 FAT3 FAT tumor suppressor homolog 3 (Drosophila) adhesion NM_153795 FERMT3 fermitin family homolog 3 (Drosophila) adhesion NM_010494 ICAM2 intercellular adhesion molecule 2 adhesion NM_023892 ICAM4 (includes EG:3386) intercellular adhesion molecule 4 (Landsteiner-Wiener blood group)adhesion NM_001001979 MEGF10 multiple EGF-like-domains 10 adhesion NM_172522 MEGF11 multiple EGF-like-domains 11 adhesion NM_010739 MUC13 mucin 13, cell surface associated adhesion NM_013610 NINJ1 ninjurin 1 adhesion NM_016718 NINJ2 ninjurin 2 adhesion NM_172932 NLGN3 neuroligin -
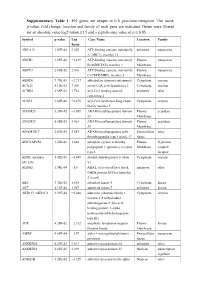
Supplementary Table 1
Supplementary Table 1. 492 genes are unique to 0 h post-heat timepoint. The name, p-value, fold change, location and family of each gene are indicated. Genes were filtered for an absolute value log2 ration 1.5 and a significance value of p ≤ 0.05. Symbol p-value Log Gene Name Location Family Ratio ABCA13 1.87E-02 3.292 ATP-binding cassette, sub-family unknown transporter A (ABC1), member 13 ABCB1 1.93E-02 −1.819 ATP-binding cassette, sub-family Plasma transporter B (MDR/TAP), member 1 Membrane ABCC3 2.83E-02 2.016 ATP-binding cassette, sub-family Plasma transporter C (CFTR/MRP), member 3 Membrane ABHD6 7.79E-03 −2.717 abhydrolase domain containing 6 Cytoplasm enzyme ACAT1 4.10E-02 3.009 acetyl-CoA acetyltransferase 1 Cytoplasm enzyme ACBD4 2.66E-03 1.722 acyl-CoA binding domain unknown other containing 4 ACSL5 1.86E-02 −2.876 acyl-CoA synthetase long-chain Cytoplasm enzyme family member 5 ADAM23 3.33E-02 −3.008 ADAM metallopeptidase domain Plasma peptidase 23 Membrane ADAM29 5.58E-03 3.463 ADAM metallopeptidase domain Plasma peptidase 29 Membrane ADAMTS17 2.67E-04 3.051 ADAM metallopeptidase with Extracellular other thrombospondin type 1 motif, 17 Space ADCYAP1R1 1.20E-02 1.848 adenylate cyclase activating Plasma G-protein polypeptide 1 (pituitary) receptor Membrane coupled type I receptor ADH6 (includes 4.02E-02 −1.845 alcohol dehydrogenase 6 (class Cytoplasm enzyme EG:130) V) AHSA2 1.54E-04 −1.6 AHA1, activator of heat shock unknown other 90kDa protein ATPase homolog 2 (yeast) AK5 3.32E-02 1.658 adenylate kinase 5 Cytoplasm kinase AK7 -

Qt38n028mr Nosplash A3e1d84
! ""! ACKNOWLEDGEMENTS I dedicate this thesis to my parents who inspired me to become a scientist through invigorating scientific discussions at the dinner table even when I was too young to understand what the hippocampus was. They also prepared me for the ups and downs of science and supported me through all of these experiences. I would like to thank my advisor Dr. Elizabeth Blackburn and my thesis committee members Dr. Eric Verdin, and Dr. Emmanuelle Passegue. Liz created a nurturing and supportive environment for me to explore my own ideas, while at the same time teaching me how to love science, test my questions, and of course provide endless ways to think about telomeres and telomerase. Eric and Emmanuelle both gave specific critical advice about the proper experiments for T cells and both volunteered their lab members for further critical advice. I always felt inspired with a sense of direction after thesis committee meetings. The Blackburn lab is full of smart and dedicated scientists whom I am thankful for their support. Specifically Dr. Shang Li and Dr. Brad Stohr for their stimulating scientific debates and “arguments.” Dr. Jue Lin, Dana Smith, Kyle Lapham, Dr. Tet Matsuguchi, and Kyle Jay for their friendships and discussions about what my data could possibly mean. Dr. Eva Samal for teaching me molecular biology techniques and putting up with my late night lab exercises. Beth Cimini for her expertise with microscopy, FACs, singing, and most of all for being a caring and supportive friend. Finally, I would like to thank Dr. Imke Listerman, my scientific partner for most of the breast cancer experiments. -

Functional Enrichments of Disease Variants Indicate Hundreds of Independent Loci Across Eight Diseases
Functional enrichments of disease variants indicate hundreds of independent loci across eight diseases Abhishek K. Sarkar, Lucas D. Ward, & Manolis Kellis 1.00 0.75 Cohort correlation !"SS 0.50 #AN"$" %&!" N"!"C1 N"!"C2 Pearson 0.25 'verall )TC## 0.00 Hold-out -0.25 0 25000 50000 75000 100000 Top n SNPs (full meta-analysis) Supplementary Figure 1: Correlation between individual cohort 푧-scores and meta-analyzed 푧- scores of the remainder in a study of rheumatoid arthritis considering increasing number of SNPs. SNPs are ranked by 푝-value in the overall meta-analysis. Overall correlation is between sample-size weighted 푧-scores and published inverse-variance weighted 푧-scores. 1 15-state model, 5 marks, 127 epigenomes Cell type/ tissue group Epigenome name Addtl marks H3K4me1 H3K4me3 H3K36me3 H3K27me3 H3K9me3 H3K27ac H3K9ac DNase-Seq DNA methyl RNA-Seq EID states Chrom. E017 IMR90 fetal lung fibroblasts Cell Line 21 IMR90 E002 ES-WA7 Cell Line E008 H9 Cell Line 21 E001 ES-I3 Cell Line E015 HUES6 Cell Line ESC E014 HUES48 Cell Line E016 HUES64 Cell Line E003 H1 Cell Line 20 E024 ES-UCSF4 Cell Line E020 iPS-20b Cell Line E019 iPS-18 Cell Line iPSC E018 iPS-15b Cell Line E021 iPS DF 6.9 Cell Line E022 iPS DF 19.11 Cell Line E007 H1 Derived Neuronal Progenitor Cultured Cells 13 E009 H9 Derived Neuronal Progenitor Cultured Cells 1 E010 H9 Derived Neuron Cultured Cells 1 E013 hESC Derived CD56+ Mesoderm Cultured Cells ES-deriv E012 hESC Derived CD56+ Ectoderm Cultured Cells E011 hESC Derived CD184+ Endoderm Cultured Cells E004 H1 BMP4 Derived Mesendoderm Cultured Cells 11 E005 H1 BMP4 Derived Trophoblast Cultured Cells 15 E006 H1 Derived Mesenchymal Stem Cells 13 E062 Primary mononuclear cells from peripheral blood E034 Primary T cells from peripheral blood E045 Prim. -

Epigenetic Mechanisms Are Involved in the Oncogenic Properties of ZNF518B in Colorectal Cancer
Epigenetic mechanisms are involved in the oncogenic properties of ZNF518B in colorectal cancer Francisco Gimeno-Valiente, Ángela L. Riffo-Campos, Luis Torres, Noelia Tarazona, Valentina Gambardella, Andrés Cervantes, Gerardo López-Rodas, Luis Franco and Josefa Castillo SUPPLEMENTARY METHODS 1. Selection of genomic sequences for ChIP analysis To select the sequences for ChIP analysis in the five putative target genes, namely, PADI3, ZDHHC2, RGS4, EFNA5 and KAT2B, the genomic region corresponding to the gene was downloaded from Ensembl. Then, zoom was applied to see in detail the promoter, enhancers and regulatory sequences. The details for HCT116 cells were then recovered and the target sequences for factor binding examined. Obviously, there are not data for ZNF518B, but special attention was paid to the target sequences of other zinc-finger containing factors. Finally, the regions that may putatively bind ZNF518B were selected and primers defining amplicons spanning such sequences were searched out. Supplementary Figure S3 gives the location of the amplicons used in each gene. 2. Obtaining the raw data and generating the BAM files for in silico analysis of the effects of EHMT2 and EZH2 silencing The data of siEZH2 (SRR6384524), siG9a (SRR6384526) and siNon-target (SRR6384521) in HCT116 cell line, were downloaded from SRA (Bioproject PRJNA422822, https://www.ncbi. nlm.nih.gov/bioproject/), using SRA-tolkit (https://ncbi.github.io/sra-tools/). All data correspond to RNAseq single end. doBasics = TRUE doAll = FALSE $ fastq-dump -I --split-files SRR6384524 Data quality was checked using the software fastqc (https://www.bioinformatics.babraham. ac.uk /projects/fastqc/). The first low quality removing nucleotides were removed using FASTX- Toolkit (http://hannonlab.cshl.edu/fastxtoolkit/). -

Supplemental Methods Definition of Clinical Outcomes and Statistics
Supplemental methods Definition of clinical outcomes and statistics Overall survival was defined from time of diagnosis until death or last follow up. Time to locoregional recurrence (LRR) or distant metastasis (DM) was defined as time from diagnosis until either an event or last follow up. Clinical variables examined included tumor stage, nodal stage and primary tumor site. Univariate analysis was performed using Cox-regression (SPSS v25). Kaplan Meier curves were generated, and group comparisons were performed using log-rank statistics. In vivo shRNA screening Each library was cloned into the pRSI17 vector (Cellecta) and packed into lentivirus particles. HNSCC cell lines were infected in vitro through spinfection with virus containing the library at a low MOI (~20% infected cells as measured by flow cytometry) in order to minimize superinfection of cells. Cells were selected with puromycin for at least 2 days and grown in vitro for <3 population doublings prior to injection of 4 million cells subcutaneously in nude mouse flank. An additional 2 million cells from the day of injection were collected as a frozen reference cell pellet. Resulting xenografts were treated with 2 Gy/day of radiation once the tumor had reached approximately 100 mm3 to a total dose of 6-10 Gy depending upon the model. Following treatment the tumors were allowed to grow for approximately 2 weeks (volume ~ 500mm3). DNA was isolated from tumor and reference cells, amplified, and sequenced on Illumina sequencers as previously described 12. Hairpin counts were normalized to counts per million (CPM) per sample to enable comparison across samples. For each sample, (log2) fold-change of each hairpin in the tumor was calculated compared to the level in the reference pellet. -

Induction of Therapeutic Tissue Tolerance Foxp3 Expression Is
Downloaded from http://www.jimmunol.org/ by guest on October 2, 2021 is online at: average * The Journal of Immunology , 13 of which you can access for free at: 2012; 189:3947-3956; Prepublished online 17 from submission to initial decision 4 weeks from acceptance to publication September 2012; doi: 10.4049/jimmunol.1200449 http://www.jimmunol.org/content/189/8/3947 Foxp3 Expression Is Required for the Induction of Therapeutic Tissue Tolerance Frederico S. Regateiro, Ye Chen, Adrian R. Kendal, Robert Hilbrands, Elizabeth Adams, Stephen P. Cobbold, Jianbo Ma, Kristian G. Andersen, Alexander G. Betz, Mindy Zhang, Shruti Madhiwalla, Bruce Roberts, Herman Waldmann, Kathleen F. Nolan and Duncan Howie J Immunol cites 35 articles Submit online. Every submission reviewed by practicing scientists ? is published twice each month by Submit copyright permission requests at: http://www.aai.org/About/Publications/JI/copyright.html Receive free email-alerts when new articles cite this article. Sign up at: http://jimmunol.org/alerts http://jimmunol.org/subscription http://www.jimmunol.org/content/suppl/2012/09/17/jimmunol.120044 9.DC1 This article http://www.jimmunol.org/content/189/8/3947.full#ref-list-1 Information about subscribing to The JI No Triage! Fast Publication! Rapid Reviews! 30 days* Why • • • Material References Permissions Email Alerts Subscription Supplementary The Journal of Immunology The American Association of Immunologists, Inc., 1451 Rockville Pike, Suite 650, Rockville, MD 20852 Copyright © 2012 by The American Association of Immunologists, Inc. All rights reserved. Print ISSN: 0022-1767 Online ISSN: 1550-6606. This information is current as of October 2, 2021.