Reyna Alejandra Jiménez Flores
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

A Web-Platform for Analysis of Host Factors Involved in Viral Infections Discovered by Genome Wide Rnai Screen
Electronic Supplementary Material (ESI) for Molecular BioSystems. This journal is © The Royal Society of Chemistry 2017 vhfRNAi: A web-platform for analysis of host factors involved in viral infections discovered by genome wide RNAi screen Anamika Thakur#, Abid Qureshi# and Manoj Kumar* Bioinformatics Centre, Institute of Microbial Technology, Council of Scientific and Industrial Research, Sector 39A, Chandigarh-160036, India #Equal contribution * To whom correspondence should be addressed. Tel, 91-172-6665453; Fax, 91-172- 2690585; 91-172-2690632; Email, [email protected] Supplementary Tables Table S1: Statistics of unique and duplicate host factors in each virus Table S2: Table denoting genes common among different viruses Table S3: Statistics of GWAS analysis Table S1. Statistics of unique and duplicate host factors in each virus S. No. Virus Unique-Entries Duplicate-Entries 1. Adeno-associated virus (AAV) 926 533 2. Avian influenza virus (AIV) 0 11 3. Borna disease virus (BDV) 14 20 4. Dengue virus 2 (DEN-2) 27 13 5. Hepatitis C virus (HCV) 236 213 6. Human immunodeficiency virus 1 (HIV) 1388 857 7. Human parainfluenza virus 3 (HPIV-3) 0 27 8. Human herpesvirus 1 (HSV-1) 34 38 9. Influenza A virus (IAV) 700 513 10. Lymphocytic choriomeningitis virus (LCMV) 0 54 11. Marburgvirus (MARV) 0 11 12. Poliovirus (PV) 3340 1035 13. Rotavirus (RV) 347 175 14. Sendai virus (SeV) 32 27 15. Sindbis virus (SIV) 70 41 16. Vaccinia virus (VACV) 482 296 17. Vesicular stomatitis virus (VSV) 9 78 18. West Nile virus (WNV) 313 137 Table S1. Statistics of unique host factors for each virus having overlapping and unique factors in different viruses Non-overlap – Overall- S. -

TAS2R20 Variants Confer Dietary Adaptation to High-Quercitrin Bamboo Leaves in Qinling Giant Pandas
TAS2R20 variants confer dietary adaptation to high-quercitrin bamboo leaves in Qinling giant pandas Xiangxu Hu1, Guan Wang2, Lei Shan3, Shuyan Sun1, Yibo Hu1, and Fuwen Wei1 1Institute of Zoology Chinese Academy of Sciences 2Affiliation not available 3Nanjing Normal University College of Life Sciences May 5, 2020 Abstract Abstract: Sensitivity to bitter tastes provides animals with an important means of interacting with their environment and thus, influences their dietary preferences. Genetic variants encoding functionally distinct receptor types contribute to variation in bitter taste sensitivity. Our previous study showed that two nonsynonymous sites, A52V and Q296H, in the TAS2R20 gene are directionally selected in giant pandas from the Qinling Mountains, which are speculated to be the causative base-pair changes of Qinling pandas for the higher preference for bamboo leaves in comparison with other pandas. Here, we used functional expression in engineered cells to identify agonists of pTAS2R20 (i.e. giant panda's TAS2R20) and interrogated the differences in perception in the in vitro responses of pTAS2R20 variants to the agonists. Our results show that pTAS2R20 is specifically activated by quercitrin and that pTAS2R20 variants exhibit differences in the sensitivity of their response to the agonist. Compared to pTAS2R20 in pandas from other areas, the receptor variant with A52V and Q296H, which is most commonly found in Qinling pandas, confers a significantly decreased sensitivity to quercitrin. We subsequently quantified the quercitrin content of the leaves of bamboo distributed in the Qinling Mountains, which was found to be significantly higher than that of the leaves of bamboo from panda habitats in other areas. -

Supplementary Table S4. FGA Co-Expressed Gene List in LUAD
Supplementary Table S4. FGA co-expressed gene list in LUAD tumors Symbol R Locus Description FGG 0.919 4q28 fibrinogen gamma chain FGL1 0.635 8p22 fibrinogen-like 1 SLC7A2 0.536 8p22 solute carrier family 7 (cationic amino acid transporter, y+ system), member 2 DUSP4 0.521 8p12-p11 dual specificity phosphatase 4 HAL 0.51 12q22-q24.1histidine ammonia-lyase PDE4D 0.499 5q12 phosphodiesterase 4D, cAMP-specific FURIN 0.497 15q26.1 furin (paired basic amino acid cleaving enzyme) CPS1 0.49 2q35 carbamoyl-phosphate synthase 1, mitochondrial TESC 0.478 12q24.22 tescalcin INHA 0.465 2q35 inhibin, alpha S100P 0.461 4p16 S100 calcium binding protein P VPS37A 0.447 8p22 vacuolar protein sorting 37 homolog A (S. cerevisiae) SLC16A14 0.447 2q36.3 solute carrier family 16, member 14 PPARGC1A 0.443 4p15.1 peroxisome proliferator-activated receptor gamma, coactivator 1 alpha SIK1 0.435 21q22.3 salt-inducible kinase 1 IRS2 0.434 13q34 insulin receptor substrate 2 RND1 0.433 12q12 Rho family GTPase 1 HGD 0.433 3q13.33 homogentisate 1,2-dioxygenase PTP4A1 0.432 6q12 protein tyrosine phosphatase type IVA, member 1 C8orf4 0.428 8p11.2 chromosome 8 open reading frame 4 DDC 0.427 7p12.2 dopa decarboxylase (aromatic L-amino acid decarboxylase) TACC2 0.427 10q26 transforming, acidic coiled-coil containing protein 2 MUC13 0.422 3q21.2 mucin 13, cell surface associated C5 0.412 9q33-q34 complement component 5 NR4A2 0.412 2q22-q23 nuclear receptor subfamily 4, group A, member 2 EYS 0.411 6q12 eyes shut homolog (Drosophila) GPX2 0.406 14q24.1 glutathione peroxidase -

Effect of Genetic Variation on Salt, Sweet, Fat and Bitter Taste
Effect of Genetic Variation on Salt, Sweet, Fat and Bitter Taste by Andre Dias A thesis submitted in conformity with the requirements for the degree of Doctor of Philosophy Department of Nutritional Sciences University of Toronto © Copyright by Andre Dias 2014 Effect of Genetic Variation on Salt, Sweet, Fat and Bitter Taste Andre Dias Doctor of Philosophy Department of Nutritional Sciences University of Toronto 2014 Abstract Background: Taste is one of the primary determinants of food intake and taste function can be influenced by a number of factors including genetics. However, little is known about the relationship between genetic variation, taste function, food preference and intake. Objective: To examine the effect of variation in genes involved in the perception of salt, sweet, fat and bitter compounds on taste function, food preference and consumption. Methods: Subjects were drawn from the Toronto Nutrigenomics and Health Study, a population of healthy men (n=487) and women (n = 1058). Dietary intake was assessed using a 196-item food frequency questionnaire (FFQ) and food preference was assessed using a 63-item food preference checklist. Subsets of individuals were phenotyped to assess taste function in response to salt (n=95), sucrose (n=95), oleic acid (n=21) and naringin (n=685) stimuli. Subjects were genotyped for Single Nucleotide Polymorphisms (SNPs) in candidate genes. Results: Of the SNPs examined in putative salt taste receptor genes (SCNN1(A, B, D, G), TRPV1), the rs9939129 and rs239345 SNPs in the SCNN1B gene and rs8065080 in the TRPV1 gene were associated with salt taste. In the TAS1R2 gene, the rs12033832 was associated with sucrose taste and sugar intake. -

Supplementary Tables Supplemental Table S1: Comparison of the Coverage of Reference Panels Used for SNP Imputation. the Markers
Supplementary Tables Supplemental Table S1: Comparison of the coverage of reference panels used for SNP imputation. The markers on the Consortium on Asthma among African-ancestry Populations in the Americas (CAAPA) reference panel and Haplotype Reference Consortium (HRG) used to impute SNPs from our AA and CAU participants were compared to one thousand genomes (1kG) dataset. Loci information CAAPA vs 1kG HRC vs 1kG Total loci 45,639,158 90,558,388 Overlapping loci 24,880,301 49,826,569 Percent overlap 54.52% 55.02% 1kG-only loci 9,363,544 15,148,191 Ref-only loci 8,461,186 22,649,501 Supplemental Table S2: SNP imputation results. The total number of SNPs imputed for the AA and CAU participants either using the Michigan imputation server (Minimac) or Beagle. Targets prepared for: AA CAU Beagle 730,616 726,165 Minimac 698,343 660,733 Supplemental Table S3: Allele frequencies of TA2R38 SNPs by each ancestral group and time point. The rs number of each SNP, the location of SNP buy chromosome (CHR) and base pair position (POS) is provided along with the allele frequency (ALLELE:FREQ) for each SNP. Baseline 6-month Baseline 6-month AA (N = 297) AA (N = 234) CAU (N = 198) CAU (N = 151) SNP CHR POS ALLELE:FREQ ALLELE:FREQ ALLELE:FREQ ALLELE:FREQ rs10246939 7 141672604 T:0.49 C:0.51 T:0.50 C:0.50 T:0.54 C:0.46 T:0.54 C:0.46 rs1726866 7 141672705 G:0.68 A:0.32 G:0.68 A:0.32 G:0.46 A:0.54 G:0.46 A:0.54 rs713598 7 141673345 C:0.50 G:0.50 C:0.50 G:0.50 C:0.58 G:0.42 C:0.58 G:0.42 Supplemental Table S4: Linkage disequilibrium analysis of TAS2R38 SNPs at each time point of the intervention. -

G Protein-Coupled Receptors
S.P.H. Alexander et al. The Concise Guide to PHARMACOLOGY 2015/16: G protein-coupled receptors. British Journal of Pharmacology (2015) 172, 5744–5869 THE CONCISE GUIDE TO PHARMACOLOGY 2015/16: G protein-coupled receptors Stephen PH Alexander1, Anthony P Davenport2, Eamonn Kelly3, Neil Marrion3, John A Peters4, Helen E Benson5, Elena Faccenda5, Adam J Pawson5, Joanna L Sharman5, Christopher Southan5, Jamie A Davies5 and CGTP Collaborators 1School of Biomedical Sciences, University of Nottingham Medical School, Nottingham, NG7 2UH, UK, 2Clinical Pharmacology Unit, University of Cambridge, Cambridge, CB2 0QQ, UK, 3School of Physiology and Pharmacology, University of Bristol, Bristol, BS8 1TD, UK, 4Neuroscience Division, Medical Education Institute, Ninewells Hospital and Medical School, University of Dundee, Dundee, DD1 9SY, UK, 5Centre for Integrative Physiology, University of Edinburgh, Edinburgh, EH8 9XD, UK Abstract The Concise Guide to PHARMACOLOGY 2015/16 provides concise overviews of the key properties of over 1750 human drug targets with their pharmacology, plus links to an open access knowledgebase of drug targets and their ligands (www.guidetopharmacology.org), which provides more detailed views of target and ligand properties. The full contents can be found at http://onlinelibrary.wiley.com/doi/ 10.1111/bph.13348/full. G protein-coupled receptors are one of the eight major pharmacological targets into which the Guide is divided, with the others being: ligand-gated ion channels, voltage-gated ion channels, other ion channels, nuclear hormone receptors, catalytic receptors, enzymes and transporters. These are presented with nomenclature guidance and summary information on the best available pharmacological tools, alongside key references and suggestions for further reading. -

G Protein‐Coupled Receptors
S.P.H. Alexander et al. The Concise Guide to PHARMACOLOGY 2019/20: G protein-coupled receptors. British Journal of Pharmacology (2019) 176, S21–S141 THE CONCISE GUIDE TO PHARMACOLOGY 2019/20: G protein-coupled receptors Stephen PH Alexander1 , Arthur Christopoulos2 , Anthony P Davenport3 , Eamonn Kelly4, Alistair Mathie5 , John A Peters6 , Emma L Veale5 ,JaneFArmstrong7 , Elena Faccenda7 ,SimonDHarding7 ,AdamJPawson7 , Joanna L Sharman7 , Christopher Southan7 , Jamie A Davies7 and CGTP Collaborators 1School of Life Sciences, University of Nottingham Medical School, Nottingham, NG7 2UH, UK 2Monash Institute of Pharmaceutical Sciences and Department of Pharmacology, Monash University, Parkville, Victoria 3052, Australia 3Clinical Pharmacology Unit, University of Cambridge, Cambridge, CB2 0QQ, UK 4School of Physiology, Pharmacology and Neuroscience, University of Bristol, Bristol, BS8 1TD, UK 5Medway School of Pharmacy, The Universities of Greenwich and Kent at Medway, Anson Building, Central Avenue, Chatham Maritime, Chatham, Kent, ME4 4TB, UK 6Neuroscience Division, Medical Education Institute, Ninewells Hospital and Medical School, University of Dundee, Dundee, DD1 9SY, UK 7Centre for Discovery Brain Sciences, University of Edinburgh, Edinburgh, EH8 9XD, UK Abstract The Concise Guide to PHARMACOLOGY 2019/20 is the fourth in this series of biennial publications. The Concise Guide provides concise overviews of the key properties of nearly 1800 human drug targets with an emphasis on selective pharmacology (where available), plus links to the open access knowledgebase source of drug targets and their ligands (www.guidetopharmacology.org), which provides more detailed views of target and ligand properties. Although the Concise Guide represents approximately 400 pages, the material presented is substantially reduced compared to information and links presented on the website. -

Bivariate Genome-Wide Association Analysis Strengthens the Role of Bitter Receptor Clusters on Chromosomes 7 and 12 in Human Bitter Taste
bioRxiv preprint doi: https://doi.org/10.1101/296269; this version posted April 6, 2018. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY-NC-ND 4.0 International license. Bivariate genome-wide association analysis strengthens the role of bitter receptor clusters on chromosomes 7 and 12 in human bitter taste Liang-Dar Hwang1,2,3,4, Puya Gharahkhani1, Paul A. S. Breslin5,6, Scott D. Gordon1, Gu Zhu1, Nicholas G. Martin1, Danielle R. Reed5, and Margaret J. Wright2,7 1 QIMR Berghofer Medical Research Institute, Herston, Queensland 4006, Australia 2 Queensland Brain Institute, University of Queensland, St Lucia, Queensland 4072, Australia 3 Faculty of Medicine, University of Queensland, Herston, Queensland 4006, Australia 4 University of Queensland Diamantina Institute, University of Queensland, Translational Research Institute, Woolloongabba, Queensland 4102, Australia 5 Monell Chemical Senses Center, Philadelphia, Pennsylvania 19104, USA 6 Department of Nutritional Sciences, School of Environmental and Biological Sciences, Rutgers University, New Brunswick NJ, 08901 USA 7 Centre for Advanced Imaging, University of Queensland, St Lucia, Queensland 4072, Australia Correspondence to be sent to: Liang-Dar Hwang University of Queensland Diamantina Institute Wolloongabba QLD 4102, Australia Email: [email protected] Telephone: +61 7 3443 7976 Fax: +61 7 3443 6966 1 bioRxiv preprint doi: https://doi.org/10.1101/296269; this version posted April 6, 2018. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. -
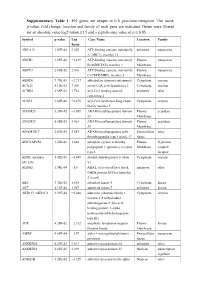
Supplementary Table 1
Supplementary Table 1. 492 genes are unique to 0 h post-heat timepoint. The name, p-value, fold change, location and family of each gene are indicated. Genes were filtered for an absolute value log2 ration 1.5 and a significance value of p ≤ 0.05. Symbol p-value Log Gene Name Location Family Ratio ABCA13 1.87E-02 3.292 ATP-binding cassette, sub-family unknown transporter A (ABC1), member 13 ABCB1 1.93E-02 −1.819 ATP-binding cassette, sub-family Plasma transporter B (MDR/TAP), member 1 Membrane ABCC3 2.83E-02 2.016 ATP-binding cassette, sub-family Plasma transporter C (CFTR/MRP), member 3 Membrane ABHD6 7.79E-03 −2.717 abhydrolase domain containing 6 Cytoplasm enzyme ACAT1 4.10E-02 3.009 acetyl-CoA acetyltransferase 1 Cytoplasm enzyme ACBD4 2.66E-03 1.722 acyl-CoA binding domain unknown other containing 4 ACSL5 1.86E-02 −2.876 acyl-CoA synthetase long-chain Cytoplasm enzyme family member 5 ADAM23 3.33E-02 −3.008 ADAM metallopeptidase domain Plasma peptidase 23 Membrane ADAM29 5.58E-03 3.463 ADAM metallopeptidase domain Plasma peptidase 29 Membrane ADAMTS17 2.67E-04 3.051 ADAM metallopeptidase with Extracellular other thrombospondin type 1 motif, 17 Space ADCYAP1R1 1.20E-02 1.848 adenylate cyclase activating Plasma G-protein polypeptide 1 (pituitary) receptor Membrane coupled type I receptor ADH6 (includes 4.02E-02 −1.845 alcohol dehydrogenase 6 (class Cytoplasm enzyme EG:130) V) AHSA2 1.54E-04 −1.6 AHA1, activator of heat shock unknown other 90kDa protein ATPase homolog 2 (yeast) AK5 3.32E-02 1.658 adenylate kinase 5 Cytoplasm kinase AK7 -

Differentially Methylated Genes
10/30/2013 Disclosures Key Rheumatoid Arthritis-Associated Pathogenic Pathways Revealed by Integrative Analysis of RA Omics Datasets Consultant: IGNYTA Funding: Rheumatology Research Foundation By John W. Whitaker, Wei Wang and Gary S. Firestein DNA methylation and gene regulation The RA methylation signature in FLS DNA methylation – DNMT1 (maintaining methylation) OA – DNMT3a, 3b (de novo methylation) RA % of CpG methylation: 0% 100% Nakano et al. 2013 ARD AA06 AANAT AARS ABCA6 ABCC12 ABCG1 ABHD8 ABL2 ABR ABRA ACACA ACAN ACAP3 ACCSL ACN9 ACOT7 ACOX2 ACP5 ACP6 ACPP ACSL1 ACSL3 ACSM5 ACVRL1 ADAM10 ADAM32 ADAM33 ADAMTS12 ADAMTS15 ADAMTS19 ADAMTS4 ADAT3 ADCK4 ADCK5 ADCY2 ADCY3 ADCY6 ADORA1 ADPGK ADPRHL1 ADTRP AFAP1 AFAP1L2 AFF3 AFG3L1P AGAP11 AGER AGTR1 AGXT AIF1L AIM2 AIRE AJUBA AK4 AKAP12 AKAP2 AKR1C2 AKR1E2 AKT2 ALAS1 ALDH1L1-AS1 ALDH3A1 ALDH3B1 ALDH8A1 ALDOB ALDOC ALOX12 ALPK3 ALS2CL ALX4 AMBRA1 AMPD2 AMPD3 ANGPT1 ANGPT2 ANGPTL5 ANGPTL6 ANK1 ANKMY2 ANKRD29 ANKRD37 ANKRD53 ANO3 ANO6 ANO7 ANP32C ANXA6 ANXA8L2 AP1G1 AP2A2 AP2M1 AP5B1 APBA2 APC APCDD1 APOBEC3B APOBEC3G APOC1 APOH APOL6 APOLD1 APOM AQP1 AQP10 AQP6 AQP9 ARAP1 ARHGAP24 ARHGAP42 ARHGEF19 ARHGEF25 ARHGEF3 ARHGEF37 ARHGEF7 ARL4C ARL6IP 5 ARL8B ARMC3 ARNTL2 ARPP21 ARRB1 ARSI ASAH2B ASB10 ASB2 ASCL2 ASIC4 ASPH ATF3 ATF7 ATL1 ATL3 ATP10A ATP1A1 ATP1A4 ATP2C1 ATP5A1 ATP5EP2 ATP5L2 ATP6V0CP3 ATP6V1C1 ATP6V1E2 ATXN7L1 ATXN7L2 AVPI1 AXIN2 B3GNT7 B3GNT8 B3GNTL1 BACH1 BAG3 Differential methylated genes in RA FLS BAIAP2L2 BANP BATF BATF2 BBS2 BCAS4 BCAT1 BCL7C BDKRB2 BEGAIN BEST1 BEST3 -

Transcriptome Analysis Reveals Upregulation of Bitter Taste Receptors in Severe Asthmatics
ORIGINAL ARTICLE ASTHMA Transcriptome analysis reveals upregulation of bitter taste receptors in severe asthmatics Christina Orsmark-Pietras1,2, Anna James2,3, Jon R. Konradsen2,4,5, Bjo¨rn Nordlund2,4,5, Cilla So¨derha¨ll1,2, Ville Pulkkinen2,3,6,7, Christophe Pedroletti2,5, Kameran Daham2,8,9, Maciek Kupczyk2,3, Barbro Dahle´n2,8,9, Juha Kere1,2,10, Sven-Erik Dahle´n2,3, Gunilla Hedlin2,4,5,11 and Erik Mele´n2,3,4,11 Affiliations: 1Dept of Biosciences and Nutrition at Novum, Karolinska Institute, Stockholm, 2The Centre for Allergy Research at Karolinska Institute, Stockholm, 3Institute of Environmental Medicine, Karolinska Institute, Stockholm, 4Astrid Lindgren Children’s Hospital, Karolinska University Hospital, Stockholm, 5Dept of Women’s and Children’s Health, Karolinska Institute, Stockholm, 8Lung/Allergy Clinic, Karolinska University Hospital Huddinge, Stockholm, 9Dept of Internal Medicine, Karolinska University Hospital Huddinge, Stockholm, and 10Science for Life Laboratory, Karolinska Institute, Stockholm, Sweden. 6Dept of Medical Genetics, Biomedicum Helsinki, University of Helsinki, Helsinki, and 7Folkha¨lsan Institute of Genetics, Helsinki, Finland. 11Both authors contributed equally. Correspondence: S-E. Dahle´n, The National Institute of Environmental Medicine, Karolinska Institutet, SE- 17177 Stockholm, Sweden. E-mail: [email protected] ABSTRACT The causes of severe childhood asthma are poorly understood. Our aim was to define global patterns of gene expression in children with severe therapy-resistant and controlled asthma. White blood cells were isolated and the global transcriptome profile was characterised using the Affymetrix Human Gene ST 1.0 chip in children with severe, therapy-resistant asthma (n517), controlled asthma (n519) and healthy controls (n518). -

Expression of Taste Receptor 2 Subtypes in Human Testis and Sperm
Journal of Clinical Medicine Article Expression of Taste Receptor 2 Subtypes in Human Testis and Sperm 1, 1, 1 1 1 Laura Governini y, Bianca Semplici y, Valentina Pavone , Laura Crifasi , Camilla Marrocco , Vincenzo De Leo 1, Elisabeth Arlt 2, Thomas Gudermann 2, Ingrid Boekhoff 2, Alice Luddi 1,* and Paola Piomboni 1 1 Department of Molecular and Developmental Medicine, Siena University, 53100 Siena, Italy; [email protected] (L.G.); [email protected] (B.S.); [email protected] (V.P.); [email protected] (L.C.); [email protected] (C.M.); [email protected] (V.D.L.); [email protected] (P.P.) 2 Walther Straub Institute of Pharmacology and Toxicology, LMU Munich, 80336 Muenchen, Germany; [email protected] (E.A.); [email protected] (T.G.); ingrid.boekhoff@lrz.uni-muenchen.de (I.B.) * Correspondence: [email protected]; Tel.: +39-0577-233521 These authors contributed equally to this work. y Received: 13 December 2019; Accepted: 14 January 2020; Published: 18 January 2020 Abstract: Taste receptors (TASRs) are expressed not only in the oral cavity but also throughout the body, thus suggesting that they may play different roles in organ systems beyond the tongue. Recent studies showed the expression of several TASRs in mammalian testis and sperm, indicating an involvement of these receptors in male gametogenesis and fertility. This notion is supported by an impaired reproductive phenotype of mouse carrying targeted deletion of taste receptor genes, as well as by a significant correlation between human semen parameters and specific polymorphisms of taste receptor genes.