'Identification of Cell Type Enriched Cofactors of NF-Κb Dependent Transcription'
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Taf7l Cooperates with Trf2 to Regulate Spermiogenesis
Taf7l cooperates with Trf2 to regulate spermiogenesis Haiying Zhoua,b, Ivan Grubisicb,c, Ke Zhengd,e, Ying Heb, P. Jeremy Wangd, Tommy Kaplanf, and Robert Tjiana,b,1 aHoward Hughes Medical Institute and bDepartment of Molecular and Cell Biology, Li Ka Shing Center for Biomedical and Health Sciences, California Institute for Regenerative Medicine Center of Excellence, University of California, Berkeley, CA 94720; cUniversity of California Berkeley–University of California San Francisco Graduate Program in Bioengineering, University of California, Berkeley, CA 94720; dDepartment of Animal Biology, University of Pennsylvania School of Veterinary Medicine, Philadelphia, PA 19104; eState Key Laboratory of Reproductive Medicine, Nanjing Medical University, Nanjing 210029, People’s Republic of China; and fSchool of Computer Science and Engineering, The Hebrew University of Jerusalem, Jerusalem 91904, Israel Contributed by Robert Tjian, September 11, 2013 (sent for review August 20, 2013) TATA-binding protein (TBP)-associated factor 7l (Taf7l; a paralogue (Taf4b; a homolog of Taf4) (9), TBP-related factor 2 (Trf2) (10, of Taf7) and TBP-related factor 2 (Trf2) are components of the core 11), and Taf7l (12, 13). For example, mice bearing mutant or promoter complex required for gene/tissue-specific transcription deficient CREM showed decreased postmeiotic gene expression of protein-coding genes by RNA polymerase II. Previous studies and defective spermiogenesis (14). Mice deficient in Taf4b, reported that Taf7l knockout (KO) mice exhibit structurally abnor- a testis-specific homolog of Taf4, are initially normal but undergo mal sperm, reduced sperm count, weakened motility, and compro- progressive germ-cell loss and become infertile by 3 mo of age −/Y mised fertility. -

Genetic Signatures of High-Altitude Adaptation and Geographic
www.nature.com/scientificreports OPEN Genetic signatures of high‑altitude adaptation and geographic distribution in Tibetan sheep Jianbin Liu1,2*, Chao Yuan1,2, Tingting Guo1,2, Fan Wang3, Yufeng Zeng1, Xuezhi Ding1, Zengkui Lu1,2, Dingkao Renqing4, Hao Zhang5, Xilan Xu6, Yaojing Yue1,2, Xiaoping Sun1,2, Chune Niu1,2, Deqing Zhuoga7* & Bohui Yang1,2* Most sheep breeding programs designed for the tropics and sub‑tropics have to take into account the impacts of environmental adaptive traits. However, the genetic mechanism regulating the multiple biological processes driving adaptive responses remains unclear. In this study, we applied a selective sweep analysis by combing 1% top values of Fst and ZHp on both altitude and geographic subpopulations (APS) in 636 indigenous Tibetan sheep breeds. Results show that 37 genes were identifed within overlapped genomic regions regarding Fst signifcantly associated with APS. Out of the 37 genes, we found that 8, 3 and 6 genes at chromosomes (chr.) 13, 23 and 27, respectively, were identifed in the genomic regions with 1% top values of ZHp. We further analyzed the INDEL variation of 6 genes at chr.27 (X chromosome) in APS together with corresponding orthologs of 6 genes in Capra, Pantholops, and Bos Taurus. We found that an INDEL was located within 5′UTR region of HAG1 gene. This INDEL of HAG1 was strongly associated with the variation of APS, which was further confrmed by qPCR. Sheep breeds carrying “C‑INDEL” of HAG1 have signifcantly greater body weight, shear amount, corpuscular hemoglobin and globulin levels, but lower body height, than those carrying “CA‑INDEL” of HAG1. -

A Familial Study of Azoospermic Men Identifies Three Novel Causative Mutations in Three New Human Azoospermia Genes
ORIGINAL RESEARCH ARTICLE © American College of Medical Genetics and Genomics A familial study of azoospermic men identifies three novel causative mutations in three new human azoospermia genes Moran Gershoni, PhD1, Ron Hauser, MD2, Leah Yogev, PhD2, Ofer Lehavi, MD2, Foad Azem, MD2, Haim Yavetz, MD2, Shmuel Pietrokovski, PhD1 and Sandra E. Kleiman PhD2 Purpose: Up to 1% of all men experience azoospermia, a condition of Results: We identified three novel likely causative mutations of azo- complete absence of sperm in the semen. The mechanisms and genes ospermia in three genes: MEIOB, TEX14, and DNAH6. These genes involved in spermatogenesis are mainly studied in model organisms, are associated with different meiotic processes: meiotic crossovers, and their relevance to humans is unclear because human genetic daughter cell abscission, and possibly rapid prophase movements. studies are very scarce. Our objective was to uncover novel human Conclusion: The genes and pathways we identified are fundamental mutations and genes causing azoospermia due to testicular meiotic for delineating common causes of azoospermia originating in muta- maturation arrest. tions affecting diverse meiotic processes and have great potential for Methods: Affected and unaffected siblings from three families were accelerating approaches to diagnose, treat, and prevent infertility. subjected to whole-exome or whole-genome sequencing, followed by comprehensive bioinformatics analyses to identify mutations suspected Genet Med advance online publication 16 February 2017 to cause azoospermia. These likely mutations were further screened in azoospermic and normozoospermic men and in men proven to be fer- Key Words: azoospermia; crossover; meiosis; rapid prophase tile, as well as in a reference database of local populations. -

Genetics of Azoospermia
International Journal of Molecular Sciences Review Genetics of Azoospermia Francesca Cioppi , Viktoria Rosta and Csilla Krausz * Department of Biochemical, Experimental and Clinical Sciences “Mario Serio”, University of Florence, 50139 Florence, Italy; francesca.cioppi@unifi.it (F.C.); viktoria.rosta@unifi.it (V.R.) * Correspondence: csilla.krausz@unifi.it Abstract: Azoospermia affects 1% of men, and it can be due to: (i) hypothalamic-pituitary dysfunction, (ii) primary quantitative spermatogenic disturbances, (iii) urogenital duct obstruction. Known genetic factors contribute to all these categories, and genetic testing is part of the routine diagnostic workup of azoospermic men. The diagnostic yield of genetic tests in azoospermia is different in the different etiological categories, with the highest in Congenital Bilateral Absence of Vas Deferens (90%) and the lowest in Non-Obstructive Azoospermia (NOA) due to primary testicular failure (~30%). Whole- Exome Sequencing allowed the discovery of an increasing number of monogenic defects of NOA with a current list of 38 candidate genes. These genes are of potential clinical relevance for future gene panel-based screening. We classified these genes according to the associated-testicular histology underlying the NOA phenotype. The validation and the discovery of novel NOA genes will radically improve patient management. Interestingly, approximately 37% of candidate genes are shared in human male and female gonadal failure, implying that genetic counselling should be extended also to female family members of NOA patients. Keywords: azoospermia; infertility; genetics; exome; NGS; NOA; Klinefelter syndrome; Y chromosome microdeletions; CBAVD; congenital hypogonadotropic hypogonadism Citation: Cioppi, F.; Rosta, V.; Krausz, C. Genetics of Azoospermia. 1. Introduction Int. J. Mol. Sci. -

Targeted Next-Generation Sequencing Identifies Novel Sequence Variations of Genes Associated with Nonobstructive Azoospermia In
CLINICAL RESEARCH e-ISSN 1643-3750 © Med Sci Monit, 2019; 25: 5801-5812 DOI: 10.12659/MSM.915375 Received: 2019.01.26 Accepted: 2019.04.09 Targeted Next-Generation Sequencing Identifies Published: 2019.08.04 Novel Sequence Variations of Genes Associated with Nonobstructive Azoospermia in the Han Population of Northeast China Authors’ Contribution: BCE 1 Xiangyin Liu 1 Center for Reproductive Medicine and Center of Prenatal Diagnosis, First Hospital, Study Design A BDF 1 Qi Xi Jilin University, Changchun, P.R. China Data Collection B 2 Changchun Jida Middle School Experimental School, Changchun, Jilin, P.R. China Statistical Analysis C BF 1 Leilei Li Data Interpretation D CF 2 Qiyuan Wang Manuscript Preparation E BD 1 Yuting Jiang Literature Search F Funds Collection G EF 1 Hongguo Zhang CFG 1 Ruizhi Liu AG 1 Ruixue Wang Corresponding Author: Ruixue Wang, e-mail: [email protected] Source of support: Funding was provided by the Science and Technology Department of Jilin Province (20160101048JC) Background: This study aimed to screen common and low-frequency variants of nonobstructive azoospermia (NOA)-associated genes, and to construct a database for NOA-associated single nucleotide variants (SNVs). Material/Methods: Next-generation sequencing of 466 NOA-associated genes was performed in 34 patients with NOA (mean age, 29.06±4.49 years) and 40 sperm donors (mean age, 25.08±5.75 years) from the Han population of northeast China. The SNV database was constructed by summarizing NOA non-negatively-associated SNVs showing sta- tistical differences between NOA cases and controls, and then selecting low-frequency variants using Baylor’s pipeline, to identify statistically valid SNVs. -

Novel Targets of Apparently Idiopathic Male Infertility
International Journal of Molecular Sciences Review Molecular Biology of Spermatogenesis: Novel Targets of Apparently Idiopathic Male Infertility Rossella Cannarella * , Rosita A. Condorelli , Laura M. Mongioì, Sandro La Vignera * and Aldo E. Calogero Department of Clinical and Experimental Medicine, University of Catania, 95123 Catania, Italy; [email protected] (R.A.C.); [email protected] (L.M.M.); [email protected] (A.E.C.) * Correspondence: [email protected] (R.C.); [email protected] (S.L.V.) Received: 8 February 2020; Accepted: 2 March 2020; Published: 3 March 2020 Abstract: Male infertility affects half of infertile couples and, currently, a relevant percentage of cases of male infertility is considered as idiopathic. Although the male contribution to human fertilization has traditionally been restricted to sperm DNA, current evidence suggest that a relevant number of sperm transcripts and proteins are involved in acrosome reactions, sperm-oocyte fusion and, once released into the oocyte, embryo growth and development. The aim of this review is to provide updated and comprehensive insight into the molecular biology of spermatogenesis, including evidence on spermatogenetic failure and underlining the role of the sperm-carried molecular factors involved in oocyte fertilization and embryo growth. This represents the first step in the identification of new possible diagnostic and, possibly, therapeutic markers in the field of apparently idiopathic male infertility. Keywords: spermatogenetic failure; embryo growth; male infertility; spermatogenesis; recurrent pregnancy loss; sperm proteome; DNA fragmentation; sperm transcriptome 1. Introduction Infertility is a widespread condition in industrialized countries, affecting up to 15% of couples of childbearing age [1]. It is defined as the inability to achieve conception after 1–2 years of unprotected sexual intercourse [2]. -

Taf4b Regulates Oocyte-Specific Genes Essential for Meiosis
UCSF UC San Francisco Previously Published Works Title TAF4b Regulates Oocyte-Specific Genes Essential for Meiosis. Permalink https://escholarship.org/uc/item/5x40p2vg Journal PLoS genetics, 12(6) ISSN 1553-7390 Authors Grive, Kathryn J Gustafson, Eric A Seymour, Kimberly A et al. Publication Date 2016-06-24 DOI 10.1371/journal.pgen.1006128 Peer reviewed eScholarship.org Powered by the California Digital Library University of California RESEARCH ARTICLE TAF4b Regulates Oocyte-Specific Genes Essential for Meiosis Kathryn J. Grive1, Eric A. Gustafson2, Kimberly A. Seymour2, Melody Baddoo3, Christoph Schorl2, Kayla Golnoski4, Aleksandar Rajkovic4, Alexander S. Brodsky5, Richard N. Freiman2* 1 MCB Graduate Program, Brown University, Providence, Rhode Island, United States of America, 2 MCB Department, Brown University, Providence, Rhode Island, United States of America, 3 School of Medicine, Tulane University, New Orleans, Louisiana, United States of America, 4 Magee Women’s Research Institute, Pittsburgh, Pennsylvania, United States of America, 5 Department of Pathology, Rhode Island Hospital and Brown University, Providence, Rhode Island, United States of America a11111 * [email protected] Abstract TAF4b is a gonadal-enriched subunit of the general transcription factor TFIID that is impli- OPEN ACCESS cated in promoting healthy ovarian aging and female fertility in mice and humans. To further Citation: Grive KJ, Gustafson EA, Seymour KA, explore the potential mechanism of TAF4b in promoting ovarian follicle development, we Baddoo M, Schorl C, Golnoski K, et al. (2016) TAF4b Regulates Oocyte-Specific Genes Essential for analyzed global gene expression at multiple time points in the human fetal ovary. This Meiosis. PLoS Genet 12(6): e1006128. doi:10.1371/ computational analysis revealed coordinate expression of human TAF4B and critical regula- journal.pgen.1006128 tors and effectors of meiosis I including SYCP3, YBX2, STAG3, and DAZL. -
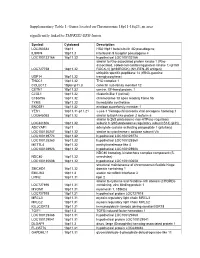
Supplementary Table 1: Genes Located on Chromosome 18P11-18Q23, an Area Significantly Linked to TMPRSS2-ERG Fusion
Supplementary Table 1: Genes located on Chromosome 18p11-18q23, an area significantly linked to TMPRSS2-ERG fusion Symbol Cytoband Description LOC260334 18p11 HSA18p11 beta-tubulin 4Q pseudogene IL9RP4 18p11.3 interleukin 9 receptor pseudogene 4 LOC100132166 18p11.32 hypothetical LOC100132166 similar to Rho-associated protein kinase 1 (Rho- associated, coiled-coil-containing protein kinase 1) (p160 LOC727758 18p11.32 ROCK-1) (p160ROCK) (NY-REN-35 antigen) ubiquitin specific peptidase 14 (tRNA-guanine USP14 18p11.32 transglycosylase) THOC1 18p11.32 THO complex 1 COLEC12 18pter-p11.3 collectin sub-family member 12 CETN1 18p11.32 centrin, EF-hand protein, 1 CLUL1 18p11.32 clusterin-like 1 (retinal) C18orf56 18p11.32 chromosome 18 open reading frame 56 TYMS 18p11.32 thymidylate synthetase ENOSF1 18p11.32 enolase superfamily member 1 YES1 18p11.31-p11.21 v-yes-1 Yamaguchi sarcoma viral oncogene homolog 1 LOC645053 18p11.32 similar to BolA-like protein 2 isoform a similar to 26S proteasome non-ATPase regulatory LOC441806 18p11.32 subunit 8 (26S proteasome regulatory subunit S14) (p31) ADCYAP1 18p11 adenylate cyclase activating polypeptide 1 (pituitary) LOC100130247 18p11.32 similar to cytochrome c oxidase subunit VIc LOC100129774 18p11.32 hypothetical LOC100129774 LOC100128360 18p11.32 hypothetical LOC100128360 METTL4 18p11.32 methyltransferase like 4 LOC100128926 18p11.32 hypothetical LOC100128926 NDC80 homolog, kinetochore complex component (S. NDC80 18p11.32 cerevisiae) LOC100130608 18p11.32 hypothetical LOC100130608 structural maintenance -

Integrative Framework for Identification of Key Cell Identity Genes Uncovers
Integrative framework for identification of key cell PNAS PLUS identity genes uncovers determinants of ES cell identity and homeostasis Senthilkumar Cinghua,1, Sailu Yellaboinaa,b,c,1, Johannes M. Freudenberga,b, Swati Ghosha, Xiaofeng Zhengd, Andrew J. Oldfielda, Brad L. Lackfordd, Dmitri V. Zaykinb, Guang Hud,2, and Raja Jothia,b,2 aSystems Biology Section and dStem Cell Biology Section, Laboratory of Molecular Carcinogenesis, and bBiostatistics Branch, National Institute of Environmental Health Sciences, National Institutes of Health, Research Triangle Park, NC 27709; and cCR Rao Advanced Institute of Mathematics, Statistics, and Computer Science, Hyderabad, Andhra Pradesh 500 046, India Edited by Norbert Perrimon, Harvard Medical School and Howard Hughes Medical Institute, Boston, MA, and approved March 17, 2014 (received for review October 2, 2013) Identification of genes associated with specific biological pheno- (mESCs) for genes essential for the maintenance of ESC identity types is a fundamental step toward understanding the molecular resulted in only ∼8% overlap (8, 9), although many of the unique basis underlying development and pathogenesis. Although RNAi- hits in each screen were known or later validated to be real. The based high-throughput screens are routinely used for this task, lack of concordance suggest that these screens have not reached false discovery and sensitivity remain a challenge. Here we describe saturation (14) and that additional genes of importance remain a computational framework for systematic integration of published to be discovered. gene expression data to identify genes defining a phenotype of Motivated by the need for an alternative approach for iden- interest. We applied our approach to rank-order all genes based on tification of key cell identity genes, we developed a computa- their likelihood of determining ES cell (ESC) identity. -

Taf4b Transcription Networks Regulating Early Oocyte Differentiation
bioRxiv preprint doi: https://doi.org/10.1101/2021.07.18.452838; this version posted July 18, 2021. The copyright holder for this preprint (which was not certified by peer review) is the author/funder. All rights reserved. No reuse allowed without permission. TAF4b transcription networks regulating early oocyte differentiation Megan A. Gura1, Sona Relovska2, Kimberly M. Abt1, Kimberly A. Seymour2, Tong Wu3, Haskan Kaya4, James M. A. Turner4, Thomas G. Fazzio3 and Richard N. Freiman1,2,5 1MCB Graduate Program and 2Department of Molecular Biology, Cell Biology, and Biochemistry, Brown University, 70 Ship St., Box G-E4, Providence, RI 02903, USA 3Department of Molecular, Cell, and Cancer Biology, University of Massachusetts Medical School, Worcester, MA 01605, USA 4The Francis Crick Institute, London, UK 5Corresponding author EMAIL: [email protected] Phone: (401)-863-9633 FAX: (401) 863-9653 Running Title: TAF4b transcription in oocyte differentiation Keywords: Transcription, oogenesis, oocytes, meiosis I, TFIID, TAF4b, POI, fetal oocyte attrition 1 bioRxiv preprint doi: https://doi.org/10.1101/2021.07.18.452838; this version posted July 18, 2021. The copyright holder for this preprint (which was not certified by peer review) is the author/funder. All rights reserved. No reuse allowed without permission. ABSTRACT Establishment of a healthy ovarian reserve is contingent upon numerous regulatory pathways during embryogenesis. Previously, mice lacking TBP-associated factor 4b (Taf4b) were shown to exhibit a diminished ovarian reserve. However, potential oocyte-intrinsic functions of TAF4b have not been examined. Here we use a combination of gene expression profiling and chromatin mapping to characterize the TAF4b gene regulatory network in mouse oocytes. -

Time-Resolved Transcriptome of Barley Anthers and Meiocytes Reveals Robust and Largely Stable Gene Expression Changes at Meiosis
bioRxiv preprint doi: https://doi.org/10.1101/2020.04.20.051425; this version posted April 20, 2020. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY-NC-ND 4.0 International license. 1 LARGE-SCALE BIOLOGY ARTICLE 2 Time-resolved transcriptome of barley anthers and meiocytes reveals 3 robust and largely stable gene expression changes at meiosis entry 4 Abdellah Barakatea,*, Jamie Orra,*, Miriam Schreibera,*, Isabelle Colasa, Dominika 5 Lewandowskaa, Nicola McCalluma, Malcolm Macaulaya, Jenny Morrisa, Mikel Arrietaa, Pete E. 6 Hedleya, Luke Ramsaya and Robbie Waugha,b,c,§ 7 a Cell and Molecular Sciences, The James Hutton Institute, Errol Road, Invergowrie, Dundee, DD2 8 5DA, Scotland, UK. 9 b School of Life Sciences, University of Dundee, Dow Street, Dundee, DD1 5EH, Scotland, UK 10 c School of Agriculture and Wine, University of Adelaide, Plant Genome Building, Waite Campus, 11 Urrbrae, Adelaide, South Australia 12 * These authors contributed equally to this work 13 § Corresponding author: [email protected] 14 Short title: Barley anther transcriptome 15 One sentence summary: Analysis of RNA-seq data from meiotically staged barley anthers and 16 meiocytes highlights the role of lncRNAs within a complex network of transcriptional and post- 17 transcriptional regulation accompanied by a hiatus in differential gene expression during prophase I. 18 The author responsible for distribution of materials integral to the findings presented in this article in 19 accordance with the policy described in the Instructions for Authors (www.plantcell.org) is: Robbie 20 Waugh ([email protected]) 21 22 ABSTRACT 23 In flowering plants, successful germinal cell development and meiotic recombination depend upon a 24 combination of environmental and genetic factors. -

Germline Deletion of Cdyl Causes Teratozoospermia and Progressive
Xia et al. Cell Death and Disease (2019) 10:229 https://doi.org/10.1038/s41419-019-1455-y Cell Death & Disease ARTICLE Open Access Germline deletion of Cdyl causes teratozoospermia and progressive infertility in male mice Xiaoyu Xia1, Xiaowei Zhou2,YanmeiQuan1,YanqinHu1, Fengying Xing3,ZhengzhengLi1,BufangXu2,ChenXu1 and Aijun Zhang1,2 Abstract Chromodomain Y (CDY) is one of the candidate genes for male dyszoospermia related to Y chromosome microdeletion (YCM). However, the function of CDY in regulating spermatogenesis has not been completely determined. The mouse Cdyl (CDY-like) gene is the homolog of human CDY. In the present study, we generated a germline conditional knockout (cKO) model of mouse Cdyl. Significantly, the CdylcKO male mice suffered from the defects in spermatogonia maintenance and spermatozoon morphogenesis, demonstrating teratozoospermia and a progressive infertility phenotype in early adulthood. Importantly, patterns of specific histone methylation and acetylation were extensively changed, which disturbed the transcriptome in CdylcKO testis. Our findings indicated that Cdyl is crucial for spermatogenesis and male fertility, which provides novel insights into the function of CDY gene, as well as the pathogenesis of YCM-related reproductive failure. 1234567890():,; 1234567890():,; 1234567890():,; 1234567890():,; Introduction transmission to the offspring. To date, genes including Since the turn of the millennium, the global incidence DAZ (deleted in azoospermia)2, RBMY (RNA binding rate of infertility has been rising continuously. Among all motif protein, Y-linked)3, USP9Y (ubiquitin specific pep- infertility cases, ~50% of them can be attributed to male tidase 9, Y-linked)4, TSPY (testis-specific protein, Y- factors, such as idiopathic oligozoospermia or azoos- linked)5, and CDY (chromodomain Y)6 have been identi- permia.