Supplementary Table 2
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

The Global Architecture Shaping the Heterogeneity and Tissue-Dependency of the MHC Class I Immunopeptidome Is Evolutionarily Conserved
bioRxiv preprint doi: https://doi.org/10.1101/2020.09.28.317750; this version posted September 29, 2020. The copyright holder for this preprint (which was not certified by peer review) is the author/funder. All rights reserved. No reuse allowed without permission. The Global Architecture Shaping the Heterogeneity and Tissue-Dependency of the MHC Class I Immunopeptidome is Evolutionarily Conserved Authors Peter Kubiniok†1, Ana Marcu†2,3, Leon Bichmann†2,4, Leon Kuchenbecker4, Heiko Schuster1,5, David Hamelin1, Jérome Despault1, Kevin Kovalchik1, Laura Wessling1, Oliver Kohlbacher4,7,8,9,10 Stefan Stevanovic2,3,6, Hans-Georg Rammensee2,3,6, Marian C. Neidert11, Isabelle Sirois1, Etienne Caron1,12* Affiliations *Corresponding and Leading author: Etienne Caron ([email protected]) †Equal contribution to this work 1CHU Sainte-Justine Research Center, Montreal, QC H3T 1C5, Canada 2Department of Immunology, Interfaculty Institute for Cell Biology, University of Tübingen, Tübingen, Baden-Württemberg, 72076, Germany. 3Cluster of Excellence iFIT (EXC 2180) "Image-Guided and Functionally Instructed Tumor Therapies", University of Tübingen, Tübingen, Baden-Württemberg, 72076, Germany. 4Applied Bioinformatics, Dept. of Computer Science, University of Tübingen, Tübingen, Baden- Württemberg, 72074, Germany. 5Immatics Biotechnologies GmbH, Tübingen, 72076, Baden-Württemberg, Germany. 6DKFZ Partner Site Tübingen, German Cancer Consortium (DKTK), Tübingen, Baden- Württemberg, 72076, Germany. 7Institute for Bioinformatics and Medical Informatics, -

A Computational Approach for Defining a Signature of Β-Cell Golgi Stress in Diabetes Mellitus
Page 1 of 781 Diabetes A Computational Approach for Defining a Signature of β-Cell Golgi Stress in Diabetes Mellitus Robert N. Bone1,6,7, Olufunmilola Oyebamiji2, Sayali Talware2, Sharmila Selvaraj2, Preethi Krishnan3,6, Farooq Syed1,6,7, Huanmei Wu2, Carmella Evans-Molina 1,3,4,5,6,7,8* Departments of 1Pediatrics, 3Medicine, 4Anatomy, Cell Biology & Physiology, 5Biochemistry & Molecular Biology, the 6Center for Diabetes & Metabolic Diseases, and the 7Herman B. Wells Center for Pediatric Research, Indiana University School of Medicine, Indianapolis, IN 46202; 2Department of BioHealth Informatics, Indiana University-Purdue University Indianapolis, Indianapolis, IN, 46202; 8Roudebush VA Medical Center, Indianapolis, IN 46202. *Corresponding Author(s): Carmella Evans-Molina, MD, PhD ([email protected]) Indiana University School of Medicine, 635 Barnhill Drive, MS 2031A, Indianapolis, IN 46202, Telephone: (317) 274-4145, Fax (317) 274-4107 Running Title: Golgi Stress Response in Diabetes Word Count: 4358 Number of Figures: 6 Keywords: Golgi apparatus stress, Islets, β cell, Type 1 diabetes, Type 2 diabetes 1 Diabetes Publish Ahead of Print, published online August 20, 2020 Diabetes Page 2 of 781 ABSTRACT The Golgi apparatus (GA) is an important site of insulin processing and granule maturation, but whether GA organelle dysfunction and GA stress are present in the diabetic β-cell has not been tested. We utilized an informatics-based approach to develop a transcriptional signature of β-cell GA stress using existing RNA sequencing and microarray datasets generated using human islets from donors with diabetes and islets where type 1(T1D) and type 2 diabetes (T2D) had been modeled ex vivo. To narrow our results to GA-specific genes, we applied a filter set of 1,030 genes accepted as GA associated. -

To Study Mutant P53 Gain of Function, Various Tumor-Derived P53 Mutants
Differential effects of mutant TAp63γ on transactivation of p53 and/or p63 responsive genes and their effects on global gene expression. A thesis submitted in partial fulfillment of the requirements for the degree of Master of Science By Shama K Khokhar M.Sc., Bilaspur University, 2004 B.Sc., Bhopal University, 2002 2007 1 COPYRIGHT SHAMA K KHOKHAR 2007 2 WRIGHT STATE UNIVERSITY SCHOOL OF GRADUATE STUDIES Date of Defense: 12-03-07 I HEREBY RECOMMEND THAT THE THESIS PREPARED UNDER MY SUPERVISION BY SHAMA KHAN KHOKHAR ENTITLED Differential effects of mutant TAp63γ on transactivation of p53 and/or p63 responsive genes and their effects on global gene expression BE ACCEPTED IN PARTIAL FULFILLMENT OF THE REQUIREMENTS FOR THE DEGREE OF Master of Science Madhavi P. Kadakia, Ph.D. Thesis Director Daniel Organisciak , Ph.D. Department Chair Committee on Final Examination Madhavi P. Kadakia, Ph.D. Steven J. Berberich, Ph.D. Michael Leffak, Ph.D. Joseph F. Thomas, Jr., Ph.D. Dean, School of Graduate Studies 3 Abstract Khokhar, Shama K. M.S., Department of Biochemistry and Molecular Biology, Wright State University, 2007 Differential effect of TAp63γ mutants on transactivation of p53 and/or p63 responsive genes and their effects on global gene expression. p63, a member of the p53 gene family, known to play a role in development, has more recently also been implicated in cancer progression. Mice lacking p63 exhibit severe developmental defects such as limb truncations, abnormal skin, and absence of hair follicles, teeth, and mammary glands. Germline missense mutations of p63 have been shown to be responsible for several human developmental syndromes including SHFM, EEC and ADULT syndromes and are associated with anomalies in the development of organs of epithelial origin. -

UNIVERSITY of CALIFORNIA, IRVINE Combinatorial Regulation By
UNIVERSITY OF CALIFORNIA, IRVINE Combinatorial regulation by maternal transcription factors during activation of the endoderm gene regulatory network DISSERTATION submitted in partial satisfaction of the requirements for the degree of DOCTOR OF PHILOSOPHY in Biological Sciences by Kitt D. Paraiso Dissertation Committee: Professor Ken W.Y. Cho, Chair Associate Professor Olivier Cinquin Professor Thomas Schilling 2018 Chapter 4 © 2017 Elsevier Ltd. © 2018 Kitt D. Paraiso DEDICATION To the incredibly intelligent and talented people, who in one way or another, helped complete this thesis. ii TABLE OF CONTENTS Page LIST OF FIGURES vii LIST OF TABLES ix LIST OF ABBREVIATIONS X ACKNOWLEDGEMENTS xi CURRICULUM VITAE xii ABSTRACT OF THE DISSERTATION xiv CHAPTER 1: Maternal transcription factors during early endoderm formation in 1 Xenopus Transcription factors co-regulate in a cell type-specific manner 2 Otx1 is expressed in a variety of cell lineages 4 Maternal otx1 in the endodermal conteXt 5 Establishment of enhancers by maternal transcription factors 9 Uncovering the endodermal gene regulatory network 12 Zygotic genome activation and temporal control of gene eXpression 14 The role of maternal transcription factors in early development 18 References 19 CHAPTER 2: Assembly of maternal transcription factors initiates the emergence 26 of tissue-specific zygotic cis-regulatory regions Introduction 28 Identification of maternal vegetally-localized transcription factors 31 Vegt and OtX1 combinatorially regulate the endodermal 33 transcriptome iii -

Supplemental Materials Supplemental Table 1
Electronic Supplementary Material (ESI) for RSC Advances. This journal is © The Royal Society of Chemistry 2016 Supplemental Materials Supplemental Table 1. The differentially expressed proteins from rat pancreas identified by proteomics (SAP vs. SO) No. Protein name Gene name ratio P value 1 Metallothionein Mt1m 3.35 6.34E-07 2 Neutrophil antibiotic peptide NP-2 Defa 3.3 8.39E-07 3 Ilf2 protein Ilf2 3.18 1.75E-06 4 Numb isoform o/o rCG 3.12 2.73E-06 5 Lysozyme Lyz2 3.01 5.63E-06 6 Glucagon Gcg 2.89 1.17E-05 7 Serine protease HTRA1 Htra1 2.75 2.97E-05 8 Alpha 2 macroglobulin cardiac isoform (Fragment) 2.75 2.97E-05 9 Myosin IF (Predicted) Myo1f 2.65 5.53E-05 10 Neuroendocrine secretory protein 55 Gnas 2.61 7.60E-05 11 Matrix metallopeptidase 8 Mmp8 2.57 9.47E-05 12 Protein Tnks1bp1 Tnks1bp1 2.53 1.22E-04 13 Alpha-parvin Parva 2.47 1.78E-04 14 C4b-binding protein alpha chain C4bpa 2.42 2.53E-04 15 Protein KTI12 homolog Kti12 2.41 2.74E-04 16 Protein Rab11fip5 Rab11fip5 2.41 2.84E-04 17 Protein Mcpt1l3 Mcpt1l3 2.33 4.43E-04 18 Phospholipase B-like 1 Plbd1 2.33 4.76E-04 Aldehyde dehydrogenase (NAD), cytosolic 19 2.32 4.93E-04 (Fragments) 20 Protein Dpy19l2 Dpy19l2 2.3 5.68E-04 21 Regenerating islet-derived 3 alpha, isoform CRA_a Reg3a 2.27 6.74E-04 22 60S acidic ribosomal protein P1 Rplp1 2.26 7.22E-04 23 Serum albumin Alb 2.25 7.98E-04 24 Ribonuclease 4 Rnase4 2.24 8.25E-04 25 Cct-5 protein (Fragment) Cct5 2.24 8.52E-04 26 Protein S100-A9 S100a9 2.22 9.71E-04 27 Creatine kinase M-type Ckm 2.21 1.00E-03 28 Protein Larp4b Larp4b 2.18 1.25E-03 -

Leishmania (L.) Amazonensis Peptidase Activities Inside the Living Cells and in Their Lysates
Molecular & Biochemical Parasitology 184 (2012) 82–89 Contents lists available at SciVerse ScienceDirect Molecular & Biochemical Parasitology Leishmania (L.) amazonensis peptidase activities inside the living cells and in their lysates a a b c a Elide E. Caroselli , Diego M. Assis , Clara L. Barbiéri , Wagner A.S. Júdice , Maria A. Juliano , d a,∗ Marcos L. Gazarini , Luiz Juliano a Department of Biophysics, Escola Paulista de Medicina, Universidade Federal de São Paulo, SP, Brazil b Department of Microbiology, Immunology and Parasitology, Escola Paulista de Medicina, Universidade Federal de São Paulo, SP, Brazil c Centro Interdisciplinar de Investigac¸ ão Bioquímica, Universidade de Mogi das Cruzes, Av. Dr. Cândido Xavier de Almeida Souza 200, 08780-911 Mogi das Cruzes, Brazil d Department of Biosciences, Universidade Federal de São Paulo, Santos, Brazil a r t i c l e i n f o a b s t r a c t Article history: In this study we investigated the peptidase activity in Leishmania (L.) amazonensis live amastigote by con- Received 24 November 2011 focal microscopy using peptidyl-MCA as substrates, the hydrolysis of which releases the MCA fluorophore Received in revised form 13 March 2012 inside the cells. Cell pre-treatment with peptidase inhibitors indicated the presence of cysteine and ser- Accepted 27 April 2012 ine peptidases. It was noteworthy that Leishmania amastigotes incorporate only substrates (Z-FR-MCA, Available online 6 May 2012 Z-RR-MCA) or inhibitors (E64, TLCK) containing positively charged groups. The peptidase activities in the supernatants of amastigotes and promastigotes lysates were also evaluated with the same peptidyl-MCA Keywords: substrates and inhibitors in the pH range 4.5–9.0. -

Supplementary Table 9. Functional Annotation Clustering Results for the Union (GS3) of the Top Genes from the SNP-Level and Gene-Based Analyses (See ST4)
Supplementary Table 9. Functional Annotation Clustering Results for the union (GS3) of the top genes from the SNP-level and Gene-based analyses (see ST4) Column Header Key Annotation Cluster Name of cluster, sorted by descending Enrichment score Enrichment Score EASE enrichment score for functional annotation cluster Category Pathway Database Term Pathway name/Identifier Count Number of genes in the submitted list in the specified term % Percentage of identified genes in the submitted list associated with the specified term PValue Significance level associated with the EASE enrichment score for the term Genes List of genes present in the term List Total Number of genes from the submitted list present in the category Pop Hits Number of genes involved in the specified term (category-specific) Pop Total Number of genes in the human genome background (category-specific) Fold Enrichment Ratio of the proportion of count to list total and population hits to population total Bonferroni Bonferroni adjustment of p-value Benjamini Benjamini adjustment of p-value FDR False Discovery Rate of p-value (percent form) Annotation Cluster 1 Enrichment Score: 3.8978262119731335 Category Term Count % PValue Genes List Total Pop Hits Pop Total Fold Enrichment Bonferroni Benjamini FDR GOTERM_CC_DIRECT GO:0005886~plasma membrane 383 24.33290978 5.74E-05 SLC9A9, XRCC5, HRAS, CHMP3, ATP1B2, EFNA1, OSMR, SLC9A3, EFNA3, UTRN, SYT6, ZNRF2, APP, AT1425 4121 18224 1.18857065 0.038655922 0.038655922 0.086284383 UP_KEYWORDS Membrane 626 39.77128335 1.53E-04 SLC9A9, HRAS, -

The Microbiota-Produced N-Formyl Peptide Fmlf Promotes Obesity-Induced Glucose
Page 1 of 230 Diabetes Title: The microbiota-produced N-formyl peptide fMLF promotes obesity-induced glucose intolerance Joshua Wollam1, Matthew Riopel1, Yong-Jiang Xu1,2, Andrew M. F. Johnson1, Jachelle M. Ofrecio1, Wei Ying1, Dalila El Ouarrat1, Luisa S. Chan3, Andrew W. Han3, Nadir A. Mahmood3, Caitlin N. Ryan3, Yun Sok Lee1, Jeramie D. Watrous1,2, Mahendra D. Chordia4, Dongfeng Pan4, Mohit Jain1,2, Jerrold M. Olefsky1 * Affiliations: 1 Division of Endocrinology & Metabolism, Department of Medicine, University of California, San Diego, La Jolla, California, USA. 2 Department of Pharmacology, University of California, San Diego, La Jolla, California, USA. 3 Second Genome, Inc., South San Francisco, California, USA. 4 Department of Radiology and Medical Imaging, University of Virginia, Charlottesville, VA, USA. * Correspondence to: 858-534-2230, [email protected] Word Count: 4749 Figures: 6 Supplemental Figures: 11 Supplemental Tables: 5 1 Diabetes Publish Ahead of Print, published online April 22, 2019 Diabetes Page 2 of 230 ABSTRACT The composition of the gastrointestinal (GI) microbiota and associated metabolites changes dramatically with diet and the development of obesity. Although many correlations have been described, specific mechanistic links between these changes and glucose homeostasis remain to be defined. Here we show that blood and intestinal levels of the microbiota-produced N-formyl peptide, formyl-methionyl-leucyl-phenylalanine (fMLF), are elevated in high fat diet (HFD)- induced obese mice. Genetic or pharmacological inhibition of the N-formyl peptide receptor Fpr1 leads to increased insulin levels and improved glucose tolerance, dependent upon glucagon- like peptide-1 (GLP-1). Obese Fpr1-knockout (Fpr1-KO) mice also display an altered microbiome, exemplifying the dynamic relationship between host metabolism and microbiota. -

Studies of Structure and Function of Tripeptidyl-Peptidase II
Till familj och vänner List of Papers This thesis is based on the following papers, which are referred to in the text by their Roman numerals. I. Eriksson, S.; Gutiérrez, O.A.; Bjerling, P.; Tomkinson, B. (2009) De- velopment, evaluation and application of tripeptidyl-peptidase II se- quence signatures. Archives of Biochemistry and Biophysics, 484(1):39-45 II. Lindås, A-C.; Eriksson, S.; Josza, E.; Tomkinson, B. (2008) Investiga- tion of a role for Glu-331 and Glu-305 in substrate binding of tripepti- dyl-peptidase II. Biochimica et Biophysica Acta, 1784(12):1899-1907 III. Eklund, S.; Lindås, A-C.; Hamnevik, E.; Widersten, M.; Tomkinson, B. Inter-species variation in the pH dependence of tripeptidyl- peptidase II. Manuscript IV. Eklund, S.; Kalbacher, H.; Tomkinson, B. Characterization of the endopeptidase activity of tripeptidyl-peptidase II. Manuscript Paper I and II were published under maiden name (Eriksson). Reprints were made with permission from the respective publishers. Contents Introduction ..................................................................................................... 9 Enzymes ..................................................................................................... 9 Enzymes and pH dependence .............................................................. 11 Peptidases ................................................................................................. 12 Serine peptidases ................................................................................. 14 Intracellular protein -

Genome-Wide Transcriptome Analysis of Laminar Tissue During the Early Stages of Experimentally Induced Equine Laminitis
GENOME-WIDE TRANSCRIPTOME ANALYSIS OF LAMINAR TISSUE DURING THE EARLY STAGES OF EXPERIMENTALLY INDUCED EQUINE LAMINITIS A Dissertation by JIXIN WANG Submitted to the Office of Graduate Studies of Texas A&M University in partial fulfillment of the requirements for the degree of DOCTOR OF PHILOSOPHY December 2010 Major Subject: Biomedical Sciences GENOME-WIDE TRANSCRIPTOME ANALYSIS OF LAMINAR TISSUE DURING THE EARLY STAGES OF EXPERIMENTALLY INDUCED EQUINE LAMINITIS A Dissertation by JIXIN WANG Submitted to the Office of Graduate Studies of Texas A&M University in partial fulfillment of the requirements for the degree of DOCTOR OF PHILOSOPHY Approved by: Chair of Committee, Bhanu P. Chowdhary Committee Members, Terje Raudsepp Paul B. Samollow Loren C. Skow Penny K. Riggs Head of Department, Evelyn Tiffany-Castiglioni December 2010 Major Subject: Biomedical Sciences iii ABSTRACT Genome-wide Transcriptome Analysis of Laminar Tissue During the Early Stages of Experimentally Induced Equine Laminitis. (December 2010) Jixin Wang, B.S., Tarim University of Agricultural Reclamation; M.S., South China Agricultural University; M.S., Texas A&M University Chair of Advisory Committee: Dr. Bhanu P. Chowdhary Equine laminitis is a debilitating disease that causes extreme sufferring in afflicted horses and often results in a lifetime of chronic pain. The exact sequence of pathophysiological events culminating in laminitis has not yet been characterized, and this is reflected in the lack of any consistently effective therapeutic strategy. For these reasons, we used a newly developed 21,000 element equine-specific whole-genome oligoarray to perform transcriptomic analysis on laminar tissue from horses with experimentally induced models of laminitis: carbohydrate overload (CHO), hyperinsulinaemia (HI), and oligofructose (OF). -
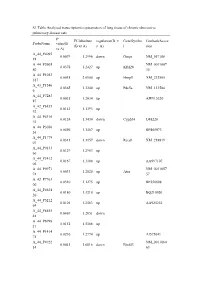
S1 Table Analyzed Transcriptomics Parameters of Lung Tissue of Chronic
S1 Table Analyzed transcriptomics parameters of lung tissue of chronic obstructive pulmonary disease rats p- FCAbsolute regulation(B_v GeneSymbo GenbankAcces ProbeName value(B (B vs A) s_A) l sion vs A) A_44_P4095 0.0079 1.2996 down Gmpr NM_057188 18 A_44_P2605 NM_0011067 0.0378 1.2427 up Klhl28 80 35 A_44_P1052 0.0018 2.0900 up Hnrpll XM_233805 387 A_43_P1546 0.0365 1.3240 up Pde5a NM_133584 6 A_44_P7283 0.0035 1.2634 up AW915320 87 A_42_P8435 0.0143 1.1393 up 92 A_44_P4310 0.0124 1.3434 down Cyp2d4 U48220 92 A_44_P3056 0.0496 1.1467 up BP465973 36 A_44_P1779 0.0311 1.1157 down Rccd1 XM_218819 07 A_44_P9133 0.0129 1.2963 up 60 A_44_P3412 0.0167 1.3108 up AA997107 04 A_44_P9971 NM_0011057 0.0033 1.2820 up Atrx 74 57 A_42_P7763 0.0310 1.1275 up BF556698 00 A_44_P4634 0.0180 1.3210 up BQ210920 20 A_44_P5212 0.0101 1.2583 up AA925252 64 A_44_P8855 0.0469 1.2051 down 48 A_44_P6998 0.0151 1.5168 up 21 A_44_P1414 0.0236 1.2774 up AI575641 75 A_44_P4922 NM_0011064 0.0041 1.6816 down Eps8l3 14 63 A_43_P2202 0.0488 1.2176 up Setx XM_342400 0 A_44_P2066 0.0205 1.2767 up BF417274 31 A_44_P6213 0.0007 2.0237 up 04 A_44_P3775 0.0068 1.2952 up 20 A_44_P6058 0.0365 1.1240 up 38 A_44_P2861 0.0022 1.4896 up XM_226020 58 A_44_P9139 0.0108 1.5661 up 62 A_43_P1214 0.0058 1.9100 up Hnf4a NM_022180 6 A_44_P6525 NM_0011300 0.0036 1.1586 down Kdm1 85 98 A_44_P3541 0.0061 1.3319 down Cep250 XR_009560 50 A_44_P5160 RGD131055 NM_0011068 0.0293 1.2854 down 73 2 38 A_44_P2237 0.0192 1.3087 up AW142694 41 A_43_P1552 0.0096 1.2036 up Ccnd3 NM_012766 4 A_44_P4098 0.0479 -

Handbook of Proteolytic Enzymes Second Edition Volume 1 Aspartic and Metallo Peptidases
Handbook of Proteolytic Enzymes Second Edition Volume 1 Aspartic and Metallo Peptidases Alan J. Barrett Neil D. Rawlings J. Fred Woessner Editor biographies xxi Contributors xxiii Preface xxxi Introduction ' Abbreviations xxxvii ASPARTIC PEPTIDASES Introduction 1 Aspartic peptidases and their clans 3 2 Catalytic pathway of aspartic peptidases 12 Clan AA Family Al 3 Pepsin A 19 4 Pepsin B 28 5 Chymosin 29 6 Cathepsin E 33 7 Gastricsin 38 8 Cathepsin D 43 9 Napsin A 52 10 Renin 54 11 Mouse submandibular renin 62 12 Memapsin 1 64 13 Memapsin 2 66 14 Plasmepsins 70 15 Plasmepsin II 73 16 Tick heme-binding aspartic proteinase 76 17 Phytepsin 77 18 Nepenthesin 85 19 Saccharopepsin 87 20 Neurosporapepsin 90 21 Acrocylindropepsin 9 1 22 Aspergillopepsin I 92 23 Penicillopepsin 99 24 Endothiapepsin 104 25 Rhizopuspepsin 108 26 Mucorpepsin 11 1 27 Polyporopepsin 113 28 Candidapepsin 115 29 Candiparapsin 120 30 Canditropsin 123 31 Syncephapepsin 125 32 Barrierpepsin 126 33 Yapsin 1 128 34 Yapsin 2 132 35 Yapsin A 133 36 Pregnancy-associated glycoproteins 135 37 Pepsin F 137 38 Rhodotorulapepsin 139 39 Cladosporopepsin 140 40 Pycnoporopepsin 141 Family A2 and others 41 Human immunodeficiency virus 1 retropepsin 144 42 Human immunodeficiency virus 2 retropepsin 154 43 Simian immunodeficiency virus retropepsin 158 44 Equine infectious anemia virus retropepsin 160 45 Rous sarcoma virus retropepsin and avian myeloblastosis virus retropepsin 163 46 Human T-cell leukemia virus type I (HTLV-I) retropepsin 166 47 Bovine leukemia virus retropepsin 169 48