PARTE I Herramientas Básicas
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Summary of Offerings in the PBS Bulb Exchange, Dec 2012- Nov 2019
Summary of offerings in the PBS Bulb Exchange, Dec 2012- Nov 2019 3841 Number of items in BX 301 thru BX 463 1815 Number of unique text strings used as taxa 990 Taxa offered as bulbs 1056 Taxa offered as seeds 308 Number of genera This does not include the SXs. Top 20 Most Oft Listed: BULBS Times listed SEEDS Times listed Oxalis obtusa 53 Zephyranthes primulina 20 Oxalis flava 36 Rhodophiala bifida 14 Oxalis hirta 25 Habranthus tubispathus 13 Oxalis bowiei 22 Moraea villosa 13 Ferraria crispa 20 Veltheimia bracteata 13 Oxalis sp. 20 Clivia miniata 12 Oxalis purpurea 18 Zephyranthes drummondii 12 Lachenalia mutabilis 17 Zephyranthes reginae 11 Moraea sp. 17 Amaryllis belladonna 10 Amaryllis belladonna 14 Calochortus venustus 10 Oxalis luteola 14 Zephyranthes fosteri 10 Albuca sp. 13 Calochortus luteus 9 Moraea villosa 13 Crinum bulbispermum 9 Oxalis caprina 13 Habranthus robustus 9 Oxalis imbricata 12 Haemanthus albiflos 9 Oxalis namaquana 12 Nerine bowdenii 9 Oxalis engleriana 11 Cyclamen graecum 8 Oxalis melanosticta 'Ken Aslet'11 Fritillaria affinis 8 Moraea ciliata 10 Habranthus brachyandrus 8 Oxalis commutata 10 Zephyranthes 'Pink Beauty' 8 Summary of offerings in the PBS Bulb Exchange, Dec 2012- Nov 2019 Most taxa specify to species level. 34 taxa were listed as Genus sp. for bulbs 23 taxa were listed as Genus sp. for seeds 141 taxa were listed with quoted 'Variety' Top 20 Most often listed Genera BULBS SEEDS Genus N items BXs Genus N items BXs Oxalis 450 64 Zephyranthes 202 35 Lachenalia 125 47 Calochortus 94 15 Moraea 99 31 Moraea -

GENOME EVOLUTION in MONOCOTS a Dissertation
GENOME EVOLUTION IN MONOCOTS A Dissertation Presented to The Faculty of the Graduate School At the University of Missouri In Partial Fulfillment Of the Requirements for the Degree Doctor of Philosophy By Kate L. Hertweck Dr. J. Chris Pires, Dissertation Advisor JULY 2011 The undersigned, appointed by the dean of the Graduate School, have examined the dissertation entitled GENOME EVOLUTION IN MONOCOTS Presented by Kate L. Hertweck A candidate for the degree of Doctor of Philosophy And hereby certify that, in their opinion, it is worthy of acceptance. Dr. J. Chris Pires Dr. Lori Eggert Dr. Candace Galen Dr. Rose‐Marie Muzika ACKNOWLEDGEMENTS I am indebted to many people for their assistance during the course of my graduate education. I would not have derived such a keen understanding of the learning process without the tutelage of Dr. Sandi Abell. Members of the Pires lab provided prolific support in improving lab techniques, computational analysis, greenhouse maintenance, and writing support. Team Monocot, including Dr. Mike Kinney, Dr. Roxi Steele, and Erica Wheeler were particularly helpful, but other lab members working on Brassicaceae (Dr. Zhiyong Xiong, Dr. Maqsood Rehman, Pat Edger, Tatiana Arias, Dustin Mayfield) all provided vital support as well. I am also grateful for the support of a high school student, Cady Anderson, and an undergraduate, Tori Docktor, for their assistance in laboratory procedures. Many people, scientist and otherwise, helped with field collections: Dr. Travis Columbus, Hester Bell, Doug and Judy McGoon, Julie Ketner, Katy Klymus, and William Alexander. Many thanks to Barb Sonderman for taking care of my greenhouse collection of many odd plants brought back from the field. -

Ornithogalum L
Sistemática del género Ornithogalum L. (Hyacinthaceae) en el Mediterráneo occidental: implicaciones taxonómicas, filogenéticas y biogeográficas Mario Martínez Azorín Departamento de Ciencias Ambientales y Recursos Naturales Sistemática del género Ornithogalum L. (Hyacinthaceae) en el Mediterráneo occidental: implicaciones taxonómicas, filogenéticas y biogeográficas Tesis Doctoral Mario Martínez Azorín Abril 2008 Departamento de Ciencias Ambientales y Recursos Naturales MANUEL B. CRESPO VILLALBA, CATEDRÁTICO DE UNIVERSIDAD, Y ANA JUAN GALLARDO, PROFESORA CONTRATADA DOCTORA, MIEMBROS DEL ÁREA DE BOTÁNICA DEL DEPARTAMENTO DE CIENCIAS AMBIENTALES Y RECURSOS NATURALES, Y DEL I.U. CIBIO, DE LA UNIVERSIDAD DE ALICANTE, C E R T I F I C A N: Que la presente memoria titulada “Sistemática del género Ornithogalum L. (Hyacinthaceae) en el Mediterráneo occidental: implicaciones taxonómicas, filogenéticas y biogeográficas”, que para aspirar al grado de Doctor presenta el Licenciado en Biología D. Mario Martínez Azorín, ha sido realizada bajo nuestra dirección en este Departamento, y considerando que reúne los requisitos necesarios a tal efecto, autorizamos que siga los trámites preceptivos para su exposición y defensa. Y para que así conste donde convenga al interesado, y a petición suya, expedimos el presente certificado en Alicante, a veinticinco de febrero de dos mil ocho. A mis padres, Encarna y Antonio a Adrian y a Almudena Agradecimientos AGRADECIMIENTOS Ante todo, deseo agradecer a todas las personas que con su ayuda, apoyo y comprensión han hecho posible este trabajo. En primer lugar, quiero dar mi más sincero agradecimiento a mis directores de Tesis, los Drs. Manuel B. Crespo Villalba y Ana Juan Gallardo, por haberme animado a emprender el largo camino que culmina con la realización del presente trabajo. -

Plant Geography of Chile PLANT and VEGETATION
Plant Geography of Chile PLANT AND VEGETATION Volume 5 Series Editor: M.J.A. Werger For further volumes: http://www.springer.com/series/7549 Plant Geography of Chile by Andrés Moreira-Muñoz Pontificia Universidad Católica de Chile, Santiago, Chile 123 Dr. Andrés Moreira-Muñoz Pontificia Universidad Católica de Chile Instituto de Geografia Av. Vicuña Mackenna 4860, Santiago Chile [email protected] ISSN 1875-1318 e-ISSN 1875-1326 ISBN 978-90-481-8747-8 e-ISBN 978-90-481-8748-5 DOI 10.1007/978-90-481-8748-5 Springer Dordrecht Heidelberg London New York © Springer Science+Business Media B.V. 2011 No part of this work may be reproduced, stored in a retrieval system, or transmitted in any form or by any means, electronic, mechanical, photocopying, microfilming, recording or otherwise, without written permission from the Publisher, with the exception of any material supplied specifically for the purpose of being entered and executed on a computer system, for exclusive use by the purchaser of the work. ◦ ◦ Cover illustration: High-Andean vegetation at Laguna Miscanti (23 43 S, 67 47 W, 4350 m asl) Printed on acid-free paper Springer is part of Springer Science+Business Media (www.springer.com) Carlos Reiche (1860–1929) In Memoriam Foreword It is not just the brilliant and dramatic scenery that makes Chile such an attractive part of the world. No, that country has so very much more! And certainly it has a rich and beautiful flora. Chile’s plant world is strongly diversified and shows inter- esting geographical and evolutionary patterns. This is due to several factors: The geographical position of the country on the edge of a continental plate and stretch- ing along an extremely long latitudinal gradient from the tropics to the cold, barren rocks of Cape Horn, opposite Antarctica; the strong differences in altitude from sea level to the icy peaks of the Andes; the inclusion of distant islands in the country’s territory; the long geological and evolutionary history of the biota; and the mixture of tropical and temperate floras. -

Bufadienolide Und DNA-Sequenzen
ZOBODAT - www.zobodat.at Zoologisch-Botanische Datenbank/Zoological-Botanical Database Digitale Literatur/Digital Literature Zeitschrift/Journal: Stapfia Jahr/Year: 2001 Band/Volume: 0075 Autor(en)/Author(s): Pfosser Martin, Speta Franz Artikel/Article: Bufadienolide und DNA-Sequenzen: Über Zusammenhalt und Aufteilung der Urgineoideae (Hyacinthaceae) 177-250 © Biologiezentrum Linz/Austria; download unter www.biologiezentrum.at Bufadienolide und DNA-Sequenzen: Über Zusammenhalt und Aufteilung der Urgineoideae (Hyacinthaceae) MARTIN F. PFOSSER & FRANZ SPETA Abstract data even presents enough variation to delimit geographical races. Molecular ana- Bufadienolides and DNA lysis of maternally inherited plastid DNA sequences: on lumping and smashing of subfamily Urgi- genomes suggests several cases of hybri- neoideae {Hyacinthaceae) dizations resulting in allopolyploid com- plexes. For example, the hexaploid species Subfamily Urgineoideae is chemotaxo- Charybdis maritima is polyphyletic and nomically characterised by the presence of represents at least two different relation- bufadienolides, a group of cardiac active ships, one with C. hesperia as the female compounds absent in other subfamilies in parent and another relationship with pro- Hyacinthaceae. However, many genera bably C. numidica as the female parent. possess specific and unique compounds, Sister group to Charybdis is genus Seka- which show enough variation for further nama, which is distributed south of the and more detailed systematic analyses. Sahara. Our data provide evidence -

And Type the TITLE of YOUR WORK in All Caps
PHYLOGENOMIC PLACEMENT OF ANCIENT POLYPLOIDY EVENTS WITHIN THE POALES AND AGAVOIDEAE (ASPARAGALES) by MICHAEL RAMON MCKAIN (Under the Direction of James H. Leebens-Mack) ABSTRACT Polyploidy has been an important component to the evolution of angiosperms. Recent studies have shown that an ancient polyploid (paleopolyploid) event can be traced to the lineage leading to the diversification of all angiosperms, and it has long been known that recurring polyploid events can be found throughout the angiosperm tree of life. With the advent of high- throughput sequencing, the prominent place of paleopolyploid events in the evolutionary history of angiosperms has become increasingly clear. Polyploidy is thought to spur both diversification and trait innovation through the duplication and reworking of gene networks. Understanding the evolutionary impact of paleopolyploidy within the angiosperms requires knowing when these events occurred during angiosperm evolution. This study utilizes a high-throughput phylogenomic approach to identify the timing of paleopolyploid events by comparing the origin of paralogous genes within a gene family to a known species tree. Transcriptome data derived from taxa in lineages with previously little to no genomic data, were utilized to assess the timing of duplication events within hundreds of gene families. Previously described paleopolyploid events in the history of grasses, identified through analyses of syntenic blocks within Poaceae genomes, were placed on the Poales phylogeny and the implications of these events were considered. Additionally, a previously unverified paleopolyploidy event was found to have occurred in a common ancestor of all members of the Asparagales and commelinids (including Poales, Zingiberales, Commelinales, Arecales and Dasypogonales). The phylogeny of the Asparagaceae subfamily Agavoideae was resolved using whole chloroplast genomes, and two previously unknown paleopolyploid events were described within the context of that phylogeny. -

Multilocus Phylogenetic Inference in Subfamily Chlorogaloideae and Related Genera of Agavaceae – Informing Questions in Taxonomy at Multiple Ranks ⇑ Jenny K
Molecular Phylogenetics and Evolution 84 (2015) 266–283 Contents lists available at ScienceDirect Molecular Phylogenetics and Evolution journal homepage: www.elsevier.com/locate/ympev Multilocus phylogenetic inference in subfamily Chlorogaloideae and related genera of Agavaceae – Informing questions in taxonomy at multiple ranks ⇑ Jenny K. Archibald a, , Susan R. Kephart b, Kathryn E. Theiss b, Anna L. Petrosky c, Theresa M. Culley d a Biodiversity Institute and Department of Ecology and Evolutionary Biology, University of Kansas, Lawrence, KS 66045, USA b Department of Biology, Willamette University, Salem, OR 97301, USA c Department of Integrative Biology, University of California, Berkeley, CA 94709, USA d Department of Biological Sciences, University of Cincinnati, Cincinnati, OH 45221, USA article info abstract Article history: A series of taxonomic questions at the subfamilial, generic, and intrageneric levels have remained within Received 21 July 2014 subfamily Chlorogaloideae s.s. (comprising Camassia, Chlorogalum, Hastingsia, and Schoenolirion) and Revised 8 December 2014 relatives in Agavaceae. We present the first phylogenetic hypotheses focused on Chlorogaloideae that Accepted 16 December 2014 are based on multiple independent loci and include a wide sampling of outgroups across Agavaceae. In Available online 10 January 2015 addition to chloroplast regions ndhF and trnL–trnF, we used nrDNA ITS for phylogenetic inference. Incom- plete concerted evolution of the latter is indicated by intra-individual site polymorphisms for nearly half Keywords: of the individuals. Comparisons of four coding and analysis methods for these characters indicate that the Agavaceae region remains phylogenetically informative. Our results confirm that Chlorogaloideae s.s. is not Chlorogaloideae Chlorogalum monophyletic, due to the close relationship of Schoenolirion with Hesperaloe and Hesperoyucca, as well Hesperaloe as the likely sister relationship between Hesperocallis and core Chlorogaloideae (Camassia, Chlorogalum, Hesperocallis and Hastingsia). -

José A. Mari Mut 2019
Rafinesque’s nymphs José A. Mari Mut 2019 Rafinesque’s nymphs On the cover- The Dance of Nymphs. By Willam Gale (1823-1909) © 2019 edicionesditales.info. Ediciones Digitales, Aguadilla, Puerto Rico This publication may be freely copied and distributed for educational non-profit use. i The inspiration for this short contribution was the statement made by Peter Bernhardt, on page 12 of his enjoyable Gods and Goddesses in the Garden... (2008), to the 1 effect that botanist Constantine Samuel Rafinesque had made up “out of thin air” the names of four plant genera Introduction, dedicated to nymphs whose names “no one has ever Methods and found... in the classic poems, plays, or books of mythology.” Results Rafinesque dedicated to nymphs fifty plant genera, only seven of which can be matched to a nymph known from classic works. This situation has unavoidably led to educated guesses by researchers interested in the etymology of plant names. For example, Schultes and Pease (1963) suggest that Adipe may refer to the generally fleshy flower and Cordula to a club-shaped column, of orchids. In an attempt to determine the origin of Rafinesque’s names, I extracted from Burkhardt (2018) 45 generic names dedicated by Rafinesque to nymphs. Given that with a single exception the names originated in Rafinesque’s Flora Te lu r i a n a and Sylva Teluriana, both publications were 2 browsed page by page and five more names were found, reaching the total of fifty. I then began searching for the names on the Internet and it quickly became apparent that they did not originate in the names of the classic nymphs of antiquity, but rather on the names of what Vane- Wright (2005) has called winged nymphs: butterflies! All but six of the 50 names, and all but two of the remaining 44, can be traced to a butterfly included in two works by Johann Christian Fabricius published during the late 18th century. -

Multilocus Phylogenetic Inference in Subfamily Chlorogaloideae and Related Genera of Agavaceae – Informing Questions in Taxonomy at Multiple Ranks
Molecular Phylogenetics and Evolution 84 (2015) 266–283 Contents lists available at ScienceDirect Molecular Phylogenetics and Evolution journal homepage: www.elsevier.com/locate/ympev Multilocus phylogenetic inference in subfamily Chlorogaloideae and related genera of Agavaceae – Informing questions in taxonomy at multiple ranks a, b b c d Jenny K. Archibald ⇑, Susan R. Kephart , Kathryn E. Theiss , Anna L. Petrosky , Theresa M. Culley a Biodiversity Institute and Department of Ecology and Evolutionary Biology, University of Kansas, Lawrence, KS 66045, USA b Department of Biology, Willamette University, Salem, OR 97301, USA c Department of Integrative Biology, University of California, Berkeley, CA 94709, USA d Department of Biological Sciences, University of Cincinnati, Cincinnati, OH 45221, USA article info abstract Article history: A series of taxonomic questions at the subfamilial, generic, and intrageneric levels have remained within Received 21 July 2014 subfamily Chlorogaloideae s.s. (comprising Camassia, Chlorogalum, Hastingsia, and Schoenolirion) and Revised 8 December 2014 relatives in Agavaceae. We present the first phylogenetic hypotheses focused on Chlorogaloideae that Accepted 16 December 2014 are based on multiple independent loci and include a wide sampling of outgroups across Agavaceae. In Available online 10 January 2015 addition to chloroplast regions ndhF and trnL–trnF, we used nrDNA ITS for phylogenetic inference. Incom- plete concerted evolution of the latter is indicated by intra-individual site polymorphisms for nearly half Keywords: of the individuals. Comparisons of four coding and analysis methods for these characters indicate that the Agavaceae region remains phylogenetically informative. Our results confirm that Chlorogaloideae s.s. is not Chlorogaloideae Chlorogalum monophyletic, due to the close relationship of Schoenolirion with Hesperaloe and Hesperoyucca, as well Hesperaloe as the likely sister relationship between Hesperocallis and core Chlorogaloideae (Camassia, Chlorogalum, Hesperocallis and Hastingsia). -
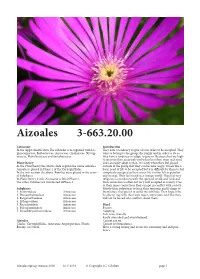
Aizoales 3-663.20.00
Aizoales 3-663.20.00 Taxonomy Introduction In the Apg2 classifcation Te suborder is recognised with Lo- Tey have a tendency to give a lot in order to be accepted. Tey phiocarpaceae, Barbeuiaceae, Aizoaceae, Gisekiaceae, Nyctag- want to belong to the group, the family and in order to do so inaceae, Phytolaccaceae and Sarcobataceae. they have a tendency to adapt, to give in. Because they are high- ly sensitive they accurately feel what the others want and need Plant theory and can easily adapt to that. It is only when they feel placed In the Plant theory the above clade is given the name Aizoales. outside of the group that they can become angry. It feels like a Aizoales is placed in Phase 2 of the Caryophyllidae. basic need of life to be accepted but it is difcult for them to feel In the frst version the above Families were placed in the sever- completely accepted as their inner life is ofen felt as peculiar al Subphases. and strange. Tey feel weird in a strange world. Tey feel very In Plant theory 2 only Aizoaceae is lef inPhase 2. religious, a connection with the spiritual world and God and Te other Families are transferred toPhase 3. that connection is ofen not very well accepted in society. Due to their inner convictions they can get in confict with society. Subphases Mostly their solution is to keep their opinions and feelings to 1. Sesuvioideae Aizoaceae themselves; they prefer to avoid the conficts. Tey hope to be 2. Drosanthemoideae Aizoaceae be able to stay with their own inner convictions and that they 3. -

Universidad Ricardo Palma
UNIVERSIDAD RICARDO PALMA ESCUELA DE POSGRADO MAESTRÍA EN ECOLOGÍA Y GESTIÓN AMBIENTAL Para optar el Grado Académico de Maestro en Ecología y Gestión Ambiental Impacto del crecimiento urbano en la alteración y degradación del ecosistema de las lomas de Villa María del Triunfo Autor: Bach. APEDJINOU, Komlavi Asesor: Dr. Arce Rojas, Rodrigo LIMA - PERÚ 2019 Página del Jurado II Dedicatoria A Dios por ser mi fortaleza y creador de nuestra casa común. A mis amadísimos padres que me dieron la vida y me enseñaron luchar para logar la meta propuesta. A mis hermanos y hermanas que siempre han sido mis apoyos en todo momento. A mis amigos y amigas que estuvieron siempre conmigo y me apoyaron en muchas situaciones. III Agradecimiento A mi asesor Dr. Arce Rojas, Rodrigo por su incondicional apoyo, interés, orientación, revisiones y observaciones oportunas durante el trayecto de este camino de investigación. Al profesor Alejandro Vílchez De los Ríos por su cariño, seguimiento, pasión; y por apoyarme a acceder a algunos datos con menos dificultad. A mis profesores de postgrado que me enseñaron y me bridaron las herramientas necesarias para abrir más mis conocimientos y mi pasión por los ecosistemas naturales. A mis compañeros y compañeras de la Escuela de Posgrado con los cuales se ha vivido e intercambiado costumbres y conocimientos. A la Asociación circuito Ecoturístico Lomas del Paraíso, cuyos integrantes me apoyaron en la recolección de información y en las visitas; a los pobladores del Paraíso y Manantial del Edén que me brindaron datos trascendentales durante las encuestas. A todas aquellas personas gentiles que me brindaron apoyo de cualquier índole. -

Phylogenetic Relationships Among Genera of Massonieae (Hyacinthaceae) Inferred from Plastid DNA and Seed Morphology
Springer-VerlagTokyo102650918-94401618-086030669031Journal of Plant ResearchJ Plant Res007610.1007/s10265-003-0076-8 J Plant Res (2003) 116:115–132 © The Botanical Society of Japan and Springer-Verlag Tokyo 2003 Digital Object Identifier (DOI) 10.1007/s10265-003-0076-8 ORIGINAL ARTICLE M. Pfosser • W. Wetschnig • S. Ungar • G. Prenner Phylogenetic relationships among genera of Massonieae (Hyacinthaceae) inferred from plastid DNA and seed morphology Received: June 26, 2002 / Accepted: December 5, 2002 / Published online: February 22, 2003 Abstract The tribe Massonieae Baker (Hyacinthaceae- Key words Hyacinthaceae · Massonieae · Molecular phy- Hyacinthoideae) presently consists of about 19 genera and logeny · Plastid DNA sequences · Scanning electron micros- 230 species distributed from Africa (south of the Sahara) to copy · Seed morphology Madagascar and India. Based on atpB and trnL-F DNA sequences the tribe is monophyletic only when the genus Pseudoprospero is excluded from Massonieae. In most trnL-F trees, this genus occupies a basal position within Introduction subfamily Hyacinthoideae and is sister to the rest of the subfamily. Molecular data suggest that the remaining gen- Previous analyses using atpB, rbcL and trnL-F data have era of Massonieae do not share common ancestry with the shown that the family Hyacinthaceae is monophyletic Eurasian/North-African tribe Hyacintheae Dumort. (Scilla, and is nested within Asparagales. Its closest relatives are Hyacinthus and allies), and thus a narrow concept of the the family Themidaceae and Aphyllanthes monspeliensis essentially Eurasian genus Scilla is supported. Members of (Chase et al. 2000; Fay et al. 2000; Pfosser and Speta 1999). well-supported clades in Massonieae usually show similari- Based on molecular, morphological, karyological and ties in seed characteristics as determined by scanning chemotaxonomical data the family can be split into the four electron microscopy.