Copia De 2019 Tesis Drmed INDICE
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

ABSTRACT MITCHELL III, ROBERT DRAKE. Global Human Health
ABSTRACT MITCHELL III, ROBERT DRAKE. Global Human Health Risks for Arthropod Repellents or Insecticides and Alternative Control Strategies. (Under the direction of Dr. R. Michael Roe). Protein-coding genes and environmental chemicals. New paradigms for human health risk assessment of environmental chemicals emphasize the use of molecular methods and human-derived cell lines. In this study, we examined the effects of the insect repellent DEET (N, N-diethyl-m-toluamide) and the phenylpyrazole insecticide fipronil (fluocyanobenpyrazole) on transcript levels in primary human hepatocytes. These chemicals were tested individually and as a mixture. RNA-Seq showed that 100 µM DEET significantly increased transcript levels for 108 genes and lowered transcript levels for 64 genes and fipronil at 10 µM increased the levels of 2,246 transcripts and decreased the levels for 1,428 transcripts. Fipronil was 21-times more effective than DEET in eliciting changes, even though the treatment concentration was 10-fold lower for fipronil versus DEET. The mixture of DEET and fipronil produced a more than additive effect (levels increased for 3,017 transcripts and decreased for 2,087 transcripts). The transcripts affected in our treatments influenced various biological pathways and processes important to normal cellular functions. Long non-protein coding RNAs and environmental chemicals. While the synthesis and use of new chemical compounds is at an all-time high, the study of their potential impact on human health is quickly falling behind. We chose to examine the effects of two common environmental chemicals, the insect repellent DEET and the insecticide fipronil, on transcript levels of long non-protein coding RNAs (lncRNAs) in primary human hepatocytes. -

Browsing Genes and Genomes with Ensembl
The Bioinformatics Roadshow Tórshavn, The Faroe Islands 28-29 November 2012 BROWSING GENES AND GENOMES WITH ENSEMBL EXERCISES AND ANSWERS 1 BROWSER 3 BIOMART 8 VARIATION 13 COMPARATIVE GENOMICS 18 2 Note: These exercises are based on Ensembl version 69 (October 2012). After in future a new version has gone live, version 69 will still be available at http://e69.ensembl.org/. If your answer doesn’t correspond with the given answer, please consult the instructor. ______________________________________________________________ BROWSER ______________________________________________________________ Exercise 1 – Exploring a gene (a) Find the human F9 (coagulation factor IX) gene. On which chromosome and which strand of the genome is this gene located? How many transcripts (splice variants) have been annotated for it? (b) What is the longest transcript? How long is the protein it encodes? Has this transcript been annotated automatically (by Ensembl) or manually (by Havana)? How many exons does it have? Are any of the exons completely or partially untranslated? (c) Have a look at the external references for ENST00000218099. What is the function of F9? (d) Is it possible to monitor expression of ENST00000218099 with the ILLUMINA HumanWG_6_V2 microarray? If so, can it also be used to monitor expression of the other two transcripts? (e) In which part (i.e. the N-terminal or C-terminal half) of the protein encoded by ENST00000218099 does its peptidase activity reside? (f) Have any missense variants been discovered for the protein encoded by ENST00000218099? (g) Is there a mouse orthologue predicted for the human F9 gene? (h) If you have yourself a gene of interest, explore what information Ensembl displays about it! ______________________________________________________________ Answer (a) 8 Go to the Ensembl homepage (http://www.ensembl.org/). -

Emerging Genetic Alterations Linked to Male Infertility: X-Chromosome Copy Number Variation and Spermatogenesis Regulatory Genes’ Expression
Central Annals of Reproductive Medicine and Treatment Bringing Excellence in Open Access Research Article *Corresponding author Susana Fernandes, Department of Genetics, Faculty of Medicine of University of Porto, Al. Prof. Hernâni Emerging Genetic Alterations Monteiro, 4200 - 319 Porto, Portugal; Tel: 351 22 551 36 47; Email: Submitted: 12 October 2016 Linked to Male Infertility: Accepted: 10 February 2017 Published: 14 February 2017 X-Chromosome Copy Number Copyright © 2017 Fernandes et al. Variation and Spermatogenesis OPEN ACCESS Keywords Regulatory Genes’ Expression • Male infertility • Spermatogenesis Catarina Costa1, Maria João Pinho1,2, Alberto Barros1,2,3, and • DNA copy number variations • Gene expression Susana Fernandes1,2* 1Department of Genetics, Faculty of Medicine of University of Porto, Portugal 2i3S – Instituto de Investigação e Inovação em Saúde, University of Porto, Portugal 3Centre for Reproductive Genetics A Barros, Portugal Abstract The etiopathogenesis of primary testicular failure remains undefined in 50% of cases. Most of these idiopathic cases probably result from genetic mutations/anomalies. Novel causes, like Copy Number Variation and gene expression profile, are being explored thanks to recent advances in the field of genetics. Our aim was to study Copy Number Variation (CNV) 67, a patient-specific CNV related to spermatogenic anomaly and evaluate the expression of regulatory genes AKAP4, responsible for sperm fibrous sheet assembly, and STAG3, essential for sister chromatid cohesion during meiosis. One hundred infertile men were tested for CNV67 with quantitative PCR (qPCR). Quantitative real-time PCR was performed to evaluate gene expression patterns of the two mentioned genes in testicular biopsies from 22 idiopathic infertile patients. CNV67 deletion was found in 2% of patients, with the same semen phenotype described in previous studies. -
![Downloaded from [266]](https://docslib.b-cdn.net/cover/7352/downloaded-from-266-347352.webp)
Downloaded from [266]
Patterns of DNA methylation on the human X chromosome and use in analyzing X-chromosome inactivation by Allison Marie Cotton B.Sc., The University of Guelph, 2005 A THESIS SUBMITTED IN PARTIAL FULFILLMENT OF THE REQUIREMENTS FOR THE DEGREE OF DOCTOR OF PHILOSOPHY in The Faculty of Graduate Studies (Medical Genetics) THE UNIVERSITY OF BRITISH COLUMBIA (Vancouver) January 2012 © Allison Marie Cotton, 2012 Abstract The process of X-chromosome inactivation achieves dosage compensation between mammalian males and females. In females one X chromosome is transcriptionally silenced through a variety of epigenetic modifications including DNA methylation. Most X-linked genes are subject to X-chromosome inactivation and only expressed from the active X chromosome. On the inactive X chromosome, the CpG island promoters of genes subject to X-chromosome inactivation are methylated in their promoter regions, while genes which escape from X- chromosome inactivation have unmethylated CpG island promoters on both the active and inactive X chromosomes. The first objective of this thesis was to determine if the DNA methylation of CpG island promoters could be used to accurately predict X chromosome inactivation status. The second objective was to use DNA methylation to predict X-chromosome inactivation status in a variety of tissues. A comparison of blood, muscle, kidney and neural tissues revealed tissue-specific X-chromosome inactivation, in which 12% of genes escaped from X-chromosome inactivation in some, but not all, tissues. X-linked DNA methylation analysis of placental tissues predicted four times higher escape from X-chromosome inactivation than in any other tissue. Despite the hypomethylation of repetitive elements on both the X chromosome and the autosomes, no changes were detected in the frequency or intensity of placental Cot-1 holes. -

Reduced Transfer Coefficient of Carbon Monoxide in Pulmonary Arterial Hypertension Implicates Rare Protein-Truncating Variants I
bioRxiv preprint doi: https://doi.org/10.1101/2019.12.11.871210; this version posted December 12, 2019. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY-NC-ND 4.0 International license. Reduced transfer coefficient of carbon monoxide in pulmonary arterial hypertension implicates rare protein- truncating variants in KDR Genotype-phenotype inference reveals novel PAH risk genes Emilia M. Swietlik, M.D.[1], Daniel Greene, PhD[2,3], Na Zhu, PhD[4,5], Karyn Megy, PhD[2,3], Marcella Cogliano, M.Sc.[6], Smitha Rajaram, M.D.[7], Divya Pandya, M.Sc.[1], Tobias Tilly, M.Sc.[1], Katie A. Lutz, BS[8], Carrie C. L. Welch, PhD[4], Michael W. Pauciulo, BS, MBA[8,9], Laura Southgate, PhD[10], Jennifer M. Martin, M.St.[3], Carmen M. Treacy, B.Sc.[1], Harm J. Bogaard, M.D., PhD[11], Colin Church, PhD[12], Gerry Coghlan, M.D.[13], Anna W. Coleman, MS[8], Robin Condliffe, M.D.[14], Mélanie Eyries, PhD[15], Henning Gall, M.D., PhD[16], Stefano Ghio, M.D.[17], Simon Holden, PhD[18], Luke Howard, M.D., PhD.[19], David G. Kiely, M.D.[14], Gabor Kovacs, M.D.[20,21], Jim Lordan, PhD[22], Rajiv D. Machado, PhD[10], Robert V. MacKenzie Ross, M.B, B.Chir[23], Shahin Moledina, M.B.Ch.B.[24], David Montani, M.D., PhD[25], Horst Olschewski, M.D.[20,21], Joanna Pepke-Zaba, PhD[26], Christopher J. -

Type of the Paper (Article
Article Differential Expression Profile of lncRNAs from Primary Human Hepatocytes Following DEET and Fipronil Exposure Robert D. Mitchell III 1, Andrew D. Wallace 2, Ernest Hodgson 2,3 and R. Michael Roe 1,* 1 Department of Entomology and Plant Pathology, North Carolina Agromedicine Institute, Campus Box 7647, 3230 Ligon Street, North Carolina State University, Raleigh, NC 27695, USA; [email protected] (R.D.M.) 2 Toxicology Program, Department of Biology, North Carolina State University, Raleigh, NC 27695, USA; [email protected] (A.D.W.); [email protected] (E.H.) 3 Department of Applied Ecology, Toxicology Program, Department of Biology, North Carolina Agromedicine Institute, North Carolina State University, Raleigh, NC 27695, USA * Correspondence: [email protected]; Tel.: +1-919-515-4325 Received: 15 August 2017; Accepted: 4 October 2017; Published: 7 October 2017 Abstract: While the synthesis and use of new chemical compounds is at an all-time high, the study of their potential impact on human health is quickly falling behind, and new methods are needed to assess their impact. We chose to examine the effects of two common environmental chemicals, the insect repellent N,N-diethyl-m-toluamide (DEET) and the insecticide fluocyanobenpyrazole (fipronil), on transcript levels of long non-protein coding RNAs (lncRNAs) in primary human hepatocytes using a global RNA-Seq approach. While lncRNAs are believed to play a critical role in numerous important biological processes, many still remain uncharacterized, and their functions and modes of action remain largely unclear, especially in relation to environmental chemicals. RNA- Seq showed that 100 µM DEET significantly increased transcript levels for 2 lncRNAs and lowered transcript levels for 18 lncRNAs, while fipronil at 10 µM increased transcript levels for 76 lncRNAs and decreased levels for 193 lncRNAs. -

Mechanism of Ant1-Induced Human Diseases and Stress Signaling
MECHANISM OF ANT1-INDUCED HUMAN DISEASES AND STRESS SIGNALING Item Type Dissertation Authors Liu, Yaxin Rights Attribution-NonCommercial-NoDerivatives 4.0 International Download date 09/10/2021 19:50:21 Item License http://creativecommons.org/licenses/by-nc-nd/4.0/ Link to Item http://hdl.handle.net/20.500.12648/1772 MECHANISM OF ANT1-INDUCED HUMAN DISEASES AND STRESS SIGNALING Yaxin Liu A Dissertation in Department of Biochemistry and Molecular Biology Submitted in partial fulfillment of the requirements for the degree of Doctor of Philosophy in the College of Graduate Studies of State University of New York, Upstate Medical University. Approved Date Abstract Mechanism of Ant1-induced Human Diseases and Stress Signaling Yaxin Liu Xin Jie Chen Adenine nucleotide translocase (Ant) is a mitochondrial inner membrane protein, the primary function of which is to mediate the ADP/ATP exchange across the inner membrane. Missense mutations in Ant1, the skeletal muscle- and heart-specific isoform, induce human disorders including autosomal dominant Progressive External Opthalmoplegia (adPEO), cardiomyopathy and myopathy. Several models were proposed to interpret the pathogenesis of mutant Ant1-induced diseases, but no consensus has been reached. Our lab has previously found that mutant Aac2, the homolog of Ant1 in yeast, causes cell death due to the mitochondrial biogenesis defect. In the present study, we provided biochemical evidence supporting the idea that the mutant Aac2 proteins are misfolded, which derails the proteostasis on the inner membrane. We found that the assembly and stability of multiple protein complexes on the inner membrane are affected, including those involved in mitochondrial respiration and protein transport. -

Identification of Cancer-Specific Transcripts
Identification of cancer-specific transcripts: With emphasis on the hunt for fusion genes in colorectal cancer Marthe Eken Thesis for the master’s degree in Molecular Bioscience at Department of Molecular Bioscience (IMBV), Faculty of Mathematics and Natural sciences UNIVERSITY OF OSLO December 2008 2 Acknowledgements This work was carried out in the project Group of Genome Biology, at the Department of Cancer Prevention, Rikshospitalet-Radiumhospitalet Medical Center, from March 2007 to December 2008. First of all, I would like to thank my supervisor, Rolf I. Skotheim, for his great support throughout the project, for always taking the time to answer my many questions, and for his everlasting patience with my forgotten italics. I also wish to thank my co-supervisor and head of the department, Ragnhild A. Lothe, for giving me the opportunity to be part of such an excellent group. I greatly appreciate the other members of the department for making this a great place to work, especially Guro for being my lab-oracle, Anita for providing microarray data, Zere for helping me with the cloning, and Hilde for our many discussions. I am grateful to my mother for all her great advices through the years and for always being there for me, and to my father for telling me I could do anything. Finally, special thanks go to my fiancé, Joachim, for his overwhelming patience and support, for always believing in me, and for making me believe in myself. Oslo, December 2008 Marthe Eken Table of contents TABLE OF CONTENTS..................................................................................................................... 4 SUMMARY .......................................................................................................................................... 6 ABBREVIATIONS .............................................................................................................................. 8 GENE SYMBOLS ............................................................................................................................... -

LCR-Initiated Rearrangements at the IDS Locus, Completed with Alu-Mediated Recombination Or Non-Homologous End Joining
Journal of Human Genetics (2011) 56, 516–523 & 2011 The Japan Society of Human Genetics All rights reserved 1434-5161/11 $32.00 www.nature.com/jhg ORIGINAL ARTICLE LCR-initiated rearrangements at the IDS locus, completed with Alu-mediated recombination or non-homologous end joining Junko Oshima1,4, Jennifer A Lee1, Amy M Breman1, Priscilla H Fernandes1, Dusica Babovic-Vuksanovic2, Patricia A Ward1, Lynne A Wolfe3,5, Christine M Eng1 and Daniela del Gaudio1,6 Mucopolysaccharidosis type II (MPS II) is caused by mutations in the IDS gene, which encodes the lysosomal enzyme iduronate-2-sulfatase. In B20% of MPS II patients the disorder is caused by gross IDS structural rearrangements. We identified two male cases harboring complex rearrangements involving the IDS gene and the nearby pseudogene, IDSP1, which has been annotated as a low-copy repeat (LCR). In both cases the rearrangement included a partial deletion of IDS and an inverted insertion of the neighboring region. In silico analyses revealed the presence of repetitive elements as well as LCRs at the junctions of rearrangements. Our models illustrate two alternative consequences of rearrangements initiated by non-allelic homologous recombination of LCRs: resolution by a second recombination event (that is, Alu-mediated recombination), or resolution by non-homologous end joining repair. These complex rearrangements have the potential to be recurrent and may be present among those MSP II cases with previously uncharacterized aberrations involving IDS. Journal of Human Genetics (2011) 56, 516–523; doi:10.1038/jhg.2011.51; published online 19 May 2011 Keywords: array CGH; chromosomal rearrangements; clinical molecular genetics; Hunter syndrome; lysosomal storage diseases; mucopolysaccharidoses; X-linked diseases INTRODUCTION chromosome Xq28.2.2 Its 1650-bp open reading frame encodes the Mucopolysaccharidosis type II (MPS II; Hunter syndrome; OMIM # 550 amino-acid lysosomal enzyme iduronate-2-sulfatase.3 A1.4-kb 309900) is a rare X-linked recessive lysosomal storage disorder. -
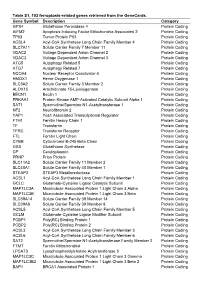
Table S1. 103 Ferroptosis-Related Genes Retrieved from the Genecards
Table S1. 103 ferroptosis-related genes retrieved from the GeneCards. Gene Symbol Description Category GPX4 Glutathione Peroxidase 4 Protein Coding AIFM2 Apoptosis Inducing Factor Mitochondria Associated 2 Protein Coding TP53 Tumor Protein P53 Protein Coding ACSL4 Acyl-CoA Synthetase Long Chain Family Member 4 Protein Coding SLC7A11 Solute Carrier Family 7 Member 11 Protein Coding VDAC2 Voltage Dependent Anion Channel 2 Protein Coding VDAC3 Voltage Dependent Anion Channel 3 Protein Coding ATG5 Autophagy Related 5 Protein Coding ATG7 Autophagy Related 7 Protein Coding NCOA4 Nuclear Receptor Coactivator 4 Protein Coding HMOX1 Heme Oxygenase 1 Protein Coding SLC3A2 Solute Carrier Family 3 Member 2 Protein Coding ALOX15 Arachidonate 15-Lipoxygenase Protein Coding BECN1 Beclin 1 Protein Coding PRKAA1 Protein Kinase AMP-Activated Catalytic Subunit Alpha 1 Protein Coding SAT1 Spermidine/Spermine N1-Acetyltransferase 1 Protein Coding NF2 Neurofibromin 2 Protein Coding YAP1 Yes1 Associated Transcriptional Regulator Protein Coding FTH1 Ferritin Heavy Chain 1 Protein Coding TF Transferrin Protein Coding TFRC Transferrin Receptor Protein Coding FTL Ferritin Light Chain Protein Coding CYBB Cytochrome B-245 Beta Chain Protein Coding GSS Glutathione Synthetase Protein Coding CP Ceruloplasmin Protein Coding PRNP Prion Protein Protein Coding SLC11A2 Solute Carrier Family 11 Member 2 Protein Coding SLC40A1 Solute Carrier Family 40 Member 1 Protein Coding STEAP3 STEAP3 Metalloreductase Protein Coding ACSL1 Acyl-CoA Synthetase Long Chain Family Member 1 Protein -

And Tetranucleotide Short Tandem Repeats N
Nazaripanah et al. Human Genomics (2018) 12:17 https://doi.org/10.1186/s40246-018-0149-3 PRIMARYRESEARCH Open Access Genome-scale portrait and evolutionary significance of human-specific core promoter tri- and tetranucleotide short tandem repeats N. Nazaripanah1, F. Adelirad1, A. Delbari1, R. Sahaf1, T. Abbasi-Asl2 and M. Ohadi1* Abstract Background: While there is an ongoing trend to identify single nucleotide substitutions (SNSs) that are linked to inter/intra-species differences and disease phenotypes, short tandem repeats (STRs)/microsatellites may be of equal (if not more) importance in the above processes. Genes that contain STRs in their promoters have higher expression divergence compared to genes with fixed or no STRs in the gene promoters. In line with the above, recent reports indicate a role of repetitive sequences in the rise of young transcription start sites (TSSs) in human evolution. Results: Following a comparative genomics study of all human protein-coding genes annotated in the GeneCards database, here we provide a genome-scale portrait of human-specific short- and medium-size (≥ 3-repeats) tri- and tetranucleotide STRs and STR motifs in the critical core promoter region between − 120 and + 1 to the TSS and evidence of skewing of this compartment in reference to the STRs that are not human-specific (Levene’s test p <0. 001). Twenty-five percent and 26% enrichment of human-specific transcripts was detected in the tri and tetra human-specific compartments (mid-p < 0.00002 and mid-p < 0.002, respectively). Conclusion: Our findings provide the first evidence of genome-scale skewing of STRs at a specific region of the human genome and a link between a number of these STRs and TSS selection/transcript specificity. -
High Resolution X Chromosome-Specific Array-CGH Detects New Cnvs in Infertile Males
High Resolution X Chromosome-Specific Array-CGH Detects New CNVs in Infertile Males Csilla Krausz1,2*, Claudia Giachini1, Deborah Lo Giacco2,3, Fabrice Daguin1, Chiara Chianese1, Elisabet Ars3, Eduard Ruiz-Castane2, Gianni Forti4, Elena Rossi5 1 Unit of Sexual Medicine and Andrology, Molecular Genetic Laboratory, Department of Clinical Physiopathology, University of Florence, Florence, Italy, 2 Andrology Service, Fundacio´ Puigvert, Barcelona, Spain, 3 Molecular Biology Laboratory, Fundacio´ Puigvert, Universitat Auto`noma de Barcelona, Barcelona, Spain, 4 Endocrinology Unit, Department of Clinical Physiopathology, University of Florence, Florence, Italy, 5 Biology and Medical Genetics, University of Pavia, Pavia, Italy Abstract Context: The role of CNVs in male infertility is poorly defined, and only those linked to the Y chromosome have been the object of extensive research. Although it has been predicted that the X chromosome is also enriched in spermatogenesis genes, no clinically relevant gene mutations have been identified so far. Objectives: In order to advance our understanding of the role of X-linked genetic factors in male infertility, we applied high resolution X chromosome specific array-CGH in 199 men with different sperm count followed by the analysis of selected, patient-specific deletions in large groups of cases and normozoospermic controls. Results: We identified 73 CNVs, among which 55 are novel, providing the largest collection of X-linked CNVs in relation to spermatogenesis. We found 12 patient-specific deletions with potential clinical implication. Cancer Testis Antigen gene family members were the most frequently affected genes, and represent new genetic targets in relationship with altered spermatogenesis. One of the most relevant findings of our study is the significantly higher global burden of deletions in patients compared to controls due to an excessive rate of deletions/person (0.57 versus 0.21, respectively; p = 8.78561026) and to a higher mean sequence loss/person (11.79 Kb and 8.13 Kb, respectively; p = 3.43561024).