Swissprot Genetrail Acc. No. ID Exp1 Exp2 Exp1 Exp2 Exp1 Exp2
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Reprogramming of Trna Modifications Controls the Oxidative Stress Response by Codon-Biased Translation of Proteins
Reprogramming of tRNA modifications controls the oxidative stress response by codon-biased translation of proteins The MIT Faculty has made this article openly available. Please share how this access benefits you. Your story matters. Citation Chan, Clement T.Y. et al. “Reprogramming of tRNA Modifications Controls the Oxidative Stress Response by Codon-biased Translation of Proteins.” Nature Communications 3 (2012): 937. As Published http://dx.doi.org/10.1038/ncomms1938 Publisher Nature Publishing Group Version Author's final manuscript Citable link http://hdl.handle.net/1721.1/76775 Terms of Use Article is made available in accordance with the publisher's policy and may be subject to US copyright law. Please refer to the publisher's site for terms of use. Reprogramming of tRNA modifications controls the oxidative stress response by codon-biased translation of proteins Clement T.Y. Chan,1,2 Yan Ling Joy Pang,1 Wenjun Deng,1 I. Ramesh Babu,1 Madhu Dyavaiah,3 Thomas J. Begley3 and Peter C. Dedon1,4* 1Department of Biological Engineering, 2Department of Chemistry and 4Center for Environmental Health Sciences, Massachusetts Institute of Technology, Cambridge, MA 02139; 3College of Nanoscale Science and Engineering, University at Albany, SUNY, Albany, NY 12203 * Corresponding author: PCD, Department of Biological Engineering, NE47-277, Massachusetts Institute of Technology, 77 Massachusetts Avenue, Cambridge, MA 02139; tel 617-253-8017; fax 617-324-7554; email [email protected] 2 ABSTRACT Selective translation of survival proteins is an important facet of cellular stress response. We recently demonstrated that this translational control involves a stress-specific reprogramming of modified ribonucleosides in tRNA. -

Allele-Specific Expression of Ribosomal Protein Genes in Interspecific Hybrid Catfish
Allele-specific Expression of Ribosomal Protein Genes in Interspecific Hybrid Catfish by Ailu Chen A dissertation submitted to the Graduate Faculty of Auburn University in partial fulfillment of the requirements for the Degree of Doctor of Philosophy Auburn, Alabama August 1, 2015 Keywords: catfish, interspecific hybrids, allele-specific expression, ribosomal protein Copyright 2015 by Ailu Chen Approved by Zhanjiang Liu, Chair, Professor, School of Fisheries, Aquaculture and Aquatic Sciences Nannan Liu, Professor, Entomology and Plant Pathology Eric Peatman, Associate Professor, School of Fisheries, Aquaculture and Aquatic Sciences Aaron M. Rashotte, Associate Professor, Biological Sciences Abstract Interspecific hybridization results in a vast reservoir of allelic variations, which may potentially contribute to phenotypical enhancement in the hybrids. Whether the allelic variations are related to the downstream phenotypic differences of interspecific hybrid is still an open question. The recently developed genome-wide allele-specific approaches that harness high- throughput sequencing technology allow direct quantification of allelic variations and gene expression patterns. In this work, I investigated allele-specific expression (ASE) pattern using RNA-Seq datasets generated from interspecific catfish hybrids. The objective of the study is to determine the ASE genes and pathways in which they are involved. Specifically, my study investigated ASE-SNPs, ASE-genes, parent-of-origins of ASE allele and how ASE would possibly contribute to heterosis. My data showed that ASE was operating in the interspecific catfish system. Of the 66,251 and 177,841 SNPs identified from the datasets of the liver and gill, 5,420 (8.2%) and 13,390 (7.5%) SNPs were identified as significant ASE-SNPs, respectively. -

Supplementary Materials
1 Supplementary Materials: Supplemental Figure 1. Gene expression profiles of kidneys in the Fcgr2b-/- and Fcgr2b-/-. Stinggt/gt mice. (A) A heat map of microarray data show the genes that significantly changed up to 2 fold compared between Fcgr2b-/- and Fcgr2b-/-. Stinggt/gt mice (N=4 mice per group; p<0.05). Data show in log2 (sample/wild-type). 2 Supplemental Figure 2. Sting signaling is essential for immuno-phenotypes of the Fcgr2b-/-lupus mice. (A-C) Flow cytometry analysis of splenocytes isolated from wild-type, Fcgr2b-/- and Fcgr2b-/-. Stinggt/gt mice at the age of 6-7 months (N= 13-14 per group). Data shown in the percentage of (A) CD4+ ICOS+ cells, (B) B220+ I-Ab+ cells and (C) CD138+ cells. Data show as mean ± SEM (*p < 0.05, **p<0.01 and ***p<0.001). 3 Supplemental Figure 3. Phenotypes of Sting activated dendritic cells. (A) Representative of western blot analysis from immunoprecipitation with Sting of Fcgr2b-/- mice (N= 4). The band was shown in STING protein of activated BMDC with DMXAA at 0, 3 and 6 hr. and phosphorylation of STING at Ser357. (B) Mass spectra of phosphorylation of STING at Ser357 of activated BMDC from Fcgr2b-/- mice after stimulated with DMXAA for 3 hour and followed by immunoprecipitation with STING. (C) Sting-activated BMDC were co-cultured with LYN inhibitor PP2 and analyzed by flow cytometry, which showed the mean fluorescence intensity (MFI) of IAb expressing DC (N = 3 mice per group). 4 Supplemental Table 1. Lists of up and down of regulated proteins Accession No. -

Immunoprecipitation and Mass Spectrometry Defines an Extensive
BRES : 44759 Model7 pp: À 1221ðcol:fig: : NILÞ brain research ] ( ]]]]) ]]]– ]]] Available online at www.sciencedirect.com 121 122 123 124 125 126 www.elsevier.com/locate/brainres 127 128 129 Review 130 131 fi 132 Immunoprecipitation and mass spectrometry de nes 133 – 134 an extensive RBM45 protein protein interaction 135 Q2 136 network 137 138 a a,b a a c 139 Yang Li , Mahlon Collins , Jiyan An , Rachel Geiser , Tony Tegeler , c c c a,b,n 140 Q1 Kristine Tsantilas , Krystine Garcia , Patrick Pirrotte , Robert Bowser 141 aDivisions of Neurology and Neurobiology, Barrow Neurological Institute, St. Joseph's Hospital and Medical Center, 142 Phoenix, AZ 85013, USA 143 bUniversity of Pittsburgh School of Medicine, Pittsburgh, PA 15261, USA 144 cCenter for Proteomics, TGen (Translational Genomics Research Institute), Phoenix, AZ 85004, USA 145 146 147 article info abstract 148 149 Article history: The pathological accumulation of RNA-binding proteins (RBPs) within inclusion bodies is a 150 Received 30 January 2016 hallmark of amyotrophic lateral sclerosis (ALS) and frontotemporal lobar degeneration 151 Received in revised form (FTLD). RBP aggregation results in both toxic gain and loss of normal function. Determining 152 25 February 2016 the protein binding partners and normal functions of disease-associated RBPs is necessary 153 Accepted 28 February 2016 to fully understand molecular mechanisms of RBPs in disease. Herein, we characterized 154 the protein–protein interactions (PPIs) of RBM45, a RBP that localizes to inclusions in ALS/ 155 – fi Keywords: FTLD. Using immunoprecipitation coupled to mass spectrometry (IP MS), we identi ed 132 156 fi RBM45 proteins that speci cally interact with RBM45 within HEK293 cells. -
Attachment PDF Icon
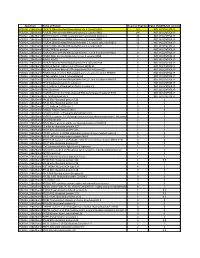
Spectrum Name of Protein Count of Peptides Ratio (POL2RA/IgG control) POLR2A_228kdBand POLR2A DNA-directed RNA polymerase II subunit RPB1 197 NOT IN CONTROL IP POLR2A_228kdBand POLR2B DNA-directed RNA polymerase II subunit RPB2 146 NOT IN CONTROL IP POLR2A_228kdBand RPAP2 Isoform 1 of RNA polymerase II-associated protein 2 24 NOT IN CONTROL IP POLR2A_228kdBand POLR2G DNA-directed RNA polymerase II subunit RPB7 23 NOT IN CONTROL IP POLR2A_228kdBand POLR2H DNA-directed RNA polymerases I, II, and III subunit RPABC3 19 NOT IN CONTROL IP POLR2A_228kdBand POLR2C DNA-directed RNA polymerase II subunit RPB3 17 NOT IN CONTROL IP POLR2A_228kdBand POLR2J RPB11a protein 7 NOT IN CONTROL IP POLR2A_228kdBand POLR2E DNA-directed RNA polymerases I, II, and III subunit RPABC1 8 NOT IN CONTROL IP POLR2A_228kdBand POLR2I DNA-directed RNA polymerase II subunit RPB9 9 NOT IN CONTROL IP POLR2A_228kdBand ALMS1 ALMS1 3 NOT IN CONTROL IP POLR2A_228kdBand POLR2D DNA-directed RNA polymerase II subunit RPB4 6 NOT IN CONTROL IP POLR2A_228kdBand GRINL1A;Gcom1 Isoform 12 of Protein GRINL1A 6 NOT IN CONTROL IP POLR2A_228kdBand RECQL5 Isoform Beta of ATP-dependent DNA helicase Q5 3 NOT IN CONTROL IP POLR2A_228kdBand POLR2L DNA-directed RNA polymerases I, II, and III subunit RPABC5 5 NOT IN CONTROL IP POLR2A_228kdBand KRT6A Keratin, type II cytoskeletal 6A 3 NOT IN CONTROL IP POLR2A_228kdBand POLR2K DNA-directed RNA polymerases I, II, and III subunit RPABC4 2 NOT IN CONTROL IP POLR2A_228kdBand RFC4 Replication factor C subunit 4 1 NOT IN CONTROL IP POLR2A_228kdBand RFC2 -
Supplementary Table 1. the List of Proteins with at Least 2 Unique
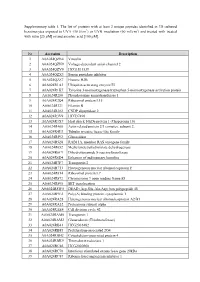
Supplementary table 1. The list of proteins with at least 2 unique peptides identified in 3D cultured keratinocytes exposed to UVA (30 J/cm2) or UVB irradiation (60 mJ/cm2) and treated with treated with rutin [25 µM] or/and ascorbic acid [100 µM]. Nr Accession Description 1 A0A024QZN4 Vinculin 2 A0A024QZN9 Voltage-dependent anion channel 2 3 A0A024QZV0 HCG1811539 4 A0A024QZX3 Serpin peptidase inhibitor 5 A0A024QZZ7 Histone H2B 6 A0A024R1A3 Ubiquitin-activating enzyme E1 7 A0A024R1K7 Tyrosine 3-monooxygenase/tryptophan 5-monooxygenase activation protein 8 A0A024R280 Phosphoserine aminotransferase 1 9 A0A024R2Q4 Ribosomal protein L15 10 A0A024R321 Filamin B 11 A0A024R382 CNDP dipeptidase 2 12 A0A024R3V9 HCG37498 13 A0A024R3X7 Heat shock 10kDa protein 1 (Chaperonin 10) 14 A0A024R408 Actin related protein 2/3 complex, subunit 2, 15 A0A024R4U3 Tubulin tyrosine ligase-like family 16 A0A024R592 Glucosidase 17 A0A024R5Z8 RAB11A, member RAS oncogene family 18 A0A024R652 Methylenetetrahydrofolate dehydrogenase 19 A0A024R6C9 Dihydrolipoamide S-succinyltransferase 20 A0A024R6D4 Enhancer of rudimentary homolog 21 A0A024R7F7 Transportin 2 22 A0A024R7T3 Heterogeneous nuclear ribonucleoprotein F 23 A0A024R814 Ribosomal protein L7 24 A0A024R872 Chromosome 9 open reading frame 88 25 A0A024R895 SET translocation 26 A0A024R8W0 DEAD (Asp-Glu-Ala-Asp) box polypeptide 48 27 A0A024R9E2 Poly(A) binding protein, cytoplasmic 1 28 A0A024RA28 Heterogeneous nuclear ribonucleoprotein A2/B1 29 A0A024RA52 Proteasome subunit alpha 30 A0A024RAE4 Cell division cycle 42 31 -

Inhibition of the MID1 Protein Complex
Matthes et al. Cell Death Discovery (2018) 4:4 DOI 10.1038/s41420-017-0003-8 Cell Death Discovery ARTICLE Open Access Inhibition of the MID1 protein complex: a novel approach targeting APP protein synthesis Frank Matthes1,MoritzM.Hettich1, Judith Schilling1, Diana Flores-Dominguez1, Nelli Blank1, Thomas Wiglenda2, Alexander Buntru2,HannaWolf1, Stephanie Weber1,InaVorberg 1, Alina Dagane2, Gunnar Dittmar2,3,ErichWanker2, Dan Ehninger1 and Sybille Krauss1 Abstract Alzheimer’s disease (AD) is characterized by two neuropathological hallmarks: senile plaques, which are composed of amyloid-β (Aβ) peptides, and neurofibrillary tangles, which are composed of hyperphosphorylated tau protein. Aβ peptides are derived from sequential proteolytic cleavage of the amyloid precursor protein (APP). In this study, we identified a so far unknown mode of regulation of APP protein synthesis involving the MID1 protein complex: MID1 binds to and regulates the translation of APP mRNA. The underlying mode of action of MID1 involves the mTOR pathway. Thus, inhibition of the MID1 complex reduces the APP protein level in cultures of primary neurons. Based on this, we used one compound that we discovered previously to interfere with the MID1 complex, metformin, for in vivo experiments. Indeed, long-term treatment with metformin decreased APP protein expression levels and consequently Aβ in an AD mouse model. Importantly, we have initiated the metformin treatment late in life, at a time-point where mice were in an already progressed state of the disease, and could observe an improved behavioral phenotype. These 1234567890 1234567890 findings together with our previous observation, showing that inhibition of the MID1 complex by metformin also decreases tau phosphorylation, make the MID1 complex a particularly interesting drug target for treating AD. -

PNAS 07-04849-SI Table 3. 6-18-2007
Table 5. β-arrestin 2-interacting proteins under nonstimulated (-) condition IPI accession Swiss- Number of Prot Gene symbol Protein name experiments accession number number detected Signal transduction Adaptor proteins IPI00027355 P32121 ARRB2 β-arrestin 2 6 IPI00293857 P49407 ARRB1 β-arrestin 1 6 IPI00021353 P10523 SAG S-arrestin (Retinal S-antigen) (48 kDa protein) (S-AG) (Rod photoreceptor arrestin) 2 IPI00003917 P36575 ARR3 X-arrestin (Arrestin-C) (Cone arrestin) (cArr) (Retinal cone arrestin-3) (C-arrestin) 2 IPI00216318 P31946 YWHAB 14-3-3 β/α (14-3-3 protein beta/alpha) (Protein kinase C inhibitor protein 1) (KCIP-1) (Protein 1054) 2 IPI00220642 P61981 YWHAG 14-3-3 γ (14-3-3 protein gamma) (Protein kinase C inhibitor protein 1) (KCIP-1) 2 IPI00018146 P27348 YWHAQ 14-3-3 θ (14-3-3 protein tau) (14-3-3 protein theta) (14-3-3 protein T-cell) (HS1 protein) 2 IPI00216319 Q04917 YWHAH 14-3-3 η (14-3-3 protein eta) (Protein AS1) 2 IPI00000816 P62258 YWHAE 14-3-3 ε (14-3-3 protein epsilon) (14-3-3E) 2 Protein kinases IPI00027251 Q15208 STK38 STK38 (Serine/threonine-protein kinase 38) (NDR1 protein kinase) (Nuclear Dbf2-related kinase 1) 4 SCY1-like 2 (SCY1-like 2 protein) (coated vesicle-associated kinase of 104 kDa) (Eukaryotic protein IPI00396218 Q6P3W7 SCYL2 2 kinase family protein) (CDNA FLJ10074 fis, clone HEMBA1001744, weakly similar to SCY1 IPI00013835 Q13574 DGKZ DGK ζ (Diacylglycerol kinase zeta) (Diglyceride kinase zeta) (DGK-zeta) (DAG kinase zeta) 2 DGK ε (Diacylglycerol kinase epsilon) (Diglyceride kinase epsilon) (DGK-epsilon) -

Supplementary Information
1 Supplementary Information 2 PARK2 promotes mitochondrial pathway of apoptosis and 3 antimicrotubule drugs chemosensitivity via degradation of 4 phospho-BCL-2 5 Hengxing Chen1 †, Yun Li1 †, Yu Li1, Zhen Chen1, Limin Xie1, Wenjia Li1, Yuanxin Zhu1, Hong 6 Xue2, H. Phillip Koeffler3,4, Wenjing Wu5, Kaishun Hu1 *, Dong Yin1, * 7 8 Table of contents 9 Supplementary Materials and Methods……………………………………………2 10 Supplementary Figure. 1…………………………………………………………….6 11 Supplementary Figure. 2…………………………………………………………….7 12 Supplementary Figure. 3…………………………………………………………….8 13 Supplementary Figure. 4…………………………………………………………….9 14 Supplementary Figure. 5……………………………………………………………10 15 Supplementary Figure. 6……………………………………………………………11 16 Supplementary Figure. 7……………………………………………………………12 17 Supplementary Table 1……………………………………………...………………13 18 Supplementary Table 2……………………………………………………...………22 19 20 21 22 1 23 24 25 Supplementary Materials and Methods 26 Cell lines 27 The human breast cell lines MCF-7, MDA-MB-231, T47D, ZR-75-30 and MDA-MB-134-VI 28 were obtained from ATCC. MDA-MB-231 were maintained in DMEM supplemented with 29 10% (v/v) FBS and 1% (v/v) Penicillin-Streptomycin, at 37°C, 5% CO2 in a humidified 30 atmosphere. MCF-7, T47D and ZR-75-30 grew in RPMI 1640 with 10% fetal bovine serum 31 and 1% Penicillin-Streptomycin. MDA-MB-134-VI were cultured in Leibovitz's L-15 32 Medium with 20% (v/v) FBS and 1% (v/v) Penicillin-Streptomycin. 33 34 Flow cytometric analysis 35 Cells were treated with designated chemotherapy drugs and then collected for Annexin V and 36 JC-1 analyses. For Annexin V analysis, apoptosis in breast cancer cells was detected using the 37 Annexin V-FITC Apoptosis Detection Kit (Invitrogen) according to the manufacturer's 38 protocol. -

Supplementary Table S1. Protein Expression Changes in the Functionally Annotated Categories Within Cluster from Figure 8A
Supplementary Table S1. Protein expression changes in the functionally annotated categories within cluster from Figure 8A. Acute phase response signaling Human equivalent Zq (log2 FC) FDR Accession No. Ac. No. No. Protein 120 1 4 7 120 1 4 7 peptides Q13233 F1SLK9 PIG Uncharacterized protein MAP3K1 0.09 0.00 0.00 0.55 1 - I3LQR9 PIG Fibrinogen alpha chain 0.89 0.23 0.03 0.11 11 - F1RX36 PIG Fibrinogen alpha chain 0.97 0.09 0.00 0.09 4 P02675 I3L651 PIG Uncharacterized protein 0.91 0.24 0.05 0.23 27 - Q8SPS7 PIG Haptoglobin 0.11 0.02 0.00 0.36 14 P45985 F1SS51 PIG Uncharacterized protein (Fragment) MAP2K4 0.26 0.06 0.09 0.33 1 - P00747 HUMAN Plasminogen 0.93 0.00 0.02 0.67 1 - P18648 PIG Apolipoprotein A-I 0.36 0.19 0.64 0.86 34 P02679 F1RX35 PIG Uncharacterized protein LOC100627396 0.99 0.40 0.02 0.11 23 P04196 F1SFI5 PIG Uncharacterized protein HRG 0.44 0.04 0.55 0.82 12 - P27485 PIG Retinol-binding protein 4 0.34 0.30 0.55 0.81 2 - F1S0J2 PIG Uncharacterized protein C4BPA 0.58 0.17 0.15 0.65 4 - P08835 PIG Serum albumin 0.04 0.09 0.39 0.75 27 - P06867 PIG Plasminogen 0.94 0.17 0.08 0.82 13 - O19063 PIG Serum amyloid P-component 0.93 0.16 0.09 0.85 6 - O19062 PIG C-reactive protein 0.93 0.01 0.00 0.78 7 - P00450 HUMAN Ceruloplasmin 0.32 0.61 0.36 0.41 1 - F1SB81 PIG Plasminogen 0.99 0.17 0.14 0.52 2 - Q19AZ8 PIG Prothrombin 0.36 0.08 0.54 0.90 6 - I3L818 PIG Uncharacterized protein (Fragment) SERPINF2 0.91 0.59 0.12 0.65 4 P05546 F1RKY2 PIG Uncharacterized protein SERPIND1 0.38 0.44 0.72 0.86 9 P02749 I3LGN5 PIG Uncharacterized protein -

Datasheet: VPA00367 Product Details
Datasheet: VPA00367 Description: RABBIT ANTI RPS3 Specificity: RPS3 Format: Purified Product Type: PrecisionAb™ Polyclonal Isotype: Polyclonal IgG Quantity: 100 µl Product Details Applications This product has been reported to work in the following applications. This information is derived from testing within our laboratories, peer-reviewed publications or personal communications from the originators. Please refer to references indicated for further information. For general protocol recommendations, please visit www.bio-rad-antibodies.com/protocols. Yes No Not Determined Suggested Dilution Western Blotting 1/1000 PrecisionAb antibodies have been extensively validated for the western blot application. The antibody has been validated at the suggested dilution. Where this product has not been tested for use in a particular technique this does not necessarily exclude its use in such procedures. Further optimization may be required dependant on sample type. Target Species Human Species Cross Reacts with: Mouse Reactivity N.B. Antibody reactivity and working conditions may vary between species. Product Form Purified IgG - liquid Preparation Rabbit polyclonal antibody purified by affinity chromatography Buffer Solution Phosphate buffered saline Preservative 0.09% Sodium Azide (NaN3) Stabilisers 2% Sucrose Immunogen Synthetic peptide corresponding to the C-terminal region of human RPS3 External Database Links UniProt: P23396 Related reagents Entrez Gene: 6188 RPS3 Related reagents Specificity Rabbit anti Human RPS3 antibody recognizes RPS3, also known as 40S ribosomal protein S3 and IMR-90 ribosomal protein S3. Page 1 of 2 Ribosomes, the organelles that catalyze protein synthesis, consist of a small 40S subunit and a large 60S subunit. Together these subunits are composed of 4 RNA species and approximately 80 structurally distinct proteins. -

Ribosomal Protein Mutants and Their Effects on Plant Growth and Development
RIBOSOMAL PROTEIN MUTANTS AND THEIR EFFECTS ON PLANT GROWTH AND DEVELOPMENT A Thesis Submitted to the College of Graduate Studies and Research In Partial Fulfillment of the Requirements For the Degree of Doctor of Philosophy In the Department of Biology University of Saskatchewan Saskatoon By Chad Dale Stewart © Copyright Chad Stewart, November, 2012. All rights reserved. PERMISSION TO USE In presenting this thesis in partial fulfilment of the requirements for a Postgraduate degree from the University of Saskatchewan, I agree that the Libraries of this University may make it freely available for inspection. I further agree that permission for copying of this thesis in any manner, in whole or in part, for scholarly purposes may be granted by the professor or professors who supervised my thesis work or, in their absence, by the Head of the Department or the Dean of the College in which my thesis work was done. It is understood that any copying or publication or use of this thesis or parts thereof for financial gain shall not be allowed without my written permission. It is also understood that due recognition shall be given to me and to the University of Saskatchewan in any scholarly use which may be made of any material in my thesis. Requests for permission to copy or to make other use of material in this thesis in whole or part should be addressed to: Head of the Department of Biology University of Saskatchewan Saskatoon, Saskatchewan S7N 5E2 i ABSTRACT Ribosomes, large enzymatic complexes containing an RNA catalytic core, drive protein synthesis in all living organisms.