Ralgps2 Interacts with LST1 and Supports Tunneling Nanotubes Formation in Human Bladder Cancer Cells
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

LST1 Sirna (M): Sc-149136
SANTA CRUZ BIOTECHNOLOGY, INC. LST1 siRNA (m): sc-149136 BACKGROUND APPLICATIONS LST1 (leukocyte-specific transcript 1), also known as B144, is a 97 amino acid LST1 siRNA (m) is recommended for the inhibition of LST1 expression in protein single-pass membrane protein. LST1 may play a role in modulating mouse cells. immune responses, as well as dendritic cell maturation. LST1 has also been found to induce morphological changes, including microspikes and filopodia, SUPPORT REAGENTS when overexpressed in a variety of cell types. Localized to the endomembrane For optimal siRNA transfection efficiency, Santa Cruz Biotechnology’s system, LST1 is expressed as nine isoforms produced by alternative splicing. siRNA Transfection Reagent: sc-29528 (0.3 ml), siRNA Transfection Medium: Isoform 1, also designated LST1A, and isoform 2, also designated LST1C, sc-36868 (20 ml) and siRNA Dilution Buffer: sc-29527 (1.5 ml) are recom- γ have inhibitory effects on lymphocyte proliferation. Induced by IFN- , LST1 mended. Control siRNAs or Fluorescein Conjugated Control siRNAs are is expressed in adult lung, thymus, placenta, kidney and tonsil and fetal liver, available as 10 µM in 66 µl. Each contain a scrambled sequence that will spleen and brain. not lead to the specific degradation of any known cellular mRNA. Fluorescein Conjugated Control siRNAs include: sc-36869, sc-44239, sc-44240 and REFERENCES sc-44241. Control siRNAs include: sc-37007, sc-44230, sc-44231, sc-44232, 1. Neville, M.J. and Campbell, R.D. 1997. Alternative splicing of the LST1 gene sc-44233, sc-44234, sc-44235, sc-44236, sc-44237 and sc-44238. located in the major histocompatibility complex on human chromosome 6. -

Role of RUNX1 in Aberrant Retinal Angiogenesis Jonathan D
Page 1 of 25 Diabetes Identification of RUNX1 as a mediator of aberrant retinal angiogenesis Short Title: Role of RUNX1 in aberrant retinal angiogenesis Jonathan D. Lam,†1 Daniel J. Oh,†1 Lindsay L. Wong,1 Dhanesh Amarnani,1 Cindy Park- Windhol,1 Angie V. Sanchez,1 Jonathan Cardona-Velez,1,2 Declan McGuone,3 Anat O. Stemmer- Rachamimov,3 Dean Eliott,4 Diane R. Bielenberg,5 Tave van Zyl,4 Lishuang Shen,1 Xiaowu Gai,6 Patricia A. D’Amore*,1,7 Leo A. Kim*,1,4 Joseph F. Arboleda-Velasquez*1 Author affiliations: 1Schepens Eye Research Institute/Massachusetts Eye and Ear, Department of Ophthalmology, Harvard Medical School, 20 Staniford St., Boston, MA 02114 2Universidad Pontificia Bolivariana, Medellin, Colombia, #68- a, Cq. 1 #68305, Medellín, Antioquia, Colombia 3C.S. Kubik Laboratory for Neuropathology, Massachusetts General Hospital, 55 Fruit St., Boston, MA 02114 4Retina Service, Massachusetts Eye and Ear Infirmary, Department of Ophthalmology, Harvard Medical School, 243 Charles St., Boston, MA 02114 5Vascular Biology Program, Boston Children’s Hospital, Department of Surgery, Harvard Medical School, 300 Longwood Ave., Boston, MA 02115 6Center for Personalized Medicine, Children’s Hospital Los Angeles, Los Angeles, 4650 Sunset Blvd, Los Angeles, CA 90027, USA 7Department of Pathology, Harvard Medical School, 25 Shattuck St., Boston, MA 02115 Corresponding authors: Joseph F. Arboleda-Velasquez: [email protected] Ph: (617) 912-2517 Leo Kim: [email protected] Ph: (617) 912-2562 Patricia D’Amore: [email protected] Ph: (617) 912-2559 Fax: (617) 912-0128 20 Staniford St. Boston MA, 02114 † These authors contributed equally to this manuscript Word Count: 1905 Tables and Figures: 4 Diabetes Publish Ahead of Print, published online April 11, 2017 Diabetes Page 2 of 25 Abstract Proliferative diabetic retinopathy (PDR) is a common cause of blindness in the developed world’s working adult population, and affects those with type 1 and type 2 diabetes mellitus. -

B Inhibition in a Mouse Model of Chronic Colitis1
The Journal of Immunology Differential Expression of Inflammatory and Fibrogenic Genes and Their Regulation by NF-B Inhibition in a Mouse Model of Chronic Colitis1 Feng Wu and Shukti Chakravarti2 Fibrosis is a major complication of chronic inflammation, as seen in Crohn’s disease and ulcerative colitis, two forms of inflam- matory bowel diseases. To elucidate inflammatory signals that regulate fibrosis, we investigated gene expression changes under- lying chronic inflammation and fibrosis in trinitrobenzene sulfonic acid-induced murine colitis. Six weekly 2,4,6-trinitrobenzene sulfonic acid enemas were given to establish colitis and temporal gene expression patterns were obtained at 6-, 8-, 10-, and 12-wk time points. The 6-wk point, TNBS-w6, was the active, chronic inflammatory stage of the model marked by macrophage, neu- trophil, and CD3؉ and CD4؉ T cell infiltrates in the colon, consistent with the idea that this model is T cell immune response driven. Proinflammatory genes Cxcl1, Ccl2, Il1b, Lcn2, Pla2g2a, Saa3, S100a9, Nos2, Reg2, and Reg3g, and profibrogenic extra- cellular matrix genes Col1a1, Col1a2, Col3a1, and Lum (lumican), encoding a collagen-associated proteoglycan, were up-regulated at the active/chronic inflammatory stages. Rectal administration of the NF-B p65 antisense oligonucleotide reduced but did not abrogate inflammation and fibrosis completely. The antisense oligonucleotide treatment reduced total NF-B by 60% and down- regulated most proinflammatory genes. However, Ccl2, a proinflammatory chemokine known to promote fibrosis, was not down- regulated. Among extracellular matrix gene expressions Lum was suppressed while Col1a1 and Col3a1 were not. Thus, effective treatment of fibrosis in inflammatory bowel disease may require early and complete blockade of NF-B with particular attention to specific proinflammatory and profibrogenic genes that remain active at low levels of NF-B. -

Pan-Cancer Analysis of DNA Methylation Identifies Genes And
bioRxiv preprint doi: https://doi.org/10.1101/2021.06.20.449136; this version posted June 20, 2021. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY-ND 4.0 International license. Pan-Cancer Analysis of DNA Methylation Identifies Genes and Biological Functions Associated with Overall Survival Romola Cavet1, Peng Yue2, Guy Cavet*3 1The Nueva School, San Mateo, California, United States of America 2ReviR Therapeutics, Palo Alto, California, United States of America 3Paramune, Inc., Palo Alto, California, United States of America * Corresponding author E-Mail: [email protected] bioRxiv preprint doi: https://doi.org/10.1101/2021.06.20.449136; this version posted June 20, 2021. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY-ND 4.0 International license. Abstract DNA methylation influences gene expression and is altered in many cancers, but the relationship between DNA methylation and cancer outcomes is not yet fully understood. If methylation of specific genes is associated with better or worse outcomes, it could implicate genes in driving cancer and suggest therapeutic strategies. To advance our understanding of DNA methylation in cancer biology, we conducted a pan-cancer analysis of the relationship between methylation and overall survival. Using data on 28 tumor types from The Cancer Genome Atlas (TCGA), we identified genes and genomic regions whose methylation was recurrently associated with survival across multiple cancer types. -

Single-Cell Transcriptomes Reveal a Complex Cellular Landscape in the Middle Ear and Differential Capacities for Acute Response to Infection
fgene-11-00358 April 9, 2020 Time: 15:55 # 1 ORIGINAL RESEARCH published: 15 April 2020 doi: 10.3389/fgene.2020.00358 Single-Cell Transcriptomes Reveal a Complex Cellular Landscape in the Middle Ear and Differential Capacities for Acute Response to Infection Allen F. Ryan1*, Chanond A. Nasamran2, Kwang Pak1, Clara Draf1, Kathleen M. Fisch2, Nicholas Webster3 and Arwa Kurabi1 1 Departments of Surgery/Otolaryngology, UC San Diego School of Medicine, VA Medical Center, La Jolla, CA, United States, 2 Medicine/Center for Computational Biology & Bioinformatics, UC San Diego School of Medicine, VA Medical Center, La Jolla, CA, United States, 3 Medicine/Endocrinology, UC San Diego School of Medicine, VA Medical Center, La Jolla, CA, United States Single-cell transcriptomics was used to profile cells of the normal murine middle ear. Clustering analysis of 6770 transcriptomes identified 17 cell clusters corresponding to distinct cell types: five epithelial, three stromal, three lymphocyte, two monocyte, Edited by: two endothelial, one pericyte and one melanocyte cluster. Within some clusters, Amélie Bonnefond, Institut National de la Santé et de la cell subtypes were identified. While many corresponded to those cell types known Recherche Médicale (INSERM), from prior studies, several novel types or subtypes were noted. The results indicate France unexpected cellular diversity within the resting middle ear mucosa. The resolution of Reviewed by: Fabien Delahaye, uncomplicated, acute, otitis media is too rapid for cognate immunity to play a major Institut Pasteur de Lille, France role. Thus innate immunity is likely responsible for normal recovery from middle ear Nelson L. S. Tang, infection. The need for rapid response to pathogens suggests that innate immune The Chinese University of Hong Kong, China genes may be constitutively expressed by middle ear cells. -

ID AKI Vs Control Fold Change P Value Symbol Entrez Gene Name *In
ID AKI vs control P value Symbol Entrez Gene Name *In case of multiple probesets per gene, one with the highest fold change was selected. Fold Change 208083_s_at 7.88 0.000932 ITGB6 integrin, beta 6 202376_at 6.12 0.000518 SERPINA3 serpin peptidase inhibitor, clade A (alpha-1 antiproteinase, antitrypsin), member 3 1553575_at 5.62 0.0033 MT-ND6 NADH dehydrogenase, subunit 6 (complex I) 212768_s_at 5.50 0.000896 OLFM4 olfactomedin 4 206157_at 5.26 0.00177 PTX3 pentraxin 3, long 212531_at 4.26 0.00405 LCN2 lipocalin 2 215646_s_at 4.13 0.00408 VCAN versican 202018_s_at 4.12 0.0318 LTF lactotransferrin 203021_at 4.05 0.0129 SLPI secretory leukocyte peptidase inhibitor 222486_s_at 4.03 0.000329 ADAMTS1 ADAM metallopeptidase with thrombospondin type 1 motif, 1 1552439_s_at 3.82 0.000714 MEGF11 multiple EGF-like-domains 11 210602_s_at 3.74 0.000408 CDH6 cadherin 6, type 2, K-cadherin (fetal kidney) 229947_at 3.62 0.00843 PI15 peptidase inhibitor 15 204006_s_at 3.39 0.00241 FCGR3A Fc fragment of IgG, low affinity IIIa, receptor (CD16a) 202238_s_at 3.29 0.00492 NNMT nicotinamide N-methyltransferase 202917_s_at 3.20 0.00369 S100A8 S100 calcium binding protein A8 215223_s_at 3.17 0.000516 SOD2 superoxide dismutase 2, mitochondrial 204627_s_at 3.04 0.00619 ITGB3 integrin, beta 3 (platelet glycoprotein IIIa, antigen CD61) 223217_s_at 2.99 0.00397 NFKBIZ nuclear factor of kappa light polypeptide gene enhancer in B-cells inhibitor, zeta 231067_s_at 2.97 0.00681 AKAP12 A kinase (PRKA) anchor protein 12 224917_at 2.94 0.00256 VMP1/ mir-21likely ortholog -

Functional Genomic Annotation of Genetic Risk Loci Highlights Inflammation and Epithelial Biology Networks in CKD
BASIC RESEARCH www.jasn.org Functional Genomic Annotation of Genetic Risk Loci Highlights Inflammation and Epithelial Biology Networks in CKD Nora Ledo, Yi-An Ko, Ae-Seo Deok Park, Hyun-Mi Kang, Sang-Youb Han, Peter Choi, and Katalin Susztak Renal Electrolyte and Hypertension Division, Perelman School of Medicine, University of Pennsylvania, Philadelphia, Pennsylvania ABSTRACT Genome-wide association studies (GWASs) have identified multiple loci associated with the risk of CKD. Almost all risk variants are localized to the noncoding region of the genome; therefore, the role of these variants in CKD development is largely unknown. We hypothesized that polymorphisms alter transcription factor binding, thereby influencing the expression of nearby genes. Here, we examined the regulation of transcripts in the vicinity of CKD-associated polymorphisms in control and diseased human kidney samples and used systems biology approaches to identify potentially causal genes for prioritization. We interro- gated the expression and regulation of 226 transcripts in the vicinity of 44 single nucleotide polymorphisms using RNA sequencing and gene expression arrays from 95 microdissected control and diseased tubule samples and 51 glomerular samples. Gene expression analysis from 41 tubule samples served for external validation. 92 transcripts in the tubule compartment and 34 transcripts in glomeruli showed statistically significant correlation with eGFR. Many novel genes, including ACSM2A/2B, FAM47E, and PLXDC1, were identified. We observed that the expression of multiple genes in the vicinity of any single CKD risk allele correlated with renal function, potentially indicating that genetic variants influence multiple transcripts. Network analysis of GFR-correlating transcripts highlighted two major clusters; a positive correlation with epithelial and vascular functions and an inverse correlation with inflammatory gene cluster. -

Six Microsatellite Markers on the Short Arm of Chromosome 6 in Relation to HLA-DR3 and TNF−308A in Systemic Lupus Erythematosus
Genes and Immunity (2001) 2, 373–380 2001 Nature Publishing Group All rights reserved 1466-4879/01 $15.00 www.nature.com/gene Six microsatellite markers on the short arm of chromosome 6 in relation to HLA-DR3 and TNF−308A in systemic lupus erythematosus MW van der Linden1,2, AR van der Slik3, E Zanelli3, MJ Giphart3, E Pieterman2, GMTh Schreuder3, RGJ Westendorp4 and TWJ Huizinga2 Departments of 1Clinical Epidemiology, 2Rheumatology, 3Immunohaematology and Blood Transfusion, 4General Internal Medicine, Leiden University Medical Center, PO Box 9600, 2300 RC Leiden, The Netherlands Differences in allelic distribution at loci surrounding the human HLA-DRB1 and tumor necrosis factor (TNF) genes have been observed in association with systemic lupus erythematosus (SLE). We investigated whether the association of HLA- DRB1*0301 (HLA-DR3) and TNF−308A with SLE could be attributed to polymorphic markers in the chromosomal region encompassed by HLA-DRB1 and HLA-C. Ninety-one consecutive Caucasian patients with SLE and 253 controls (organ donors) were typed for HLA-DRB1, microsatellites D6S1014, D6S273, TNFa, MIB, C1F2F5, and C1F3F2 and the single nucleotide polymorphism at position −308 in the promoter of TNF. The independent contribution of alleles to disease susceptibility was estimated by cross-tabulation and multivariate logistic regression. Possession of TNF−308A was associated with susceptibility to SLE (odds ratio [95% confidence interval], 3.70 [2.24–6.11]). This remained present after stratification on possession of HLA-DR3 (pooled odds ratio, 2.53 [1.37–4.70]). Stratification revealed a possible association of possession of C1F2F5*192 with protection from SLE beyond the effects of HLA-DR3 and TNF−308A. -

Extended LTA, TNF, LST1 and HLA Gene Haplotypes and Their Association with Rubella Vaccine-Induced Immunity
Extended LTA, TNF, LST1 and HLA Gene Haplotypes and Their Association with Rubella Vaccine-Induced Immunity Inna G. Ovsyannikova1,4*, Robert A. Vierkant3, V. Shane Pankratz3, Robert M. Jacobson1,2, Gregory A. Poland1,2,4* 1 Vaccine Research Group, Mayo Clinic, Rochester, Minnesota, United States of America, 2 Department of Pediatric and Adolescent Medicine, Mayo Clinic, Rochester, Minnesota, United States of America, 3 Department of Health Sciences Research, Mayo Clinic, Rochester, Minnesota, United States of America, 4 Program in Translational Immunovirology and Biodefense, Mayo Clinic, Rochester, Minnesota, United States of America Abstract Background: Recent studies have suggested the importance of HLA genes in determining immune responses following rubella vaccine. The telomeric class III region of the HLA complex harbors several genes, including lymphotoxin alpha (LTA), tumor necrosis factor (TNF) and leukocyte specific transcript -1 (LST1) genes, located between the class I B and class II DRB1 loci. Apart from HLA, little is known about the effect of this extended genetic region on HLA haplotypic backgrounds as applied to immune responses. Methodology/Principal Findings: We examined the association between immune responses and extended class I-class II- class III haplotypes among 714 healthy children after two doses of rubella vaccination. These extended haplotypes were then compared to the HLA-only haplotypes. The most significant association was observed between haplotypes extending across the HLA class I region, ten-SNP haplotypes, and the HLA class II region (i.e. A-C-B-LTA-TNF-LST1-DRB1-DQA1-DQB1- DPA1-DPB1) and rubella-specific antibodies (global p-value of 0.03). Associations were found between both extended A*02- C*03-B*15-AAAACGGGGC-DRB1*04-DQA1*03-DQB1*03-DPA1*01-DPB1*04 (p = 0.002) and HLA-only A*02-C*03-B*15- DRB1*04-DQA1*03-DQB1*03-DPA1*01-DPB1*04 haplotypes (p = 0.009) and higher levels of rubella antibodies. -
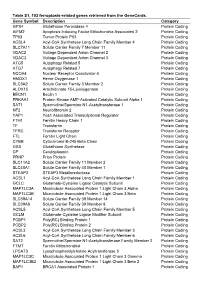
Table S1. 103 Ferroptosis-Related Genes Retrieved from the Genecards
Table S1. 103 ferroptosis-related genes retrieved from the GeneCards. Gene Symbol Description Category GPX4 Glutathione Peroxidase 4 Protein Coding AIFM2 Apoptosis Inducing Factor Mitochondria Associated 2 Protein Coding TP53 Tumor Protein P53 Protein Coding ACSL4 Acyl-CoA Synthetase Long Chain Family Member 4 Protein Coding SLC7A11 Solute Carrier Family 7 Member 11 Protein Coding VDAC2 Voltage Dependent Anion Channel 2 Protein Coding VDAC3 Voltage Dependent Anion Channel 3 Protein Coding ATG5 Autophagy Related 5 Protein Coding ATG7 Autophagy Related 7 Protein Coding NCOA4 Nuclear Receptor Coactivator 4 Protein Coding HMOX1 Heme Oxygenase 1 Protein Coding SLC3A2 Solute Carrier Family 3 Member 2 Protein Coding ALOX15 Arachidonate 15-Lipoxygenase Protein Coding BECN1 Beclin 1 Protein Coding PRKAA1 Protein Kinase AMP-Activated Catalytic Subunit Alpha 1 Protein Coding SAT1 Spermidine/Spermine N1-Acetyltransferase 1 Protein Coding NF2 Neurofibromin 2 Protein Coding YAP1 Yes1 Associated Transcriptional Regulator Protein Coding FTH1 Ferritin Heavy Chain 1 Protein Coding TF Transferrin Protein Coding TFRC Transferrin Receptor Protein Coding FTL Ferritin Light Chain Protein Coding CYBB Cytochrome B-245 Beta Chain Protein Coding GSS Glutathione Synthetase Protein Coding CP Ceruloplasmin Protein Coding PRNP Prion Protein Protein Coding SLC11A2 Solute Carrier Family 11 Member 2 Protein Coding SLC40A1 Solute Carrier Family 40 Member 1 Protein Coding STEAP3 STEAP3 Metalloreductase Protein Coding ACSL1 Acyl-CoA Synthetase Long Chain Family Member 1 Protein -

Splicing and Immunomodulatory Function : a Gene with Extensive
LST1: A Gene with Extensive Alternative Splicing and Immunomodulatory Function Ingrid Rollinger-Holzinger, Brigitte Eibl, Marc Pauly, Ute Griesser, François Hentges, Bernhard Auer, Georg Pall, This information is current as Peter Schratzberger, Dietger Niederwieser, Elisabeth H. of September 28, 2021. Weiss and Heinz Zwierzina J Immunol 2000; 164:3169-3176; ; doi: 10.4049/jimmunol.164.6.3169 http://www.jimmunol.org/content/164/6/3169 Downloaded from References This article cites 21 articles, 6 of which you can access for free at: http://www.jimmunol.org/content/164/6/3169.full#ref-list-1 http://www.jimmunol.org/ Why The JI? Submit online. • Rapid Reviews! 30 days* from submission to initial decision • No Triage! Every submission reviewed by practicing scientists • Fast Publication! 4 weeks from acceptance to publication by guest on September 28, 2021 *average Subscription Information about subscribing to The Journal of Immunology is online at: http://jimmunol.org/subscription Permissions Submit copyright permission requests at: http://www.aai.org/About/Publications/JI/copyright.html Email Alerts Receive free email-alerts when new articles cite this article. Sign up at: http://jimmunol.org/alerts The Journal of Immunology is published twice each month by The American Association of Immunologists, Inc., 1451 Rockville Pike, Suite 650, Rockville, MD 20852 Copyright © 2000 by The American Association of Immunologists All rights reserved. Print ISSN: 0022-1767 Online ISSN: 1550-6606. LST1: A Gene with Extensive Alternative Splicing and Immunomodulatory Function1 2 † † ‡ Ingrid Rollinger-Holzinger, * Brigitte Eibl, Marc Pauly*, Ute Griesser, Franc¸ois Hentges, Bernhard Auer,§ Georg Pall,† Peter Schratzberger,† Dietger Niederwieser,¶ Elisabeth H. -

(12) United States Patent (10) Patent No.: US 8,092,998 B2 Stuhlmiller Et Al
USO08092998B2 (12) United States Patent (10) Patent No.: US 8,092,998 B2 Stuhlmiller et al. (45) Date of Patent: Jan. 10, 2012 (54) BOMARKERS PREDCTIVE OF THE 2007/0172475 A1 7/2007 Matheus et al. RESPONSIVENESS TO TNFO INHIBITORS IN 2007/0172897 A1 7/2007 Maksymowych et al. 2007. O184045 A1 8, 2007 Doctor et al. AUTOIMMUNE DISORDERS 2007/0202051 A1 8/2007 Schusching 2007/0202104 A1 8/2007 Banerjee et al. (75) Inventors: Bruno Stuhlmiller, Berlin (DE): 2007/0237831 A1 10/2007 Sung et al. 2007,0269.463 A1 11/2007 Donavan Gerd-Reudiger Burmester, Berlin (DE) 2007,0292442 A1 12/2007 Wan et al. 2008.0118496 A1 5/2008 Medich et al. (73) Assignee: Abbott Laboratories, Abbott Park, IL 2008. O1313.74 A1 6/2008 Medich et al. (US) 2008. O166348 A1 7/2008 Kupper et al. 2008.0193466 A1 8/2008 Banerjee et al. 2008/0227136 A1 9, 2008 Pla et al. (*) Notice: Subject to any disclaimer, the term of this 2008/0311043 A1 12/2008 Hoffman et al. patent is extended or adjusted under 35 2009/0028794 A1 1/2009 Medich et al. U.S.C. 154(b) by 5 days. 2009/0110679 A1 4/2009 Li et al. 2009/O123378 A1 5/2009 Wong et al. (21) Appl. No.: 12/130,373 2009, O1485.13 A1 6/2009 Fraunhofer et al. (Continued) (22) Filed: May 30, 2008 FOREIGN PATENT DOCUMENTS (65) Prior Publication Data EP 1795610 A1 * 6, 2007 US 2009/OO17472 A1 Jan. 15, 2009 (Continued) Related U.S. Application Data OTHER PUBLICATIONS (60) Provisional application No.