Erik Halcsik
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Supplemental Information to Mammadova-Bach Et Al., “Laminin Α1 Orchestrates VEGFA Functions in the Ecosystem of Colorectal Carcinogenesis”
Supplemental information to Mammadova-Bach et al., “Laminin α1 orchestrates VEGFA functions in the ecosystem of colorectal carcinogenesis” Supplemental material and methods Cloning of the villin-LMα1 vector The plasmid pBS-villin-promoter containing the 3.5 Kb of the murine villin promoter, the first non coding exon, 5.5 kb of the first intron and 15 nucleotides of the second villin exon, was generated by S. Robine (Institut Curie, Paris, France). The EcoRI site in the multi cloning site was destroyed by fill in ligation with T4 polymerase according to the manufacturer`s instructions (New England Biolabs, Ozyme, Saint Quentin en Yvelines, France). Site directed mutagenesis (GeneEditor in vitro Site-Directed Mutagenesis system, Promega, Charbonnières-les-Bains, France) was then used to introduce a BsiWI site before the start codon of the villin coding sequence using the 5’ phosphorylated primer: 5’CCTTCTCCTCTAGGCTCGCGTACGATGACGTCGGACTTGCGG3’. A double strand annealed oligonucleotide, 5’GGCCGGACGCGTGAATTCGTCGACGC3’ and 5’GGCCGCGTCGACGAATTCACGC GTCC3’ containing restriction site for MluI, EcoRI and SalI were inserted in the NotI site (present in the multi cloning site), generating the plasmid pBS-villin-promoter-MES. The SV40 polyA region of the pEGFP plasmid (Clontech, Ozyme, Saint Quentin Yvelines, France) was amplified by PCR using primers 5’GGCGCCTCTAGATCATAATCAGCCATA3’ and 5’GGCGCCCTTAAGATACATTGATGAGTT3’ before subcloning into the pGEMTeasy vector (Promega, Charbonnières-les-Bains, France). After EcoRI digestion, the SV40 polyA fragment was purified with the NucleoSpin Extract II kit (Machery-Nagel, Hoerdt, France) and then subcloned into the EcoRI site of the plasmid pBS-villin-promoter-MES. Site directed mutagenesis was used to introduce a BsiWI site (5’ phosphorylated AGCGCAGGGAGCGGCGGCCGTACGATGCGCGGCAGCGGCACG3’) before the initiation codon and a MluI site (5’ phosphorylated 1 CCCGGGCCTGAGCCCTAAACGCGTGCCAGCCTCTGCCCTTGG3’) after the stop codon in the full length cDNA coding for the mouse LMα1 in the pCIS vector (kindly provided by P. -

Gene Symbol Gene Description ACVR1B Activin a Receptor, Type IB
Table S1. Kinase clones included in human kinase cDNA library for yeast two-hybrid screening Gene Symbol Gene Description ACVR1B activin A receptor, type IB ADCK2 aarF domain containing kinase 2 ADCK4 aarF domain containing kinase 4 AGK multiple substrate lipid kinase;MULK AK1 adenylate kinase 1 AK3 adenylate kinase 3 like 1 AK3L1 adenylate kinase 3 ALDH18A1 aldehyde dehydrogenase 18 family, member A1;ALDH18A1 ALK anaplastic lymphoma kinase (Ki-1) ALPK1 alpha-kinase 1 ALPK2 alpha-kinase 2 AMHR2 anti-Mullerian hormone receptor, type II ARAF v-raf murine sarcoma 3611 viral oncogene homolog 1 ARSG arylsulfatase G;ARSG AURKB aurora kinase B AURKC aurora kinase C BCKDK branched chain alpha-ketoacid dehydrogenase kinase BMPR1A bone morphogenetic protein receptor, type IA BMPR2 bone morphogenetic protein receptor, type II (serine/threonine kinase) BRAF v-raf murine sarcoma viral oncogene homolog B1 BRD3 bromodomain containing 3 BRD4 bromodomain containing 4 BTK Bruton agammaglobulinemia tyrosine kinase BUB1 BUB1 budding uninhibited by benzimidazoles 1 homolog (yeast) BUB1B BUB1 budding uninhibited by benzimidazoles 1 homolog beta (yeast) C9orf98 chromosome 9 open reading frame 98;C9orf98 CABC1 chaperone, ABC1 activity of bc1 complex like (S. pombe) CALM1 calmodulin 1 (phosphorylase kinase, delta) CALM2 calmodulin 2 (phosphorylase kinase, delta) CALM3 calmodulin 3 (phosphorylase kinase, delta) CAMK1 calcium/calmodulin-dependent protein kinase I CAMK2A calcium/calmodulin-dependent protein kinase (CaM kinase) II alpha CAMK2B calcium/calmodulin-dependent -

MAPK12 Antibody Order 021-34695924 [email protected] Support 400-6123-828 50Ul [email protected] 100 Ul √ √ Web
TD2359 MAPK12 Antibody Order 021-34695924 [email protected] Support 400-6123-828 50ul [email protected] 100 uL √ √ Web www.ab-mart.com.cn Description: Serine/threonine kinase which acts as an essential component of the MAP kinase signal transduction pathway. MAPK12 is one of the four p38 MAPKs which play an important role in the cascades of cellular responses evoked by extracellular stimuli such as proinflammatory cytokines or physical stress leading to direct activation of transcription factors such as ELK1 and ATF2. Accordingly, p38 MAPKs phosphorylate a broad range of proteins and it has been estimated that they may have approximately 200 to 300 substrates each. Some of the targets are downstream kinases such as MAPKAPK2, which are activated through phosphorylation and further phosphorylate additional targets. Plays a role in myoblast differentiation and also in the down-regulation of cyclin D1 in response to hypoxia in adrenal cells suggesting MAPK12 may inhibit cell proliferation while promoting differentiation. Phosphorylates DLG1. Following osmotic shock, MAPK12 in the cell nucleus increases its association with nuclear DLG1, thereby causing dissociation of DLG1-SFPQ complexes. This function is independent of its catalytic activity and could affect mRNA processing and/or gene transcription to aid cell adaptation to osmolarity changes in the environment. Regulates UV-induced checkpoint signaling and repair of UV-induced DNA damage and G2 arrest after gamma-radiation exposure. MAPK12 is involved in the regulation of SLC2A1 expression and basal glucose uptake in L6 myotubes; and negatively regulates SLC2A4 expression and contraction-mediated glucose uptake in adult skeletal muscle. -

Human MAPK10 Peptide (DAG-P1762) This Product Is for Research Use Only and Is Not Intended for Diagnostic Use
Human MAPK10 peptide (DAG-P1762) This product is for research use only and is not intended for diagnostic use. PRODUCT INFORMATION Antigen Description The protein encoded by this gene is a member of the MAP kinase family. MAP kinases act as an integration point for multiple biochemical signals, and are involved in a wide variety of cellular processes such as proliferation, differentiation, transcription regulation and development. This protein is a neuronal-specific form of c-Jun N-terminal kinases (JNKs). Through its phosphorylation and nuclear localization, this kinase plays regulatory roles in the signaling pathways during neuronal apoptosis. Beta-arrestin 2, a receptor-regulated MAP kinase scaffold protein, is found to interact with, and stimulate the phosphorylation of this kinase by MAP kinase kinase 4 (MKK4). Cyclin-dependent kianse 5 can phosphorylate, and inhibit the activity of this kinase, which may be important in preventing neuronal apoptosis. Four alternatively spliced transcript variants encoding distinct isoforms have been reported. [provided by RefSeq, Jul 2008] Specificity Specific to a subset of neurons in the nervous system. Present in the hippocampus and areas, cerebellum, striatum, brain stem, and weakly in the spinal cord. Very weak expression in testis and kidney. Nature Synthetic Expression System N/A Purity 70 - 90% by HPLC. Conjugate Unconjugated Sequence Similarities Belongs to the protein kinase superfamily. CMGC Ser/Thr protein kinase family. MAP kinase subfamily.Contains 1 protein kinase domain. Cellular Localization Cytoplasm. Procedure None Format Liquid Preservative None Storage Shipped at 4°C. Upon delivery aliquot and store at -20°C or -80°C. Avoid repeated freeze / thaw cycles. -

Anti-SEK1 / MKK4 Phospho (Ser80) Antibody (ARG51673)
Product datasheet [email protected] ARG51673 Package: 100 μl, 50 μl anti-SEK1 / MKK4 phospho (Ser80) antibody Store at: -20°C Summary Product Description Rabbit Polyclonal antibody recognizes SEK1 / MKK4 phospho (Ser80) Tested Reactivity Hu, Ms, Rat Tested Application ICC/IF, IHC-P, WB Host Rabbit Clonality Polyclonal Isotype IgG Target Name SEK1 / MKK4 Antigen Species Human Immunogen Peptide sequence around phosphorylation site of serine 80 (T-H-S(p)-I-E) derived from Human SEK1/MKK4. Conjugation Un-conjugated Alternate Names MEK 4; MAPK/ERK kinase 4; PRKMK4; SAPKK-1; SAPK/ERK kinase 1; SKK1; JNK-activating kinase 1; EC 2.7.12.2; MEK4; MAP kinase kinase 4; c-Jun N-terminal kinase kinase 1; SEK1; SAPKK1; MAPKK4; Stress- activated protein kinase kinase 1; JNKK1; MKK4; SERK1; SAPK kinase 1; Dual specificity mitogen- activated protein kinase kinase 4; JNKK; MAPKK 4 Application Instructions Application table Application Dilution ICC/IF 1:100 - 1:200 IHC-P 1:50 - 1:100 WB 1:500 - 1:1000 Application Note * The dilutions indicate recommended starting dilutions and the optimal dilutions or concentrations should be determined by the scientist. Calculated Mw 44 kDa Properties Form Liquid Purification Antibodies were produced by immunizing rabbits with KLH-conjugated synthetic phosphopeptide. Antibodies were purified by affinity-chromatography using epitope-specific phosphopeptide. In addition, non-phospho specific antibodies were removed by chromatogramphy using non- phosphopeptide. Buffer PBS (without Mg2+ and Ca2+, pH 7.4), 150mM NaCl, 0.02% Sodium azide and 50% Glycerol. Preservative 0.02% Sodium azide Stabilizer 50% Glycerol www.arigobio.com 1/3 Concentration 1 mg/ml Storage instruction For continuous use, store undiluted antibody at 2-8°C for up to a week. -

Genetics of Familial Non-Medullary Thyroid Carcinoma (FNMTC)
cancers Review Genetics of Familial Non-Medullary Thyroid Carcinoma (FNMTC) Chiara Diquigiovanni * and Elena Bonora Unit of Medical Genetics, Department of Medical and Surgical Sciences, University of Bologna, 40138 Bologna, Italy; [email protected] * Correspondence: [email protected]; Tel.: +39-051-208-8418 Simple Summary: Non-medullary thyroid carcinoma (NMTC) originates from thyroid follicular epithelial cells and is considered familial when occurs in two or more first-degree relatives of the patient, in the absence of predisposing environmental factors. Familial NMTC (FNMTC) cases show a high genetic heterogeneity, thus impairing the identification of pivotal molecular changes. In the past years, linkage-based approaches identified several susceptibility loci and variants associated with NMTC risk, however only few genes have been identified. The advent of next-generation sequencing technologies has improved the discovery of new predisposing genes. In this review we report the most significant genes where variants predispose to FNMTC, with the perspective that the integration of these new molecular findings in the clinical data of patients might allow an early detection and tailored therapy of the disease, optimizing patient management. Abstract: Non-medullary thyroid carcinoma (NMTC) is the most frequent endocrine tumor and originates from the follicular epithelial cells of the thyroid. Familial NMTC (FNMTC) has been defined in pedigrees where two or more first-degree relatives of the patient present the disease in absence of other predisposing environmental factors. Compared to sporadic cases, FNMTCs are often multifocal, recurring more frequently and showing an early age at onset with a worse outcome. FNMTC cases Citation: Diquigiovanni, C.; Bonora, E. -

Circrna LRIG3 Knockdown Inhibits Hepatocellular Carcinoma Progression by Regulating Mir-223-3P and MAPK/ERK Pathway
CircRNA LRIG3 knockdown inhibits hepatocellular carcinoma progression by regulating miR-223-3p and MAPK/ERK pathway Type Research paper Keywords hepatocellular carcinoma, miR-223-3p, circ_LRIG3, MAP2K6, MAPK/ERK pathway Abstract Introduction Emerging evidence suggests that circular RNAs (circRNAs) play critical roles in tumorigenesis. However, the roles and molecular mechanisms of circRNA leucine-rich repeat immunoglobulin domain-containing protein 3 (circ_LRIG3) in hepatocellular carcinoma (HCC) has not been investigated. Material and methods The expression levels of circ_LRIG3, miR-223-3p, and mitogen-activated protein kinase kinase 6 (MAP2K6) were determined by qRT-PCR. Flow cytometry was applied to determine the cell cycle distribution and apoptosis. Cell proliferation, migration and invasion were assessed by MTT, colony formation, and transwell assays. Western blot assay was employed to measure the protein levels of the snail, E-cadherin, MAP2K6, mitogen-activated protein kinase (MAPK), phospho-MAPK (p- MAPK), extracellular signal-regulated kinases (ERKs), and phospho-ERKs (p- ERKs). The relationship between miR-223-3p and circ_LRIG3 or MAP2K6 was predicted by bioinformatics tools and verified by dual-luciferase reporter assay. A xenograft tumor model was established to confirm the functions of circ_LRIG3 in vivo. Results Preprint Circ_LRIG3 and MAP2K6 expression were enhanced while miR-223-3p abundance was reduced in HCC tissues and cells. Knockdown of circ_LRIG3 inhibited cell proliferation, metastasis, and increasing apoptosis. MiR-223-3p was a target of circ_LRIG3, and its downregulation reversed the inhibitory effect of circ_LRIG3 knockdown on the progression of HCC cells. Moreover, MAP2K6 could bind to miR-223-3p, and MAP2K6 upregulation also abolished the suppressive impact of circ_LRIG3 interference on progression of HCC cells. -

Multi-Modal Meta-Analysis of 1494 Hepatocellular Carcinoma Samples Reveals
Author Manuscript Published OnlineFirst on September 21, 2018; DOI: 10.1158/1078-0432.CCR-18-0088 Author manuscripts have been peer reviewed and accepted for publication but have not yet been edited. Multi-modal meta-analysis of 1494 hepatocellular carcinoma samples reveals significant impact of consensus driver genes on phenotypes Kumardeep Chaudhary1, Olivier B Poirion1, Liangqun Lu1,2, Sijia Huang1,2, Travers Ching1,2, Lana X Garmire1,2,3* 1Epidemiology Program, University of Hawaii Cancer Center, Honolulu, HI 96813, USA 2Molecular Biosciences and Bioengineering Graduate Program, University of Hawaii at Manoa, Honolulu, HI 96822, USA 3Current affiliation: Department of Computational Medicine and Bioinformatics, Building 520, 1600 Huron Parkway, Ann Arbor, MI 48109 Short Title: Impact of consensus driver genes in hepatocellular carcinoma * To whom correspondence should be addressed. Lana X. Garmire, Department of Computational Medicine and Bioinformatics Medical School, University of Michigan Building 520, 1600 Huron Parkway Ann Arbor-48109, MI, USA, Phone: +1-(734) 615-5510 Current email address: [email protected] Grant Support: This research was supported by grants K01ES025434 awarded by NIEHS through funds provided by the trans-NIH Big Data to Knowledge (BD2K) initiative (http://datascience.nih.gov/bd2k), P20 COBRE GM103457 awarded by NIH/NIGMS, NICHD R01 HD084633 and NLM R01LM012373 and Hawaii Community Foundation Medical Research Grant 14ADVC-64566 to Lana X Garmire. 1 Downloaded from clincancerres.aacrjournals.org on October 1, 2021. © 2018 American Association for Cancer Research. Author Manuscript Published OnlineFirst on September 21, 2018; DOI: 10.1158/1078-0432.CCR-18-0088 Author manuscripts have been peer reviewed and accepted for publication but have not yet been edited. -
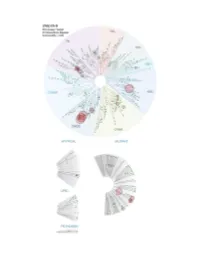
Profiling Data
Compound Name DiscoveRx Gene Symbol Entrez Gene Percent Compound Symbol Control Concentration (nM) JNK-IN-8 AAK1 AAK1 69 1000 JNK-IN-8 ABL1(E255K)-phosphorylated ABL1 100 1000 JNK-IN-8 ABL1(F317I)-nonphosphorylated ABL1 87 1000 JNK-IN-8 ABL1(F317I)-phosphorylated ABL1 100 1000 JNK-IN-8 ABL1(F317L)-nonphosphorylated ABL1 65 1000 JNK-IN-8 ABL1(F317L)-phosphorylated ABL1 61 1000 JNK-IN-8 ABL1(H396P)-nonphosphorylated ABL1 42 1000 JNK-IN-8 ABL1(H396P)-phosphorylated ABL1 60 1000 JNK-IN-8 ABL1(M351T)-phosphorylated ABL1 81 1000 JNK-IN-8 ABL1(Q252H)-nonphosphorylated ABL1 100 1000 JNK-IN-8 ABL1(Q252H)-phosphorylated ABL1 56 1000 JNK-IN-8 ABL1(T315I)-nonphosphorylated ABL1 100 1000 JNK-IN-8 ABL1(T315I)-phosphorylated ABL1 92 1000 JNK-IN-8 ABL1(Y253F)-phosphorylated ABL1 71 1000 JNK-IN-8 ABL1-nonphosphorylated ABL1 97 1000 JNK-IN-8 ABL1-phosphorylated ABL1 100 1000 JNK-IN-8 ABL2 ABL2 97 1000 JNK-IN-8 ACVR1 ACVR1 100 1000 JNK-IN-8 ACVR1B ACVR1B 88 1000 JNK-IN-8 ACVR2A ACVR2A 100 1000 JNK-IN-8 ACVR2B ACVR2B 100 1000 JNK-IN-8 ACVRL1 ACVRL1 96 1000 JNK-IN-8 ADCK3 CABC1 100 1000 JNK-IN-8 ADCK4 ADCK4 93 1000 JNK-IN-8 AKT1 AKT1 100 1000 JNK-IN-8 AKT2 AKT2 100 1000 JNK-IN-8 AKT3 AKT3 100 1000 JNK-IN-8 ALK ALK 85 1000 JNK-IN-8 AMPK-alpha1 PRKAA1 100 1000 JNK-IN-8 AMPK-alpha2 PRKAA2 84 1000 JNK-IN-8 ANKK1 ANKK1 75 1000 JNK-IN-8 ARK5 NUAK1 100 1000 JNK-IN-8 ASK1 MAP3K5 100 1000 JNK-IN-8 ASK2 MAP3K6 93 1000 JNK-IN-8 AURKA AURKA 100 1000 JNK-IN-8 AURKA AURKA 84 1000 JNK-IN-8 AURKB AURKB 83 1000 JNK-IN-8 AURKB AURKB 96 1000 JNK-IN-8 AURKC AURKC 95 1000 JNK-IN-8 -
HCC and Cancer Mutated Genes Summarized in the Literature Gene Symbol Gene Name References*
HCC and cancer mutated genes summarized in the literature Gene symbol Gene name References* A2M Alpha-2-macroglobulin (4) ABL1 c-abl oncogene 1, receptor tyrosine kinase (4,5,22) ACBD7 Acyl-Coenzyme A binding domain containing 7 (23) ACTL6A Actin-like 6A (4,5) ACTL6B Actin-like 6B (4) ACVR1B Activin A receptor, type IB (21,22) ACVR2A Activin A receptor, type IIA (4,21) ADAM10 ADAM metallopeptidase domain 10 (5) ADAMTS9 ADAM metallopeptidase with thrombospondin type 1 motif, 9 (4) ADCY2 Adenylate cyclase 2 (brain) (26) AJUBA Ajuba LIM protein (21) AKAP9 A kinase (PRKA) anchor protein (yotiao) 9 (4) Akt AKT serine/threonine kinase (28) AKT1 v-akt murine thymoma viral oncogene homolog 1 (5,21,22) AKT2 v-akt murine thymoma viral oncogene homolog 2 (4) ALB Albumin (4) ALK Anaplastic lymphoma receptor tyrosine kinase (22) AMPH Amphiphysin (24) ANK3 Ankyrin 3, node of Ranvier (ankyrin G) (4) ANKRD12 Ankyrin repeat domain 12 (4) ANO1 Anoctamin 1, calcium activated chloride channel (4) APC Adenomatous polyposis coli (4,5,21,22,25,28) APOB Apolipoprotein B [including Ag(x) antigen] (4) AR Androgen receptor (5,21-23) ARAP1 ArfGAP with RhoGAP domain, ankyrin repeat and PH domain 1 (4) ARHGAP35 Rho GTPase activating protein 35 (21) ARID1A AT rich interactive domain 1A (SWI-like) (4,5,21,22,24,25,27,28) ARID1B AT rich interactive domain 1B (SWI1-like) (4,5,22) ARID2 AT rich interactive domain 2 (ARID, RFX-like) (4,5,22,24,25,27,28) ARID4A AT rich interactive domain 4A (RBP1-like) (28) ARID5B AT rich interactive domain 5B (MRF1-like) (21) ASPM Asp (abnormal -

ACVR2B Recombinant Protein Cat
ACVR2B Recombinant Protein Cat. No.: 96-009 ACVR2B Recombinant Protein Specifications SPECIES: Human SOURCE SPECIES: HEK293 cells SEQUENCE: Ser 19 - Thr 137 FUSION TAG: C-His Tag TESTED APPLICATIONS: WB APPLICATIONS: This recombinant protein can be used for WB. For research use only. PREDICTED MOLECULAR 14.5 kDa WEIGHT: Properties PURITY: >97% as determined by SDS-PAGE. PHYSICAL STATE: Lyophilized BUFFER: PBS, pH7.4 Lyophilized Protein should be stored at -20˚C or lower for long term storage. Upon STORAGE CONDITIONS: reconstitution, working aliquots should be stored at -20˚C or -70˚C. Avoid repeated freeze-thaw cycles. September 27, 2021 1 https://www.prosci-inc.com/acvr2b-recombinant-protein-96-009.html Additional Info OFFICIAL SYMBOL: ACVR2B ALTERNATE NAMES: ACVR2B, ACTRIIB, MGC116908 ACCESSION NO.: NP_001097 GENE ID: 93 Background and References Activin receptor type-2B (ACVR2B) is also known as ActR-IIB and MGC116908, ACVR2B is an activin type 2 receptor. Activins are dimeric growth and differentiation factors which belong to the transforming growth factor-beta (TGF-beta) superfamily of structurally related signaling proteins. Activins signal through a heteromeric complex of receptor serine kinases which include at least two type I (I and IB) and two type II (II and IIB) receptors. These receptors are all transmembrane proteins, composed of a ligand-binding extracellular domain with cysteine-rich region, a transmembrane domain, and a BACKGROUND: cytoplasmic domain with predicted serine/threonine specificity. Type I receptors are essential for signaling; and type II receptors are required for binding ligands and for expression of type I receptors. Type I and II receptors form a stable complex after ligand binding, resulting in phosphorylation of type I receptors by type II receptors. -

PRKACA Mediates Resistance to HER2-Targeted Therapy in Breast Cancer Cells and Restores Anti-Apoptotic Signaling
Oncogene (2015) 34, 2061–2071 © 2015 Macmillan Publishers Limited All rights reserved 0950-9232/15 www.nature.com/onc ORIGINAL ARTICLE PRKACA mediates resistance to HER2-targeted therapy in breast cancer cells and restores anti-apoptotic signaling SE Moody1,2,3, AC Schinzel1, S Singh1, F Izzo1, MR Strickland1, L Luo1,2, SR Thomas3, JS Boehm3, SY Kim4, ZC Wang5,6 and WC Hahn1,2,3 Targeting HER2 with antibodies or small molecule inhibitors in HER2-positive breast cancer leads to improved survival, but resistance is a common clinical problem. To uncover novel mechanisms of resistance to anti-HER2 therapy in breast cancer, we performed a kinase open reading frame screen to identify genes that rescue HER2-amplified breast cancer cells from HER2 inhibition or suppression. In addition to multiple members of the MAPK (mitogen-activated protein kinase) and PI3K (phosphoinositide 3-kinase) signaling pathways, we discovered that expression of the survival kinases PRKACA and PIM1 rescued cells from anti-HER2 therapy. Furthermore, we observed elevated PRKACA expression in trastuzumab-resistant breast cancer samples, indicating that this pathway is activated in breast cancers that are clinically resistant to trastuzumab-containing therapy. We found that neither PRKACA nor PIM1 restored MAPK or PI3K activation after lapatinib or trastuzumab treatment, but rather inactivated the pro-apoptotic protein BAD, the BCl-2-associated death promoter, thereby permitting survival signaling through BCL- XL. Pharmacological blockade of BCL-XL/BCL-2 partially abrogated the rescue effects conferred by PRKACA and PIM1, and sensitized cells to lapatinib treatment. These observations suggest that combined targeting of HER2 and the BCL-XL/BCL-2 anti-apoptotic pathway may increase responses to anti-HER2 therapy in breast cancer and decrease the emergence of resistant disease.