A FRET Based High Content Screen Identifies DMPK As a Novel Tether of ER and Mitochondria
Total Page:16
File Type:pdf, Size:1020Kb

Load more
Recommended publications
-

Deregulated Gene Expression Pathways in Myelodysplastic Syndrome Hematopoietic Stem Cells
Leukemia (2010) 24, 756–764 & 2010 Macmillan Publishers Limited All rights reserved 0887-6924/10 $32.00 www.nature.com/leu ORIGINAL ARTICLE Deregulated gene expression pathways in myelodysplastic syndrome hematopoietic stem cells A Pellagatti1, M Cazzola2, A Giagounidis3, J Perry1, L Malcovati2, MG Della Porta2,MJa¨dersten4, S Killick5, A Verma6, CJ Norbury7, E Hellstro¨m-Lindberg4, JS Wainscoat1 and J Boultwood1 1LRF Molecular Haematology Unit, NDCLS, John Radcliffe Hospital, Oxford, UK; 2Department of Hematology Oncology, University of Pavia Medical School, Fondazione IRCCS Policlinico San Matteo, Pavia, Italy; 3Medizinische Klinik II, St Johannes Hospital, Duisburg, Germany; 4Division of Hematology, Department of Medicine, Karolinska Institutet, Stockholm, Sweden; 5Department of Haematology, Royal Bournemouth Hospital, Bournemouth, UK; 6Albert Einstein College of Medicine, Bronx, NY, USA and 7Sir William Dunn School of Pathology, University of Oxford, Oxford, UK To gain insight into the molecular pathogenesis of the the World Health Organization.6,7 Patients with refractory myelodysplastic syndromes (MDS), we performed global gene anemia (RA) with or without ringed sideroblasts, according to expression profiling and pathway analysis on the hemato- poietic stem cells (HSC) of 183 MDS patients as compared with the the French–American–British classification, were subdivided HSC of 17 healthy controls. The most significantly deregulated based on the presence or absence of multilineage dysplasia. In pathways in MDS include interferon signaling, thrombopoietin addition, patients with RA with excess blasts (RAEB) were signaling and the Wnt pathways. Among the most signifi- subdivided into two categories, RAEB1 and RAEB2, based on the cantly deregulated gene pathways in early MDS are immuno- percentage of bone marrow blasts. -

Supplemental Information to Mammadova-Bach Et Al., “Laminin Α1 Orchestrates VEGFA Functions in the Ecosystem of Colorectal Carcinogenesis”
Supplemental information to Mammadova-Bach et al., “Laminin α1 orchestrates VEGFA functions in the ecosystem of colorectal carcinogenesis” Supplemental material and methods Cloning of the villin-LMα1 vector The plasmid pBS-villin-promoter containing the 3.5 Kb of the murine villin promoter, the first non coding exon, 5.5 kb of the first intron and 15 nucleotides of the second villin exon, was generated by S. Robine (Institut Curie, Paris, France). The EcoRI site in the multi cloning site was destroyed by fill in ligation with T4 polymerase according to the manufacturer`s instructions (New England Biolabs, Ozyme, Saint Quentin en Yvelines, France). Site directed mutagenesis (GeneEditor in vitro Site-Directed Mutagenesis system, Promega, Charbonnières-les-Bains, France) was then used to introduce a BsiWI site before the start codon of the villin coding sequence using the 5’ phosphorylated primer: 5’CCTTCTCCTCTAGGCTCGCGTACGATGACGTCGGACTTGCGG3’. A double strand annealed oligonucleotide, 5’GGCCGGACGCGTGAATTCGTCGACGC3’ and 5’GGCCGCGTCGACGAATTCACGC GTCC3’ containing restriction site for MluI, EcoRI and SalI were inserted in the NotI site (present in the multi cloning site), generating the plasmid pBS-villin-promoter-MES. The SV40 polyA region of the pEGFP plasmid (Clontech, Ozyme, Saint Quentin Yvelines, France) was amplified by PCR using primers 5’GGCGCCTCTAGATCATAATCAGCCATA3’ and 5’GGCGCCCTTAAGATACATTGATGAGTT3’ before subcloning into the pGEMTeasy vector (Promega, Charbonnières-les-Bains, France). After EcoRI digestion, the SV40 polyA fragment was purified with the NucleoSpin Extract II kit (Machery-Nagel, Hoerdt, France) and then subcloned into the EcoRI site of the plasmid pBS-villin-promoter-MES. Site directed mutagenesis was used to introduce a BsiWI site (5’ phosphorylated AGCGCAGGGAGCGGCGGCCGTACGATGCGCGGCAGCGGCACG3’) before the initiation codon and a MluI site (5’ phosphorylated 1 CCCGGGCCTGAGCCCTAAACGCGTGCCAGCCTCTGCCCTTGG3’) after the stop codon in the full length cDNA coding for the mouse LMα1 in the pCIS vector (kindly provided by P. -

Gene Symbol Gene Description ACVR1B Activin a Receptor, Type IB
Table S1. Kinase clones included in human kinase cDNA library for yeast two-hybrid screening Gene Symbol Gene Description ACVR1B activin A receptor, type IB ADCK2 aarF domain containing kinase 2 ADCK4 aarF domain containing kinase 4 AGK multiple substrate lipid kinase;MULK AK1 adenylate kinase 1 AK3 adenylate kinase 3 like 1 AK3L1 adenylate kinase 3 ALDH18A1 aldehyde dehydrogenase 18 family, member A1;ALDH18A1 ALK anaplastic lymphoma kinase (Ki-1) ALPK1 alpha-kinase 1 ALPK2 alpha-kinase 2 AMHR2 anti-Mullerian hormone receptor, type II ARAF v-raf murine sarcoma 3611 viral oncogene homolog 1 ARSG arylsulfatase G;ARSG AURKB aurora kinase B AURKC aurora kinase C BCKDK branched chain alpha-ketoacid dehydrogenase kinase BMPR1A bone morphogenetic protein receptor, type IA BMPR2 bone morphogenetic protein receptor, type II (serine/threonine kinase) BRAF v-raf murine sarcoma viral oncogene homolog B1 BRD3 bromodomain containing 3 BRD4 bromodomain containing 4 BTK Bruton agammaglobulinemia tyrosine kinase BUB1 BUB1 budding uninhibited by benzimidazoles 1 homolog (yeast) BUB1B BUB1 budding uninhibited by benzimidazoles 1 homolog beta (yeast) C9orf98 chromosome 9 open reading frame 98;C9orf98 CABC1 chaperone, ABC1 activity of bc1 complex like (S. pombe) CALM1 calmodulin 1 (phosphorylase kinase, delta) CALM2 calmodulin 2 (phosphorylase kinase, delta) CALM3 calmodulin 3 (phosphorylase kinase, delta) CAMK1 calcium/calmodulin-dependent protein kinase I CAMK2A calcium/calmodulin-dependent protein kinase (CaM kinase) II alpha CAMK2B calcium/calmodulin-dependent -

Acidic Phospholipids,47, 58 Acidification Steps, 23 Adsorbent, 53
Index acidic phospholipids,47, 58 ATPase, 22, 107 acidification steps, 23 auto-oxidation, 48,49 adsorbent, 53 aequorin, 95 aggregated mitochondria, 23 bacterial cells, 10 alcohols, 49 bacteriorhodopsin (BR), 218,232 alkaline hydrolysis, I bee venom, 121 alkyl esters, 57, 59 binders,53 amide bond, 129 silica gel, 53, 65, 176 amide linkage, 14 silica gel H, 176 amino alcohol, II bioactive phospholipids, 144 amino-group labelling reagents, 121 blanching, 50 aminopeptidase, 17 buoyant density, 16 aminophospholipid, 113, 118 butyl hydroxy toluene (BHT), 49,212 aminophospholipid pump, 119, 140 aminophospholipid translocase, 11 3 amphotericin B (AmB), 107 Cal+ -ATPases, 28, 107 N-(1-deoxyD-fructos-1-yl) AmB, 107 Cal+ -uptake, 27 anchored lipids, 38 campesterol, 104 angular amplitude, 89 calciferol, 78 anilino-8-naphthalene sulfonate (ANS), Candida albicans, 59, 60, 61, 76, 78, 102, 94, 103 104, 108 animal cells, 10 caproic acid, 129, 130 animal tissues, 9 carotenoids, 37 anion transporter, 135 cell debris, 33 anisotropy, 81 , 89, 95, 96, 97, 102, 104, cell disintegrator, 19 105 ceramide, 14, 129, 194 antagonists, 178 ceramide monohexosides ( CMH), 60 antioxidant, 49, 50 cerebroside, 14 apolar lipids, 56, 57, 69 chemical probes,112 arachidonic acid (AA), 144,153, 161 chloroplast, 32, 33 artificial membrane, 83 isolation, 32, 33 ascending chromatography, 54 purification, 33 asymmetric topology, 128 cholesterol, 14, 15,212 asymmetry, 112, 113,119 choline, 7, 9 atebrin, 95 chromatograms, 55 Index 249 chromatographic analyses, 52 ELISA, 146, 148 -

ACVR1 Antibody Cat
ACVR1 Antibody Cat. No.: 4791 Western blot analysis of ACVR1 in A549 cell lysate with ACVR1 antibody at 1 μg/mL in (A) the absence and (B) the presence of blocking peptide. Specifications HOST SPECIES: Rabbit SPECIES REACTIVITY: Human, Mouse HOMOLOGY: Predicted species reactivity based on immunogen sequence: Bovine: (100%), Rat: (93%) ACVR1 antibody was raised against a 14 amino acid synthetic peptide near the amino terminus of the human ACVR1. IMMUNOGEN: The immunogen is located within the first 50 amino acids of ACVR1. TESTED APPLICATIONS: ELISA, WB ACVR1 antibody can be used for detection of ACVR1 by Western blot at 1 μg/mL. APPLICATIONS: Antibody validated: Western Blot in human samples. All other applications and species not yet tested. At least four isoforms of ACVR1 are known to exist. This antibody is predicted to have no SPECIFICITY: cross-reactivity to ACVR1B or ACVR1C. POSITIVE CONTROL: 1) Cat. No. 1203 - A549 Cell Lysate Properties October 1, 2021 1 https://www.prosci-inc.com/acvr1-antibody-4791.html PURIFICATION: ACVR1 Antibody is affinity chromatography purified via peptide column. CLONALITY: Polyclonal ISOTYPE: IgG CONJUGATE: Unconjugated PHYSICAL STATE: Liquid BUFFER: ACVR1 Antibody is supplied in PBS containing 0.02% sodium azide. CONCENTRATION: 1 mg/mL ACVR1 antibody can be stored at 4˚C for three months and -20˚C, stable for up to one STORAGE CONDITIONS: year. As with all antibodies care should be taken to avoid repeated freeze thaw cycles. Antibodies should not be exposed to prolonged high temperatures. Additional Info OFFICIAL SYMBOL: ACVR1 ACVR1 Antibody: FOP, ALK2, SKR1, TSRI, ACTRI, ACVR1A, ACVRLK2, Activin receptor type-1, ALTERNATE NAMES: Activin receptor type I, ACTR-I ACCESSION NO.: NP_001096 PROTEIN GI NO.: 4501895 GENE ID: 90 USER NOTE: Optimal dilutions for each application to be determined by the researcher. -

Funkce CDK12 a CDK13 V Regulaci Transkripce Hana Paculová
MASARYKOVA UNIVERZITA PŘÍRODOVĚDECKÁ FAKULTA ÚSTAV BIOCHEMIE Funkce CDK12 a CDK13 v regulaci transkripce Disertační práce Hana Paculová Školitel: Mgr. Jiří Kohoutek, Ph.D Brno 2018 Bibliogra cký záznam Autorka: Mgr. Hana Paculová Prrodovedecáá aaául,a鏈 Maaarkáova unvverv,a Úa,av bvochemve Název práce: Funáce CDK12 a CDK13 v regulacv ,ranaárvpce Studijní program: Bvochemve Studijní obor: Bvochemve Školitel: Mgr. Jvr Kohou,eá鏈 Ph.D Akademický rok: 2017/2018 Po et stran: 89 Klí ová slova: Ckálvn-dependen,n ávnaaa鏈 CDK12鏈 ,ranaárvpce鏈 RNA polkmeraaa II鏈 raáovvna vaječnáů鏈 CHK1 Bibliographic entry Author: Mgr. Hana Paculová Facul,k oa acvence鏈 Maaarká unvverav,k Department of Biochemistry Title oF dissertation: CDK12 and CDK13 aunc,von vn ,ranacrvp,von regula,von Degree programme: Bvochemva,rk Field oF study: Bvochemva,rk Supervisor: Mgr. Jvr Kohou,eá鏈 Ph.D Academic year: 2017/2018 Number oF pages: 89 Keywords: Ckclvn-dependen ávnaae鏈 CDK12鏈 ,ranacrvp,von鏈 RNA polkmeraae II鏈 ovarvan cancer鏈 CHK1 Abstrakt Ckálvn-dependen,n ávnaaa 12 (CDK12) je ,ranaárvpčn ávnaaa鏈 á,erá rd expreav avých clových genů ,m鏈 že aoaaorkluje RNA polkmeraau II v průbehu elongačn aáe ,ranaárvpce. CDK12 je apojena do neáolváa bunečných preceaů鏈 což ahrnuje odpoveď na pošáoen DNA鏈 vývoj a bunečnou dvaerencvacv a aea,rvh mRNA. CDK12 bkla popaána jaáo jeden genů鏈 á,eré jaou čaa,o mu,ovánk v hvgh-grade aerónm ovarválnm áarcvnomu鏈 nvcméne vlvv ,ech,o mu,ac na aunácv CDK12 a jejvch role v áarcvnogenev dopoaud nebkla a,anovena. Zjva,vlv jame鏈 že ve,švna mu,ac CDK12鏈 á,eré bklk naleenk v nádorech鏈 brán vk,voren áomplexu CDK12 a Ckálvnem K a vnhvbuj ávnaaovou aá,vvv,u CDK12. -

Bioluminescent Properties of Semi-Synthetic Obelin and Aequorin Activated by Coelenterazine Analogues with Modifications of C-2, C-6, and C-8 Substituents
International Journal of Molecular Sciences Article Bioluminescent Properties of Semi-Synthetic Obelin and Aequorin Activated by Coelenterazine Analogues with Modifications of C-2, C-6, and C-8 Substituents 1, 2,3, 1 2, Elena V. Eremeeva y , Tianyu Jiang y , Natalia P. Malikova , Minyong Li * and Eugene S. Vysotski 1,* 1 Photobiology Laboratory, Institute of Biophysics SB RAS, Federal Research Center “Krasnoyarsk Science Center SB RAS”, Krasnoyarsk 660036, Russia; [email protected] (E.V.E.); [email protected] (N.P.M.) 2 Key Laboratory of Chemical Biology (MOE), Department of Medicinal Chemistry, School of Pharmaceutical Sciences, Shandong University, Jinan 250012, China; [email protected] 3 State Key Laboratory of Microbial Technology, Shandong University–Helmholtz Institute of Biotechnology, Shandong University, Qingdao 266237, China * Correspondence: [email protected] (M.L.); [email protected] (E.S.V.); Tel.: +86-531-8838-2076 (M.L.); +7-(391)-249-44-30 (E.S.V.); Fax: +86-531-8838-2076 (M.L.); +7-(391)-290-54-90 (E.S.V.) These authors contributed equally to this work. y Received: 23 June 2020; Accepted: 27 July 2020; Published: 30 July 2020 Abstract: Ca2+-regulated photoproteins responsible for bioluminescence of a variety of marine organisms are single-chain globular proteins within the inner cavity of which the oxygenated coelenterazine, 2-hydroperoxycoelenterazine, is tightly bound. Alongside with native coelenterazine, photoproteins can also use its synthetic analogues as substrates to produce flash-type bioluminescence. However, information on the effect of modifications of various groups of coelenterazine and amino acid environment of the protein active site on the bioluminescent properties of the corresponding semi-synthetic photoproteins is fragmentary and often controversial. -

Advancing a Clinically Relevant Perspective of the Clonal Nature of Cancer
Advancing a clinically relevant perspective of the clonal nature of cancer Christian Ruiza,b, Elizabeth Lenkiewicza, Lisa Eversa, Tara Holleya, Alex Robesona, Jeffrey Kieferc, Michael J. Demeurea,d, Michael A. Hollingsworthe, Michael Shenf, Donna Prunkardf, Peter S. Rabinovitchf, Tobias Zellwegerg, Spyro Moussesc, Jeffrey M. Trenta,h, John D. Carpteni, Lukas Bubendorfb, Daniel Von Hoffa,d, and Michael T. Barretta,1 aClinical Translational Research Division, Translational Genomics Research Institute, Scottsdale, AZ 85259; bInstitute for Pathology, University Hospital Basel, University of Basel, 4031 Basel, Switzerland; cGenetic Basis of Human Disease, Translational Genomics Research Institute, Phoenix, AZ 85004; dVirginia G. Piper Cancer Center, Scottsdale Healthcare, Scottsdale, AZ 85258; eEppley Institute for Research in Cancer and Allied Diseases, Nebraska Medical Center, Omaha, NE 68198; fDepartment of Pathology, University of Washington, Seattle, WA 98105; gDivision of Urology, St. Claraspital and University of Basel, 4058 Basel, Switzerland; hVan Andel Research Institute, Grand Rapids, MI 49503; and iIntegrated Cancer Genomics Division, Translational Genomics Research Institute, Phoenix, AZ 85004 Edited* by George F. Vande Woude, Van Andel Research Institute, Grand Rapids, MI, and approved June 10, 2011 (received for review March 11, 2011) Cancers frequently arise as a result of an acquired genomic insta- on the basis of morphology alone (8). Thus, the application of bility and the subsequent clonal evolution of neoplastic cells with purification methods such as laser capture microdissection does variable patterns of genetic aberrations. Thus, the presence and not resolve the complexities of many samples. A second approach behaviors of distinct clonal populations in each patient’s tumor is to passage tumor biopsies in tissue culture or in xenografts (4, 9– may underlie multiple clinical phenotypes in cancers. -

Multi-Modal Meta-Analysis of 1494 Hepatocellular Carcinoma Samples Reveals
Author Manuscript Published OnlineFirst on September 21, 2018; DOI: 10.1158/1078-0432.CCR-18-0088 Author manuscripts have been peer reviewed and accepted for publication but have not yet been edited. Multi-modal meta-analysis of 1494 hepatocellular carcinoma samples reveals significant impact of consensus driver genes on phenotypes Kumardeep Chaudhary1, Olivier B Poirion1, Liangqun Lu1,2, Sijia Huang1,2, Travers Ching1,2, Lana X Garmire1,2,3* 1Epidemiology Program, University of Hawaii Cancer Center, Honolulu, HI 96813, USA 2Molecular Biosciences and Bioengineering Graduate Program, University of Hawaii at Manoa, Honolulu, HI 96822, USA 3Current affiliation: Department of Computational Medicine and Bioinformatics, Building 520, 1600 Huron Parkway, Ann Arbor, MI 48109 Short Title: Impact of consensus driver genes in hepatocellular carcinoma * To whom correspondence should be addressed. Lana X. Garmire, Department of Computational Medicine and Bioinformatics Medical School, University of Michigan Building 520, 1600 Huron Parkway Ann Arbor-48109, MI, USA, Phone: +1-(734) 615-5510 Current email address: [email protected] Grant Support: This research was supported by grants K01ES025434 awarded by NIEHS through funds provided by the trans-NIH Big Data to Knowledge (BD2K) initiative (http://datascience.nih.gov/bd2k), P20 COBRE GM103457 awarded by NIH/NIGMS, NICHD R01 HD084633 and NLM R01LM012373 and Hawaii Community Foundation Medical Research Grant 14ADVC-64566 to Lana X Garmire. 1 Downloaded from clincancerres.aacrjournals.org on October 1, 2021. © 2018 American Association for Cancer Research. Author Manuscript Published OnlineFirst on September 21, 2018; DOI: 10.1158/1078-0432.CCR-18-0088 Author manuscripts have been peer reviewed and accepted for publication but have not yet been edited. -

Saracatinib Is an Efficacious Clinical Candidate for Fibrodysplasia Ossificans Progressiva
RESEARCH ARTICLE Saracatinib is an efficacious clinical candidate for fibrodysplasia ossificans progressiva Eleanor Williams,1 Jana Bagarova,2 Georgina Kerr,1 Dong-Dong Xia,2 Elsie S. Place,3 Devaveena Dey,2 Yue Shen,2 Geoffrey A. Bocobo,2 Agustin H. Mohedas,2 Xiuli Huang,4 Philip E. Sanderson,4 Arthur Lee,4 Wei Zheng,4 Aris N. Economides,5 James C. Smith,3 Paul B. Yu,2 and Alex N. Bullock1 1Centre for Medicines Discovery, University of Oxford, Oxford, United Kingdom. 2Department of Medicine, Cardiovascular Division, Brigham and Women’s Hospital, Harvard Medical School, Boston, Massachusetts, USA. 3Developmental Biology Laboratory, Francis Crick Institute, London, United Kingdom. 4National Center for Advancing Translational Sciences, NIH, Bethesda, Maryland, USA. 5Regeneron Pharmaceuticals Inc., Tarrytown, New York, USA. Currently, no effective therapies exist for fibrodysplasia ossificans progressiva (FOP), a rare congenital syndrome in which heterotopic bone is formed in soft tissues owing to dysregulated activity of the bone morphogenetic protein (BMP) receptor kinase ALK2 (also known as ACVR1). From a screen of known biologically active compounds, we identified saracatinib as a potent ALK2 kinase inhibitor. In enzymatic and cell-based assays, saracatinib preferentially inhibited ALK2, compared with other receptors of the BMP/TGF-β signaling pathway, and induced dorsalization in zebrafish embryos consistent with BMP antagonism. We further tested the efficacy of saracatinib using an inducible ACVR1Q207D-transgenic mouse line, which provides a model of heterotopic ossification (HO), as well as an inducible ACVR1R206H-knockin mouse, which serves as a genetically and physiologically faithful FOP model. In both models, saracatinib was well tolerated and potently inhibited the development of HO, even when administered transiently following soft tissue injury. -
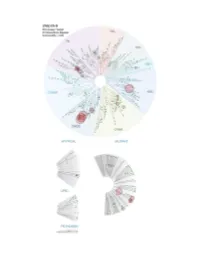
Profiling Data
Compound Name DiscoveRx Gene Symbol Entrez Gene Percent Compound Symbol Control Concentration (nM) JNK-IN-8 AAK1 AAK1 69 1000 JNK-IN-8 ABL1(E255K)-phosphorylated ABL1 100 1000 JNK-IN-8 ABL1(F317I)-nonphosphorylated ABL1 87 1000 JNK-IN-8 ABL1(F317I)-phosphorylated ABL1 100 1000 JNK-IN-8 ABL1(F317L)-nonphosphorylated ABL1 65 1000 JNK-IN-8 ABL1(F317L)-phosphorylated ABL1 61 1000 JNK-IN-8 ABL1(H396P)-nonphosphorylated ABL1 42 1000 JNK-IN-8 ABL1(H396P)-phosphorylated ABL1 60 1000 JNK-IN-8 ABL1(M351T)-phosphorylated ABL1 81 1000 JNK-IN-8 ABL1(Q252H)-nonphosphorylated ABL1 100 1000 JNK-IN-8 ABL1(Q252H)-phosphorylated ABL1 56 1000 JNK-IN-8 ABL1(T315I)-nonphosphorylated ABL1 100 1000 JNK-IN-8 ABL1(T315I)-phosphorylated ABL1 92 1000 JNK-IN-8 ABL1(Y253F)-phosphorylated ABL1 71 1000 JNK-IN-8 ABL1-nonphosphorylated ABL1 97 1000 JNK-IN-8 ABL1-phosphorylated ABL1 100 1000 JNK-IN-8 ABL2 ABL2 97 1000 JNK-IN-8 ACVR1 ACVR1 100 1000 JNK-IN-8 ACVR1B ACVR1B 88 1000 JNK-IN-8 ACVR2A ACVR2A 100 1000 JNK-IN-8 ACVR2B ACVR2B 100 1000 JNK-IN-8 ACVRL1 ACVRL1 96 1000 JNK-IN-8 ADCK3 CABC1 100 1000 JNK-IN-8 ADCK4 ADCK4 93 1000 JNK-IN-8 AKT1 AKT1 100 1000 JNK-IN-8 AKT2 AKT2 100 1000 JNK-IN-8 AKT3 AKT3 100 1000 JNK-IN-8 ALK ALK 85 1000 JNK-IN-8 AMPK-alpha1 PRKAA1 100 1000 JNK-IN-8 AMPK-alpha2 PRKAA2 84 1000 JNK-IN-8 ANKK1 ANKK1 75 1000 JNK-IN-8 ARK5 NUAK1 100 1000 JNK-IN-8 ASK1 MAP3K5 100 1000 JNK-IN-8 ASK2 MAP3K6 93 1000 JNK-IN-8 AURKA AURKA 100 1000 JNK-IN-8 AURKA AURKA 84 1000 JNK-IN-8 AURKB AURKB 83 1000 JNK-IN-8 AURKB AURKB 96 1000 JNK-IN-8 AURKC AURKC 95 1000 JNK-IN-8 -
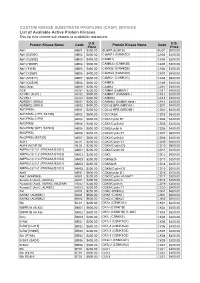
CUSTOM KINASE SUBSTRATE PROFILING (CKSP) SERVICE List of Available Active Protein Kinases This List May Change with Respect to Availability and Pricing
CUSTOM KINASE SUBSTRATE PROFILING (CKSP) SERVICE List of Available Active Protein Kinases This list may change with respect to availability and pricing. Protein Kinase Name Code U.S. Protein Kinase Name Code U.S. Price Price Abl1 AB01 $200.00 BUBR1(BUB1B) BU01 $600.00 Abl1 [E255K] AB02 $400.00 CaMK1δ (CAMK1D) CA03 $200.00 Abl1 [G250E] AB03 $400.00 CAMK1γ CA04 $200.00 Abl1 [H369P] AB04 $600.00 CAMK2α (CAMK2B) CA05 $200.00 Abl1 [T315I] AB05 $400.00 CaMK2β (CAMK2B) CA06 $200.00 Abl1 [Y253F] AB06 $400.00 CaMK2δ (CAMK2D) CA07 $400.00 Abl1 [M351T] AB07 $600.00 CaMK2γ (CAMK2G) CA08 $600.00 Abl1 [Q252H] AB08 $600.00 CAMK3γ CA09 $200.00 Abl2 (Arg) AB09 $200.00 CAMK4 CA10 $200.00 ACK AC01 $200.00 CAMK4 (CaMKIV ) CA11 $400.00 ACVR1 (ALK2 ) AC02 $400.00 CAMKK1 (CAMKKA ) CA12 $200.00 ACVRL1 AC03 $400.00 CAMKK2 CA13 $200.00 ADRBK1 (GRK2) AD01 $200.00 CAMKK2 (CaMKK beta ) CA14 $400.00 ADRBK2 (GRK3) AD02 $400.00 CDC42 BPA (MRCKA ) CD01 $400.00 Akt1/PKBα AK01 $200.00 CDC42 BPB (MRCKB ) CD02 $400.00 Akt1/PKBα [δPH, S473D] AK02 $600.00 CDC7/ASK CD03 $600.00 Akt1/PKBα [δPH] AK03 $600.00 CDK1/cyclin B1 CD04 $400.00 Akt2/PKBβ AK04 $200.00 CDK1/CyclinA2 CD05 $200.00 Akt2/PKBβ [δPH, S474D] AK05 $600.00 CDK2/cyclin A CD06 $400.00 Akt3/PKBγ AK06 $200.00 CDK2/Cyclin E1 CD07 $600.00 Akt3/PKBγ [S472D] AK07 $600.00 CDK2/CyclinA2 CD08 $200.00 ALK1 AL01 $200.00 CDK3/Cyclin E1 CD09 $600.00 ALK4 (ACVR1B) AL02 $200.00 CDK4//Cyclin D3 CD10 $600.00 AMPKα1/β1/γ1 (PRKAA1/B1/G1) AM01 $200.00 CDK4/Cyclin D1 CD11 $200.00 AMPKα1/β1/γ2 (PRKAA1/B1/G2) AM02 $200.00 CDK5 CD12 $600.00