A Comparative Genomics Study of the Genes Involved in the Catabolic Pathways of Naphthalene”
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Comparative Genomics of Pandoraea, a Genus Enriched in Xenobiotic Biodegradation and Metabolism
fmicb-10-02556 November 4, 2019 Time: 15:40 # 1 ORIGINAL RESEARCH published: 06 November 2019 doi: 10.3389/fmicb.2019.02556 Comparative Genomics of Pandoraea, a Genus Enriched in Xenobiotic Biodegradation and Metabolism Charlotte Peeters1, Evelien De Canck1, Margo Cnockaert1, Evie De Brandt1, Cindy Snauwaert2, Bart Verheyde1, Eliza Depoorter1, Theodore Spilker3, John J. LiPuma3 and Peter Vandamme1,2* 1 Laboratory of Microbiology, Department of Biochemistry and Microbiology, Faculty of Sciences, Ghent University, Ghent, Belgium, 2 BCCM/LMG Bacteria Collection, Department of Biochemistry and Microbiology, Faculty of Sciences, Ghent Edited by: University, Ghent, Belgium, 3 Department of Pediatrics, University of Michigan Medical School, Ann Arbor, MI, United States Iain Sutcliffe, Northumbria University, United Kingdom Comparative analysis of partial gyrB, recA, and gltB gene sequences of 84 Pandoraea Reviewed by: reference strains and field isolates revealed several clusters that included no taxonomic Martin W. Hahn, reference strains. The gyrB, recA, and gltB phylogenetic trees were used to select 27 University of Innsbruck, Austria Stephanus Nicolaas Venter, strains for whole-genome sequence analysis and for a comparative genomics study that University of Pretoria, South Africa also included 41 publicly available Pandoraea genome sequences. The phylogenomic Aharon Oren, The Hebrew University of Jerusalem, analyses included a Genome BLAST Distance Phylogeny approach to calculate pairwise Israel digital DNA–DNA hybridization values and their -

Genomic and Proteomic Analysis of Lignin Degrading and Polyhydroxyalkanoate Accumulating Β‑Proteobacterium Pandoraea Sp
Kumar et al. Biotechnol Biofuels (2018) 11:154 https://doi.org/10.1186/s13068-018-1148-2 Biotechnology for Biofuels RESEARCH Open Access Genomic and proteomic analysis of lignin degrading and polyhydroxyalkanoate accumulating β‑proteobacterium Pandoraea sp. ISTKB Madan Kumar1, Sandhya Verma2, Rajesh Kumar Gazara2, Manish Kumar1, Ashok Pandey3, Praveen Kumar Verma2* and Indu Shekhar Thakur1* Abstract Background: Lignin is a major component of plant biomass and is recalcitrant to degradation due to its complex and heterogeneous aromatic structure. The biomass-based research mainly focuses on polysaccharides component of biomass and lignin is discarded as waste with very limited usage. The sustainability and success of plant polysac- charide-based biorefnery can be possible if lignin is utilized in improved ways and with minimal waste generation. Discovering new microbial strains and understanding their enzyme system for lignin degradation are necessary for its conversion into fuel and chemicals. The Pandoraea sp. ISTKB was previously characterized for lignin degradation and successfully applied for pretreatment of sugarcane bagasse and polyhydroxyalkanoate (PHA) production. In this study, genomic analysis and proteomics on aromatic polymer kraft lignin and vanillic acid are performed to fnd the impor- tant enzymes for polymer utilization. Results: Genomic analysis of Pandoraea sp. ISTKB revealed the presence of strong lignin degradation machinery and identifed various candidate genes responsible for lignin degradation and PHA production. -
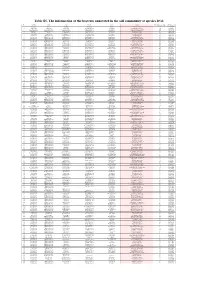
Table S5. the Information of the Bacteria Annotated in the Soil Community at Species Level
Table S5. The information of the bacteria annotated in the soil community at species level No. Phylum Class Order Family Genus Species The number of contigs Abundance(%) 1 Firmicutes Bacilli Bacillales Bacillaceae Bacillus Bacillus cereus 1749 5.145782459 2 Bacteroidetes Cytophagia Cytophagales Hymenobacteraceae Hymenobacter Hymenobacter sedentarius 1538 4.52499338 3 Gemmatimonadetes Gemmatimonadetes Gemmatimonadales Gemmatimonadaceae Gemmatirosa Gemmatirosa kalamazoonesis 1020 3.000970902 4 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas indica 797 2.344876284 5 Firmicutes Bacilli Lactobacillales Streptococcaceae Lactococcus Lactococcus piscium 542 1.594633558 6 Actinobacteria Thermoleophilia Solirubrobacterales Conexibacteraceae Conexibacter Conexibacter woesei 471 1.385742446 7 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas taxi 430 1.265115184 8 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas wittichii 388 1.141545794 9 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas sp. FARSPH 298 0.876754244 10 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sorangium cellulosum 260 0.764953367 11 Proteobacteria Deltaproteobacteria Myxococcales Polyangiaceae Sorangium Sphingomonas sp. Cra20 260 0.764953367 12 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas panacis 252 0.741416341 -

Acidovorax Citrulli
Bulletin OEPP/EPPO Bulletin (2016) 46 (3), 444–462 ISSN 0250-8052. DOI: 10.1111/epp.12330 European and Mediterranean Plant Protection Organization Organisation Europe´enne et Me´diterrane´enne pour la Protection des Plantes PM 7/127 (1) Diagnostics Diagnostic PM 7/127 (1) Acidovorax citrulli Specific scope Specific approval and amendment This Standard describes a diagnostic protocol for Approved in 2016-09. Acidovorax citrulli.1 This Standard should be used in conjunction with PM 7/76 Use of EPPO diagnostic protocols. strain, were mainly isolated from non-watermelon, cucurbit 1. Introduction hosts such as cantaloupe melon (Cucumis melo var. Acidovorax citrulli is the causal agent of bacterial fruit cantalupensis), cucumber (Cucumis sativus), honeydew blotch and seedling blight of cucurbits (Webb & Goth, melon (Cucumis melo var. indorus), squash and pumpkin 1965; Schaad et al., 1978). This disease was sporadic until (Cucurbita pepo, Cucurbita maxima and Cucurbita the late 1980s (Wall & Santos, 1988), but recurrent epi- moschata) whereas Group II isolates were mainly recovered demics have been reported in the last 20 years (Zhang & from watermelon (Walcott et al., 2000, 2004; Burdman Rhodes, 1990; Somodi et al., 1991; Latin & Hopkins, et al., 2005). While Group I isolates were moderately 1995; Demir, 1996; Assis et al., 1999; Langston et al., aggressive on a range of cucurbit hosts, Group II isolates 1999; O’Brien & Martin, 1999; Burdman et al., 2005; Har- were highly aggressive on watermelon but moderately ighi, 2007; Holeva et al., 2010; Popovic & Ivanovic, 2015). aggressive on non-watermelon hosts (Walcott et al., 2000, The disease is particularly severe on watermelon (Citrullus 2004). -

Quorum Sensing Activity in Pandoraea Pnomenusa RB38
Sensors 2014, 14, 10177-10186; doi:10.3390/s140610177 OPEN ACCESS sensors ISSN 1424-8220 www.mdpi.com/journal/sensors Article Quorum Sensing Activity in Pandoraea pnomenusa RB38 Robson Ee, Yan-Lue Lim, Lin-Xin Kin, Wai-Fong Yin and Kok-Gan Chan * Division of Genetics and Molecular Biology, Institute of Biological Sciences, Faculty of Science, University of Malaya, Kuala Lumpur 50603, Malaysia; E-Mails: [email protected] (R.E.); [email protected] (Y.-L.L.); [email protected] (L.-X.K.); [email protected] (W.-F.Y.) * Author to whom correspondence should be addressed; E-Mail: [email protected]; Tel.: +60-37967-5162; Fax: +60-37967-4509. Received: 5 March 2014; in revised form: 25 May 2014 / Accepted: 28 May 2014 / Published: 10 June 2014 Abstract: Strain RB38 was recovered from a former dumping area in Malaysia. MALDI-TOF mass spectrometry and genomic analysis identified strain RB-38 as Pandoraea pnomenusa. Various biosensors confirmed its quorum sensing properties. High resolution triple quadrupole liquid chromatography–mass spectrometry analysis was subsequently used to characterize the N-acyl homoserine lactone production profile of P. pnomenusa strain RB38, which validated that this isolate produced N-octanoyl homoserine lactone as a quorum sensing molecule. This is the first report of the production of N-octanoyl homoserine lactone by P. pnomenusa strain RB38. Keywords: matrix-assisted laser desorption ionization time-of-flight (MALDI-TOF); mass spectrometry (MS); cell-to cell communication; triple quodruopole liquid chromatography mass spectrometry (LC-MS/MS); N-octanoyl homoserine lactone (C8-HSL) 1. Introduction Pandoraea is a Gram-negative rod shape proteobacteria of the Burkholderiaceae family that is often isolated from sputa of cystic fibrosis patients and soil [1]. -

Genome of Ca. Pandoraea Novymonadis, an Endosymbiotic Bacterium of the Trypanosomatid Novymonas Esmeraldas
fmicb-08-01940 September 30, 2017 Time: 16:0 # 1 ORIGINAL RESEARCH published: 04 October 2017 doi: 10.3389/fmicb.2017.01940 Genome of Ca. Pandoraea novymonadis, an Endosymbiotic Bacterium of the Trypanosomatid Novymonas esmeraldas Alexei Y. Kostygov1,2†, Anzhelika Butenko1,3†, Anna Nenarokova3,4, Daria Tashyreva3, Pavel Flegontov1,3,5, Julius Lukeš3,4 and Vyacheslav Yurchenko1,3,6* 1 Life Science Research Centre, Faculty of Science, University of Ostrava, Ostrava, Czechia, 2 Zoological Institute of the Russian Academy of Sciences, St. Petersburg, Russia, 3 Biology Centre, Institute of Parasitology, Czech Academy of Sciences, Ceskéˇ Budejovice,ˇ Czechia, 4 Faculty of Sciences, University of South Bohemia, Ceskéˇ Budejovice,ˇ Czechia, 5 Institute for Information Transmission Problems, Russian Academy of Sciences, Moscow, Russia, 6 Institute of Environmental Technologies, Faculty of Science, University of Ostrava, Ostrava, Czechia We have sequenced, annotated, and analyzed the genome of Ca. Pandoraea novymonadis, a recently described bacterial endosymbiont of the trypanosomatid Novymonas esmeraldas. When compared with genomes of its free-living relatives, it Edited by: has all the hallmarks of the endosymbionts’ genomes, such as significantly reduced João Marcelo Pereira Alves, University of São Paulo, Brazil size, extensive gene loss, low GC content, numerous gene rearrangements, and Reviewed by: low codon usage bias. In addition, Ca. P. novymonadis lacks mobile elements, Zhao-Rong Lun, has a strikingly low number of pseudogenes, and almost all genes are single Sun Yat-sen University, China Vera Tai, copied. This suggests that it already passed the intensive period of host adaptation, University of Western Ontario, Canada which still can be observed in the genome of Polynucleobacter necessarius, a *Correspondence: certainly recent endosymbiont. -

Pandoraea Apista Bacteremia in a COVID-Positive Man: a Rare Coinfection Case Report from North India
Published online: 2021-06-28 THIEME 192 Case Pandoraea Report apistain a COVID Patient Singh et al. Pandoraea apista Bacteremia in a COVID-Positive Man: A Rare Coinfection Case Report from North India Sweta Singh1 Chinmoy Sahu1 , Sangram Singh Patel1 Atul Garg1 Ujjala Ghoshal1 1Department of Microbiology, Sanjay Gandhi Post Graduate Address for correspondence Chinmoy Sahu, MD (Microbiology) Institute of Medical Sciences, Lucknow, Uttar Pradesh, India Department of Microbiology, Sanjay Gandhi Post Graduate Institute of Medical Sciences, Raebareli Road, Lucknow, Uttar Pradesh 226014, India (e-mail: [email protected]). J Lab Physicians 2021;13:192–194. Abstract Pandoraea apista is a novel gram-negative bacillus usually isolated from respiratory specimens of cystic fibrosis patients. Few cases of bacteremia have also been reported due to this rare pathogen. Emergence of multidrug-resistant isolates of this bacillus is of grave concern. Here, we report a very interesting and unusual case of Pandoraea Keywords apista bacteremia in a coronavirus disease (COVID)–positive elderly diabetic man suf- ► Pandoraea apista fering from pneumonia. Prompt isolation and antibiotic sensitivity testing guided the ► bacillus patient’s treatment and yielded favorable outcome. The need of automated methods ► cystic fibrosis for identification and sensitivity testing limits the reporting of this rare but important ► bacteremia pathogen in hospital settings. Detailed research work and studies are needed in this ► COVID direction to better understand this pathogen and its clinical manifestations for better ► pneumonia patient outcome. Introduction report a case of bacteremia caused by Pandoraea apista in a COVID-positive elderly man suffering from pneumonia. Pandoraea species is a newly emerging multidrug-resistant pathogen usually isolated from cystic fibrosis (CF) patients. -

Microbial Degradation of Organic Micropollutants in Hyporheic Zone Sediments
Microbial degradation of organic micropollutants in hyporheic zone sediments Dissertation To obtain the Academic Degree Doctor rerum naturalium (Dr. rer. nat.) Submitted to the Faculty of Biology, Chemistry, and Geosciences of the University of Bayreuth by Cyrus Rutere Bayreuth, May 2020 This doctoral thesis was prepared at the Department of Ecological Microbiology – University of Bayreuth and AG Horn – Institute of Microbiology, Leibniz University Hannover, from August 2015 until April 2020, and was supervised by Prof. Dr. Marcus. A. Horn. This is a full reprint of the dissertation submitted to obtain the academic degree of Doctor of Natural Sciences (Dr. rer. nat.) and approved by the Faculty of Biology, Chemistry, and Geosciences of the University of Bayreuth. Date of submission: 11. May 2020 Date of defense: 23. July 2020 Acting dean: Prof. Dr. Matthias Breuning Doctoral committee: Prof. Dr. Marcus. A. Horn (reviewer) Prof. Harold L. Drake, PhD (reviewer) Prof. Dr. Gerhard Rambold (chairman) Prof. Dr. Stefan Peiffer In the battle between the stream and the rock, the stream always wins, not through strength but by perseverance. Harriett Jackson Brown Jr. CONTENTS CONTENTS CONTENTS ............................................................................................................................ i FIGURES.............................................................................................................................. vi TABLES .............................................................................................................................. -

CGM-18-001 Perseus Report Update Bacterial Taxonomy Final Errata
report Update of the bacterial taxonomy in the classification lists of COGEM July 2018 COGEM Report CGM 2018-04 Patrick L.J. RÜDELSHEIM & Pascale VAN ROOIJ PERSEUS BVBA Ordering information COGEM report No CGM 2018-04 E-mail: [email protected] Phone: +31-30-274 2777 Postal address: Netherlands Commission on Genetic Modification (COGEM), P.O. Box 578, 3720 AN Bilthoven, The Netherlands Internet Download as pdf-file: http://www.cogem.net → publications → research reports When ordering this report (free of charge), please mention title and number. Advisory Committee The authors gratefully acknowledge the members of the Advisory Committee for the valuable discussions and patience. Chair: Prof. dr. J.P.M. van Putten (Chair of the Medical Veterinary subcommittee of COGEM, Utrecht University) Members: Prof. dr. J.E. Degener (Member of the Medical Veterinary subcommittee of COGEM, University Medical Centre Groningen) Prof. dr. ir. J.D. van Elsas (Member of the Agriculture subcommittee of COGEM, University of Groningen) Dr. Lisette van der Knaap (COGEM-secretariat) Astrid Schulting (COGEM-secretariat) Disclaimer This report was commissioned by COGEM. The contents of this publication are the sole responsibility of the authors and may in no way be taken to represent the views of COGEM. Dit rapport is samengesteld in opdracht van de COGEM. De meningen die in het rapport worden weergegeven, zijn die van de auteurs en weerspiegelen niet noodzakelijkerwijs de mening van de COGEM. 2 | 24 Foreword COGEM advises the Dutch government on classifications of bacteria, and publishes listings of pathogenic and non-pathogenic bacteria that are updated regularly. These lists of bacteria originate from 2011, when COGEM petitioned a research project to evaluate the classifications of bacteria in the former GMO regulation and to supplement this list with bacteria that have been classified by other governmental organizations. -

Complete Assembly of the Genome of an Acidovorax Citrulli Strain Reveals a Naturally Occurring Plasmid in This Species
fmicb-10-01400 June 19, 2019 Time: 15:19 # 1 ORIGINAL RESEARCH published: 20 June 2019 doi: 10.3389/fmicb.2019.01400 Complete Assembly of the Genome of an Acidovorax citrulli Strain Reveals a Naturally Occurring Plasmid in This Species Rongzhi Yang1, Diego Santos Garcia2, Francisco Pérez Montaño1,3, Gustavo Mateus da Silva1, Mei Zhao4, Irene Jiménez Guerrero1, Tally Rosenberg1, Gong Chen4, Inbar Plaschkes5, Shai Morin2, Ron Walcott4 and Saul Burdman1* 1 Department of Plant Pathology and Microbiology, The Robert H. Smith Faculty of Agriculture, Food and Environment, The Hebrew University of Jerusalem, Rehovot, Israel, 2 Department of Entomology, The Robert H. Smith Faculty of Agriculture, Food and Environment, The Hebrew University of Jerusalem, Rehovot, Israel, 3 Department of Microbiology, University of Seville, Seville, Spain, 4 Department of Plant Pathology, University of Georgia, Athens, GA, United States, 5 Bioinformatics Unit, The Robert H. Smith Faculty of Agriculture, Food and Environment, The Hebrew University of Jerusalem, Rehovot, Israel Edited by: Martin G. Klotz, Washington State University, Acidovorax citrulli is the causal agent of bacterial fruit blotch (BFB), a serious threat United States to cucurbit crop production worldwide. Based on genetic and phenotypic properties, Reviewed by: A. citrulli strains are divided into two major groups: group I strains have been generally Robert Wilson Jackson, isolated from melon and other non-watermelon cucurbits, while group II strains are University of Reading, United Kingdom closely associated with watermelon. In a previous study, we reported the genome Tingchang Zhao, of the group I model strain, M6. At that time, the M6 genome was sequenced by Chinese Academy of Agricultural Sciences, China MiSeq Illumina technology, with reads assembled into 139 contigs. -

Identification of Pseudomonas Species and Other Non-Glucose Fermenters
UK Standards for Microbiology Investigations Identification of Pseudomonas species and other Non- Glucose Fermenters Issued by the Standards Unit, Microbiology Services, PHE Bacteriology – Identification | ID 17 | Issue no: 3 | Issue date: 13.04.15 | Page: 1 of 41 © Crown copyright 2015 Identification of Pseudomonas species and other Non-Glucose Fermenters Acknowledgments UK Standards for Microbiology Investigations (SMIs) are developed under the auspices of Public Health England (PHE) working in partnership with the National Health Service (NHS), Public Health Wales and with the professional organisations whose logos are displayed below and listed on the website https://www.gov.uk/uk- standards-for-microbiology-investigations-smi-quality-and-consistency-in-clinical- laboratories. SMIs are developed, reviewed and revised by various working groups which are overseen by a steering committee (see https://www.gov.uk/government/groups/standards-for-microbiology-investigations- steering-committee). The contributions of many individuals in clinical, specialist and reference laboratories who have provided information and comments during the development of this document are acknowledged. We are grateful to the Medical Editors for editing the medical content. For further information please contact us at: Standards Unit Microbiology Services Public Health England 61 Colindale Avenue London NW9 5EQ E-mail: [email protected] Website: https://www.gov.uk/uk-standards-for-microbiology-investigations-smi-quality- and-consistency-in-clinical-laboratories -

Eukaryota Fungi Bacteria
Streptomyces-antibioticus Streptomyces-griseus Streptomyces-lincolnensis Streptomyces-phaeochromogenes Streptomyces-scabiei Streptomyces-violaceoruber Streptomyces-viridochromogenes Streptomyces-griseoruber Streptomyces-sclerotialus Streptomyces-olivochromogenes Streptomyces-cellulosae Streptomyces-chartreusis Streptomyces-cattleya Streptomyces-galbus Streptomyces-caelestis Streptomyces-subrutilus Streptomyces-acidiscabies Streptomyces-diastatochromogenes Streptomyces-neyagawaensis Streptomyces-canus Streptomyces-yerevanensis Streptomyces-afghaniensis Streptomyces-bicolor Streptomyces-cellostaticus Streptomyces-griseorubiginosus Streptomyces-phaeopurpureus Streptomyces-prasinus Streptomyces-prunicolor Streptomyces-pseudovenezuelae Streptomyces-resistomycificus Streptomyces Streptomyces-iakyrus Streptomyces-katrae Streptomyces-mirabilis Streptomyces-peruviensis Streptomyces-regalis Streptomyces-torulosus Streptomyces-turgidiscabies Streptomyces-ipomoeae Streptomyces-phaeoluteigriseus Streptomyces-curacoi Streptomyces-azureus Streptomyces-europaeiscabiei Streptomyces-stelliscabiei Streptomyces-puniciscabiei Streptomyces-aureus Streptomyces-ossamyceticus Streptomyces-variegatus Streptomyces-roseochromogenus Streptomyces-xylophagus Streptomyces-sviceus Streptomyces-fulvoviolaceus Streptomyces-bungoensis Streptomyces-erythrochromogenes Streptomycetaceae Streptomyces-davaonensis Streptomyces-olindensis Streptomyces-rubellomurinus Streptomyces-indicus Streptomyces-caeruleatus Streptomyces-hokutonensis Streptomyces-humi Streptomyces-jeddahensis