Prezentace Aplikace Powerpoint
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Novel Copper-Containing Membrane Monooxygenases (Cummos) Encoded by Alkane- Utilizing Betaproteobacteria
Lawrence Berkeley National Laboratory Recent Work Title Novel copper-containing membrane monooxygenases (CuMMOs) encoded by alkane- utilizing Betaproteobacteria. Permalink https://escholarship.org/uc/item/8z48x96q Journal The ISME journal, 14(3) ISSN 1751-7362 Authors Rochman, Fauziah F Kwon, Miye Khadka, Roshan et al. Publication Date 2020-03-01 DOI 10.1038/s41396-019-0561-2 Peer reviewed eScholarship.org Powered by the California Digital Library University of California 1 1 2 1Novel copper-containing membrane monooxygenases 2(CuMMOs) encoded by alkane-utilizing Betaproteobacteria 3 4Running title: Novel CuMMOs in Betaproteobacteria 5 6Fauziah F. Rochman1, Miye Kwon2, Roshan Khadka1, Ivica Tamas1,3, Azriel 7Abraham Lopez-Jauregui1,4, Andriy Sheremet1, Angela Smirnova1, Rex R. 8Malmstrom5, Sukhwan Yoon2, Tanja Woyke5, Peter F. Dunfield1*, Tobin J. 9Verbeke1 10 111 Department of Biological Sciences, University of Calgary, 2500 University 12Dr. NW Calgary AB Canada T2N 1N4 132 Department of Civil and Environmental Engineering, Korea Advanced 14Institute of Science and Technology, Daejeon, Korea 153Department of Biology and Ecology, University of Novi Sad Novi Sad, Serbia 164 Instituto Tecnologico y de Estudios Superiores de Monterrey, Chihuahua, 17Mexico 185 Department of Energy Joint Genome Institute, Walnut Creek, California, 19USA. 20 21*Corresponding author: [email protected]; 403-220-2469 22 3 2 4 23Competing interests: The authors declare that they have no competing 24interests. 5 3 6 25Abstract 26Copper-containing membrane monooxygenases (CuMMOs) are encoded by 27xmoCAB(D) gene clusters and catalyze the oxidation of methane, ammonia, 28or some short chain alkanes and alkenes. In a metagenome constructed from 29an oilsands tailings pond we detected an xmoCABD gene cluster with <59% 30derived amino acid identity to genes from known bacteria. -

Virulence Determinants, Drug Resistance and Mobile Genetic
Lau et al. Cell & Bioscience 2011, 1:17 http://www.cellandbioscience.com/content/1/1/17 Cell & Bioscience RESEARCH Open Access Virulence determinants, drug resistance and mobile genetic elements of Laribacter hongkongensis: a genome-wide analysis Susanna KP Lau1,2,3,4*†, Gilman KM Wong4†, Alan KL Tsang4†, Jade LL Teng4, Rachel YY Fan4, Herman Tse1,2,3,4, Kwok-Yung Yuen1,2,3,4 and Patrick CY Woo1,2,3,4* Abstract Background: Laribacter hongkongensis is associated with community-acquired gastroenteritis and traveler’s diarrhea. In this study, we performed an in-depth annotation of the genes in its genome related to the various steps in the infective process, drug resistance and mobile genetic elements. Results: For acid and bile resistance, L. hongkongensis possessed a urease gene cassette, two arc gene clusters and bile salt efflux systems. For intestinal colonization, it possessed a putative adhesin of the autotransporter family homologous to those of diffusely adherent Escherichia coli (E. coli) and enterotoxigenic E. coli. To evade from host defense, it possessed superoxide dismutase and catalases. For lipopolysaccharide biosynthesis, it possessed the same set of genes that encode enzymes for synthesizing lipid A, two Kdo units and heptose units as E. coli, but different genes for its symmetrical acylation pattern, and nine genes for polysaccharide side chains biosynthesis. It contained a number of CDSs that encode putative cell surface acting (RTX toxin and hemolysins) and intracellular cytotoxins (patatin-like proteins) and enzymes for invasion (outer membrane phospholipase A). It contained a broad variety of antibiotic resistance-related genes, including genes related to b-lactam (n = 10) and multidrug efflux (n = 54). -

Isolation, Characterization, and Applications of Rhizobiophages
University of Calgary PRISM: University of Calgary's Digital Repository Graduate Studies The Vault: Electronic Theses and Dissertations 2014-10-02 Isolation, characterization, and applications of rhizobiophages Halmillawewa, Anupama Halmillawewa, A. (2014). Isolation, characterization, and applications of rhizobiophages (Unpublished doctoral thesis). University of Calgary, Calgary, AB. doi:10.11575/PRISM/26680 http://hdl.handle.net/11023/1912 doctoral thesis University of Calgary graduate students retain copyright ownership and moral rights for their thesis. You may use this material in any way that is permitted by the Copyright Act or through licensing that has been assigned to the document. For uses that are not allowable under copyright legislation or licensing, you are required to seek permission. Downloaded from PRISM: https://prism.ucalgary.ca UNIVERSITY OF CALGARY Isolation, characterization, and applications of rhizobiophages by Anupama P. Halmillawewa A THESIS SUBMITTED TO THE FACULTY OF GRADUATE STUDIES IN PARTIAL FULFILMENT OF THE REQUIREMENTS FOR THE DEGREE OF DOCTOR OF PHILOSOPHY DEPARTMENT OF BIOLOGICAL SCIENCES CALGARY, ALBERTA AUGUST, 2014 © Anupama Halmillawewa 2014 Abstract Rhizobiophages are the group of bacteriophages that infect rhizobia. The rhizobia constitute a bacterial group that includes members of several different genera and are capable of nodulating legume plant roots, and providing a source of fixed nitrogen for the plant. Rhizobiophages can play a vital role in the Rhizobium-legume symbiosis by altering the population dynamics of the rhizobia present in the rhizosphere, which can be used in agriculture to improve the efficacy of commercial Rhizobium inoculants and mitigate the Rhizobium competition problem. As a prerequisite for the application of such technology, a thorough understanding of rhizobiophage biology is required. -
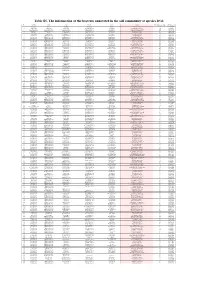
Table S5. the Information of the Bacteria Annotated in the Soil Community at Species Level
Table S5. The information of the bacteria annotated in the soil community at species level No. Phylum Class Order Family Genus Species The number of contigs Abundance(%) 1 Firmicutes Bacilli Bacillales Bacillaceae Bacillus Bacillus cereus 1749 5.145782459 2 Bacteroidetes Cytophagia Cytophagales Hymenobacteraceae Hymenobacter Hymenobacter sedentarius 1538 4.52499338 3 Gemmatimonadetes Gemmatimonadetes Gemmatimonadales Gemmatimonadaceae Gemmatirosa Gemmatirosa kalamazoonesis 1020 3.000970902 4 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas indica 797 2.344876284 5 Firmicutes Bacilli Lactobacillales Streptococcaceae Lactococcus Lactococcus piscium 542 1.594633558 6 Actinobacteria Thermoleophilia Solirubrobacterales Conexibacteraceae Conexibacter Conexibacter woesei 471 1.385742446 7 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas taxi 430 1.265115184 8 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas wittichii 388 1.141545794 9 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas sp. FARSPH 298 0.876754244 10 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sorangium cellulosum 260 0.764953367 11 Proteobacteria Deltaproteobacteria Myxococcales Polyangiaceae Sorangium Sphingomonas sp. Cra20 260 0.764953367 12 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas panacis 252 0.741416341 -

Microbial Ecology of Full-Scale Wastewater Treatment Systems in the Polar Arctic Circle: Archaea, Bacteria and Fungi
www.nature.com/scientificreports OPEN Microbial ecology of full-scale wastewater treatment systems in the Polar Arctic Circle: Archaea, Received: 28 November 2017 Accepted: 21 January 2018 Bacteria and Fungi Published: xx xx xxxx Alejandro Gonzalez-Martinez1, Maija Sihvonen1, Barbara Muñoz-Palazon2, Alejandro Rodriguez-Sanchez2, Anna Mikola1 & Riku Vahala1 Seven full-scale biological wastewater treatment systems located in the Polar Arctic Circle region in Finland were investigated to determine their Archaea, Bacteria and Fungi community structure, and their relationship with the operational conditions of the bioreactors by the means of quantitative PCR, massive parallel sequencing and multivariate redundancy analysis. The results showed dominance of Archaea and Bacteria members in the bioreactors. The activated sludge systems showed strong selection of Bacteria but not for Archaea and Fungi, as suggested by diversity analyses. Core OTUs in infuent and bioreactors were classifed as Methanobrevibacter, Methanosarcina, Terrestrial Group Thaumarchaeota and unclassifed Euryarchaeota member for Archaea; Trichococcus, Leptotrichiaceae and Comamonadaceae family, and Methylorosula for Bacteria and Trichosporonaceae family for Fungi. All infuents shared core OTUs in all domains, but in bioreactors this did not occur for Bacteria. Oligotype structure of core OTUs showed several ubiquitous Fungi oligotypes as dominant in sewage and bioreactors. Multivariate redundancy analyses showed that the majority of core OTUs were related to organic matter and nutrients removal. Also, there was evidence of competition among Archaea and Fungi core OTUs, while all Bacteria OTUs were positively correlated among them. The results obtained highlighted interesting features of extremely cold temperature bioreactors. Undoubtedly, microbial ecology of bioprocesses is a factor of major importance for the functioning of biopro- cesses, especially wastewater treatment (WWT) systems1. -

Ecogenomics and Genome Landscapes of Marine Pseudoalteromonas Phage H105/1
The ISME Journal (2010), 1–15 & 2010 International Society for Microbial Ecology All rights reserved 1751-7362/10 $32.00 www.nature.com/ismej ORIGINAL ARTICLE Ecogenomics and genome landscapes of marine Pseudoalteromonas phage H105/1 Melissa Beth Duhaime1,2, Antje Wichels3, Jost Waldmann1, Hanno Teeling1 and Frank Oliver Glo¨ckner1,2 1Microbial Genomics Group, Max Planck Institute for Marine Microbiology, Celsiusstrasse 1, Bremen, Germany; 2School of Engineering and Sciences, Jacobs University, Bremen, Germany and 3Biosciences Division, Alfred Wegener Institute for Polar and Marine Research, BAH, Helgoland, Germany Marine phages have an astounding global abundance and ecological impact. However, little knowledge is derived from phage genomes, as most of the open reading frames in their small genomes are unknown, novel proteins. To infer potential functional and ecological relevance of sequenced marine Pseudoalteromonas phage H105/1, two strategies were used. First, similarity searches were extended to include six viral and bacterial metagenomes paired with their respective environmental contextual data. This approach revealed ‘ecogenomic’ patterns of Pseudoalteromo- nas phage H105/1, such as its estuarine origin. Second, intrinsic genome signatures (phylogenetic, codon adaptation and tetranucleotide (tetra) frequencies) were evaluated on a resolved intra- genomic level to shed light on the evolution of phage functional modules. On the basis of differential codon adaptation of Phage H105/1 proteins to the sequenced Pseudoalteromonas spp., regions of the phage genome with the most ‘host’-adapted proteins also have the strongest bacterial tetra signature, whereas the least ‘host’-adapted proteins have the strongest phage tetra signature. Such a pattern may reflect the evolutionary history of the respective phage proteins and functional modules. -

Dynamics of Bacterial Communities in Two Unpolluted Soils After Spiking with Phenanthrene: Soil Type Specific and Common Responders
ORIGINAL RESEARCH ARTICLE published: 21 August 2012 doi: 10.3389/fmicb.2012.00290 Dynamics of bacterial communities in two unpolluted soils after spiking with phenanthrene: soil type specific and common responders Guo-Chun Ding, Holger Heuer and Kornelia Smalla* Julius Kühn-Institut, Federal Research Centre for Cultivated Plants, Braunschweig, Germany Edited by: Considering their key role for ecosystem processes, it is important to understand the Matthew Fields, Montana State response of microbial communities in unpolluted soils to pollution with polycyclic aro- University, USA matic hydrocarbons (PAH). Phenanthrene, a model compound for PAH, was spiked to a Reviewed by: Uwe Strotmann, Fachhochschule Cambisol and a Luvisol soil. Total community DNA from phenanthrene-spiked and con- Gelsenkirchen, Germany trol soils collected on days 0, 21, and 63 were analyzed based on PCR-amplified 16S Yingying Wang, Nankai University, rRNA gene fragments. Denaturing gradient gel electrophoresis (DGGE) fingerprints of China bacterial communities increasingly deviated with time between spiked and control soils. *Correspondence: In taxon specific DGGE, significant responses of Alphaproteobacteria and Actinobacte- Kornelia Smalla, Julius Kühn-Institut, Federal Research Centre for ria became only detectable after 63 days, while significant effects on Betaproteobacteria Cultivated Plants, Messeweg 11-12, were detectable in both soils after 21 days. Comparison of the taxonomic distribution of D-38104 Braunschweig, Germany. bacteria in spiked and control soils on day 63 as revealed by pyrosequencing indicated soil e-mail: [email protected] type specific negative effects of phenanthrene on several taxa, many of them belonging to the Gamma-, Beta-, or Deltaproteobacteria. Bacterial richness and evenness decreased in spiked soils. -

Acidovorax Citrulli
Bulletin OEPP/EPPO Bulletin (2016) 46 (3), 444–462 ISSN 0250-8052. DOI: 10.1111/epp.12330 European and Mediterranean Plant Protection Organization Organisation Europe´enne et Me´diterrane´enne pour la Protection des Plantes PM 7/127 (1) Diagnostics Diagnostic PM 7/127 (1) Acidovorax citrulli Specific scope Specific approval and amendment This Standard describes a diagnostic protocol for Approved in 2016-09. Acidovorax citrulli.1 This Standard should be used in conjunction with PM 7/76 Use of EPPO diagnostic protocols. strain, were mainly isolated from non-watermelon, cucurbit 1. Introduction hosts such as cantaloupe melon (Cucumis melo var. Acidovorax citrulli is the causal agent of bacterial fruit cantalupensis), cucumber (Cucumis sativus), honeydew blotch and seedling blight of cucurbits (Webb & Goth, melon (Cucumis melo var. indorus), squash and pumpkin 1965; Schaad et al., 1978). This disease was sporadic until (Cucurbita pepo, Cucurbita maxima and Cucurbita the late 1980s (Wall & Santos, 1988), but recurrent epi- moschata) whereas Group II isolates were mainly recovered demics have been reported in the last 20 years (Zhang & from watermelon (Walcott et al., 2000, 2004; Burdman Rhodes, 1990; Somodi et al., 1991; Latin & Hopkins, et al., 2005). While Group I isolates were moderately 1995; Demir, 1996; Assis et al., 1999; Langston et al., aggressive on a range of cucurbit hosts, Group II isolates 1999; O’Brien & Martin, 1999; Burdman et al., 2005; Har- were highly aggressive on watermelon but moderately ighi, 2007; Holeva et al., 2010; Popovic & Ivanovic, 2015). aggressive on non-watermelon hosts (Walcott et al., 2000, The disease is particularly severe on watermelon (Citrullus 2004). -

Polaromonas Eurypsychrophila Sp. Nov., Isolated from an Ice Core
International Journal of Systematic and Evolutionary Microbiology (2016), 66, 2497–2501 DOI 10.1099/ijsem.0.001079 Polaromonas eurypsychrophila sp. nov., isolated from an ice core Tingting Xing,1 Tandong Yao,2,3 Yongqin Liu,2,3 Ninglian Wang,2,4 Bainqing Xu,2,3 Liang Shen,1 Zhengquan Gu,1 Bixi Gu,1 Hongcan Liu5 and Yuguang Zhou5 Correspondence 1Key Laboratory of Alpine Ecology and Biodiversity, Institute of Tibetan Plateau Research, Yongqin Liu Chinese Academy of Sciences, Beijing 100101, China [email protected] 2CAS Center for Excellence in Tibetan Plateau Earth Sciences, Chinese Academy of Sciences, Beijing 100101, China 3Key Laboratory of Tibetan Environment Changes and Land Surface Processes, Institute of Tibetan Plateau Research, Chinese Academy of Sciences, Beijing 100101, China 4College of Urban and Environmental Science, Northwest University, Xi’an 710000, China 5Institute of Microbiology, China General Microbiological Culture Collection Center, Chinese Academy of Sciences, Beijing 100101, China A Gram-stain-negative, aerobic, rod-shaped, beige bacterium, strain B717-2T, was isolated from an ice core drilled from Muztagh Glacier on the Tibetan Plateau, China. According to phylogenetic analyses based on 16S rRNA gene sequences, the novel strain was related most closely to Polaromonas vacuolataand shared 97.7 % similarity with the type strain of this species. It grew optimally at pH 7, at 15 C and with 2 % (w/v) NaCl. Major polar lipids were phosphatidylethanolamine, phosphatidylglycerol and diphosphatidylglycerol. The major fatty acids were summed feature 3 (C16 : 1!7c and/or iso-C15 : 0 2-OH), summed feature 8 (C18 : 1!7c, C18 : 1!6c) and C16 : 0. -

Microbial Degradation of Organic Micropollutants in Hyporheic Zone Sediments
Microbial degradation of organic micropollutants in hyporheic zone sediments Dissertation To obtain the Academic Degree Doctor rerum naturalium (Dr. rer. nat.) Submitted to the Faculty of Biology, Chemistry, and Geosciences of the University of Bayreuth by Cyrus Rutere Bayreuth, May 2020 This doctoral thesis was prepared at the Department of Ecological Microbiology – University of Bayreuth and AG Horn – Institute of Microbiology, Leibniz University Hannover, from August 2015 until April 2020, and was supervised by Prof. Dr. Marcus. A. Horn. This is a full reprint of the dissertation submitted to obtain the academic degree of Doctor of Natural Sciences (Dr. rer. nat.) and approved by the Faculty of Biology, Chemistry, and Geosciences of the University of Bayreuth. Date of submission: 11. May 2020 Date of defense: 23. July 2020 Acting dean: Prof. Dr. Matthias Breuning Doctoral committee: Prof. Dr. Marcus. A. Horn (reviewer) Prof. Harold L. Drake, PhD (reviewer) Prof. Dr. Gerhard Rambold (chairman) Prof. Dr. Stefan Peiffer In the battle between the stream and the rock, the stream always wins, not through strength but by perseverance. Harriett Jackson Brown Jr. CONTENTS CONTENTS CONTENTS ............................................................................................................................ i FIGURES.............................................................................................................................. vi TABLES .............................................................................................................................. -

Gene Transfers from Diverse Bacteria Compensate for Reductive Genome Evolution in the Chromatophore of Paulinella Chromatophora
Gene transfers from diverse bacteria compensate for reductive genome evolution in the chromatophore of Paulinella chromatophora Eva C. M. Nowacka,b,1, Dana C. Pricec, Debashish Bhattacharyad, Anna Singerb, Michael Melkoniane, and Arthur R. Grossmana aDepartment of Plant Biology, Carnegie Institution for Science, Stanford, CA 94305; bDepartment of Biology, Heinrich-Heine-Universität Düsseldorf, 40225 Dusseldorf, Germany; cDepartment of Plant Biology and Pathology, Rutgers, The State University of New Jersey, New Brunswick, NJ 08901; dDepartment of Ecology, Evolution and Natural Resources, Rutgers, The State University of New Jersey, New Brunswick, NJ 08901; and eBiozentrum, Universität zu Köln, 50674 Koln, Germany Edited by John M. Archibald, Dalhousie University, Halifax, Canada, and accepted by Editorial Board Member W. Ford Doolittle September 6, 2016 (received for review May 19, 2016) Plastids, the photosynthetic organelles, originated >1 billion y ago horizontal gene transfers (HGTs) from cooccurring intracellular via the endosymbiosis of a cyanobacterium. The resulting prolifer- bacteria also supplied genes that facilitated plastid establishment ation of primary producers fundamentally changed global ecology. (6). However, the extent and sources of HGTs and their impor- Endosymbiotic gene transfer (EGT) from the intracellular cyanobac- tance to organelle evolution remain controversial topics (7, 8). terium to the nucleus is widely recognized as a critical factor in the The chromatophore of the cercozoan amoeba Paulinella evolution of photosynthetic eukaryotes. The contribution of hori- chromatophora (Rhizaria) represents the only known case of zontal gene transfers (HGTs) from other bacteria to plastid estab- acquisition of a photosynthetic organelle other than the primary lishment remains more controversial. A novel perspective on this endosymbiosis that gave rise to the Archaeplastida (9). -

Complete Assembly of the Genome of an Acidovorax Citrulli Strain Reveals a Naturally Occurring Plasmid in This Species
fmicb-10-01400 June 19, 2019 Time: 15:19 # 1 ORIGINAL RESEARCH published: 20 June 2019 doi: 10.3389/fmicb.2019.01400 Complete Assembly of the Genome of an Acidovorax citrulli Strain Reveals a Naturally Occurring Plasmid in This Species Rongzhi Yang1, Diego Santos Garcia2, Francisco Pérez Montaño1,3, Gustavo Mateus da Silva1, Mei Zhao4, Irene Jiménez Guerrero1, Tally Rosenberg1, Gong Chen4, Inbar Plaschkes5, Shai Morin2, Ron Walcott4 and Saul Burdman1* 1 Department of Plant Pathology and Microbiology, The Robert H. Smith Faculty of Agriculture, Food and Environment, The Hebrew University of Jerusalem, Rehovot, Israel, 2 Department of Entomology, The Robert H. Smith Faculty of Agriculture, Food and Environment, The Hebrew University of Jerusalem, Rehovot, Israel, 3 Department of Microbiology, University of Seville, Seville, Spain, 4 Department of Plant Pathology, University of Georgia, Athens, GA, United States, 5 Bioinformatics Unit, The Robert H. Smith Faculty of Agriculture, Food and Environment, The Hebrew University of Jerusalem, Rehovot, Israel Edited by: Martin G. Klotz, Washington State University, Acidovorax citrulli is the causal agent of bacterial fruit blotch (BFB), a serious threat United States to cucurbit crop production worldwide. Based on genetic and phenotypic properties, Reviewed by: A. citrulli strains are divided into two major groups: group I strains have been generally Robert Wilson Jackson, isolated from melon and other non-watermelon cucurbits, while group II strains are University of Reading, United Kingdom closely associated with watermelon. In a previous study, we reported the genome Tingchang Zhao, of the group I model strain, M6. At that time, the M6 genome was sequenced by Chinese Academy of Agricultural Sciences, China MiSeq Illumina technology, with reads assembled into 139 contigs.