Uno De Los Nuestros Descifrando El Código
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Biochemistrystanford00kornrich.Pdf
University of California Berkeley Regional Oral History Office University of California The Bancroft Library Berkeley, California Program in the History of the Biosciences and Biotechnology Arthur Kornberg, M.D. BIOCHEMISTRY AT STANFORD, BIOTECHNOLOGY AT DNAX With an Introduction by Joshua Lederberg Interviews Conducted by Sally Smith Hughes, Ph.D. in 1997 Copyright 1998 by The Regents of the University of California Since 1954 the Regional Oral History Office has been interviewing leading participants in or well-placed witnesses to major events in the development of Northern California, the West, and the Nation. Oral history is a method of collecting historical information through tape-recorded interviews between a narrator with firsthand knowledge of historically significant events and a well- informed interviewer, with the goal of preserving substantive additions to the historical record. The tape recording is transcribed, lightly edited for continuity and clarity, and reviewed by the interviewee. The corrected manuscript is indexed, bound with photographs and illustrative materials, and placed in The Bancroft Library at the University of California, Berkeley, and in other research collections for scholarly use. Because it is primary material, oral history is not intended to present the final, verified, or complete narrative of events. It is a spoken account, offered by the interviewee in response to questioning, and as such it is reflective, partisan, deeply involved, and irreplaceable. ************************************ All uses of this manuscript are covered by a legal agreement between The Regents of the University of California and Arthur Kornberg, M.D., dated June 18, 1997. The manuscript is thereby made available for research purposes. All literary rights in the manuscript, including the right to publish, are reserved to The Bancroft Library of the University of California, Berkeley. -

Lecture Program
EARL W. SUTHERLAND LECTURE EARL W. SUTHERLAND LECTURE The Earl W. Sutherland Lecture Series was established by the SPONSORED BY: Department of Molecular Physiology and Biophysics in 1997 DEPARTMENT OF MOLECULAR PHYSIOLOGY AND BIOPHYSICS to honor Dr. Sutherland, a former member of this department and winner of the 1971 Nobel Prize in Physiology or Medicine. This series highlights important advances in cell signaling. ROBERT J. LEFKOWITZ, MD NOBEL PRIZE IN CHEMISTRY, 2012 SPEAKERS IN THIS SERIES HAVE INCLUDED: SEVEN TRANSMEMBRANE RECEPTORS Edmond H. Fischer (1997) Alfred G. Gilman (1999) Ferid Murad (2001) Louis J. Ignarro (2003) MARCH 31, 2016 Paul Greengard (2007) 4:00 P.M. 208 LIGHT HALL Eric Kandel (2009) Roger Tsien (2011) Michael S. Brown (2013) 867-2923-Institution-Discovery Lecture Series-Lefkowitz-BK-CH.indd 1 3/11/16 9:39 AM EARL W. SUTHERLAND, 1915-1974 ROBERT J. LEFKOWITZ, MD JAMES B. DUKE PROFESSOR, Earl W. Sutherland grew up in Burlingame, Kansas, a small farming community DUKE UNIVERSITY MEDICAL CENTER that nourished his love for the outdoors and fishing, which he retained throughout INVESTIGATOR, HOWARD HUGHES MEDICAL INSTITUTE his life. He graduated from Washburn College in 1937 and then received his MEMBER, NATIONAL ACADEMY OF SCIENCES M.D. from Washington University School of Medicine in 1942. After serving as a MEMBER, INSTITUTE OF MEDICINE medical officer during World War II, he returned to Washington University to train NOBEL PRIZE IN CHEMISTRY, 2012 with Carl and Gerty Cori. During those years he was influenced by his interactions with such eminent scientists as Louis Leloir, Herman Kalckar, Severo Ochoa, Arthur Kornberg, Christian deDuve, Sidney Colowick, Edwin Krebs, Theodore Robert J. -

| Sydney Brenner |
| SYDNEY BRENNER | TOP THREE AWARDS • Nobel Prize in Physiology, 2002 • Albert Lasker Special Achievement Award, 2000 • National Order of Mapungubwe (Gold), 2004 DEFINING MOMENT To view the DNA model for the first time. 32 |LEGENDS OF SOUTH AFRICAN SCIENCE| A LIFE DEDICATED TO SCIENCE C. ELEGANS WORK In the more than eight decades that Nobel Laureate, Prof Sydney Brenner, “To start with we propose to identify every cell in the worm and trace line- has all-consumingly devoted his life to science, he twice wrote powerful age. We shall also investigate the constancy of development and study proposals of no longer than a page. Short but sweet, these kick-started the its control by looking for mutants,” is how Brenner ended his proposal on two projects that are part of his lasting legacy. Caenorhabditis elegans to the UK Medical Research Council in October 1963. He was looking for a new challenge after already having helped to The first was to request funding to study a worm, because he saw in the show that genetic code is composed of non-overlapping triplets and that nematode Caenorhabditis elegans the ideal genetic model organism. messenger ribonucleic acid (mRNA) exists. He was right, and received the Nobel Prize for his efforts. The other pro- posal, which set out how Singapore could become a hub for biomedical His first paper on C. elegans appeared in Genetics in 1974, and in all, the research, earned him the title of “mentor to a nation’s science ambitions”. work took about 20 years to reach its full potential. -
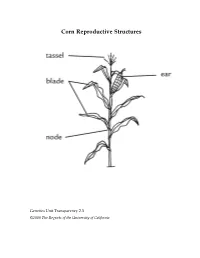
Corn Reproductive Structures
Corn Reproductive Structures Genetics Unit Transparency 2.3 ©2008 The Regents of the University of California Creating a Punnett Square Genetics Unit Transparency 2.4 ©2008 The Regents of the University of California Case Study Summary Sheet Case Study Type of Benefits Risks and concerns Remaining questions Genetic Modification Genetics Unit Transparency 4.1 ©2008 The Regents of the University of California Class Data for Rice Breeding Simulation aromatic, non-aromatic, aromatic, non-aromatic ,flood-tolear nt flood-tolerant flood-intolerant flood-intolerant AAFF AAFf AaFF AaFf aaFF aaFf AAff Aaff aaff Group 1 2 3 4 5 6 7 8 Genetics Unit Transparency 5.1 ©2008 The Regents of the University of California Three Types of Cells Genetics Unit Transparency 6.1 ©2008 The Regents of the University of California DNA base percentages in a variety of organisms Source of A T G C DNA Human 31.0% 31.5% 19.1% 18.4% Mouse 29.1% 29.0% 21.1% 21.1% Frog 26.3% 26.4% 23.5% 23.8% Fruit fly 27.3% 27.6% 22.5% 22.5% Corn 25.6% 25.3% 24.5% 24.6% E. coli 24.6% 24.3% 25.5% 25.6% Genetics Unit Transparency 7.1 ©2008 The Regents of the University of California Matthew Meselson and Franklin Stahl’s Experiment to Investigate DNA Replication First: Scientists James Watson and Francis Crick propose a method of semi-conservative replication in their paper on the structure of DNA. However, they have no data to support their hypothesis. Next: Matthew Meselson and Franklin Stahl use the procedure that follows to investigate DNA created by the process of DNA Replication. -

Asilomar Conference on Laboratory Precautions When Conducting Recombinant DNA Research – Case Summary M.J
International Dimensions of Ethics Education in Science and Engineering Case Study Series Asilomar Conference on Laboratory Precautions When Conducting Recombinant DNA Research – Case Summary M.J. Peterson with research assistance from Paul White Version 1, June 2010 Safe laboratory practices are an important element in the responsible conduct of research. They protect scientists, research assistants, laboratory technicians, and others from hazards that can arise during experimentation. The particular hazards that might arise vary according to the type of experimentation: the explosions or toxic gases that could result from chemical experiments are prevented or contained by different kinds of measures then are used to limit the health hazards posed by working with radioactive isotopes. In most areas, scientists are allowed – and even encouraged – to decide for themselves what experiments to undertake and how to construct and run their labs. In 1970, there were three main exceptions, and they applied more to questions of how to carry out research than to selecting topics for research: use of radioactive materials, use of tumor-causing viruses, and use of human subjects. In these areas, levels of risk to persons inside and outside the lab were perceived as great enough to justify having the government, either through the regular bureaucracy or through scientific agencies, set and enforce mandatory safety standards. When genetics was poised at the threshold of recombinant DNA research and genetic manipulation techniques in the 1960s, the hazards such work might pose to lab personnel or to others were unknown. Many scientists and others believed they were likely to be significant. The primary concern in public discussion was whether hybrid organisms bred from recombinant DNA would cause new diseases or create varieties of plants or animals that would overwhelm and irretrievably change the natural environment. -

Barbara Mcclintock's World
Barbara McClintock’s World Timeline adapted from Dolan DNA Learning Center exhibition 1902-1908 Barbara McClintock is born in Hartford, Connecticut, the third of four children of Sarah and Thomas Henry McClintock, a physician. She spends periods of her childhood in Massachusetts with her paternal aunt and uncle. Barbara at about age five. This prim and proper picture betrays the fact that she was, in fact, a self-reliant tomboy. Barbara’s individualism and self-sufficiency was apparent even in infancy. When Barbara was four months old, her parents changed her birth name, Eleanor, which they considered too delicate and feminine for such a rugged child. In grade school, Barbara persuaded her mother to have matching bloomers (shorts) made for her dresses – so she could more easily join her brother Tom in tree climbing, baseball, volleyball, My father tells me that at the and football. age of five I asked for a set of tools. He My mother used to did not get me the tools that you get for an adult; he put a pillow on the floor and give got me tools that would fit in my hands, and I didn’t me one toy and just leave me there. think they were adequate. Though I didn’t want to tell She said I didn’t cry, didn’t call for him that, they were not the tools I wanted. I wanted anything. real tools not tools for children. 1908-1918 McClintock’s family moves to Brooklyn in 1908, where she attends elementary and secondary school. In 1918, she graduates one semester early from Erasmus Hall High School in Brooklyn. -

The Contributions of George Beadle and Edward Tatum
| PERSPECTIVES Biochemical Genetics and Molecular Biology: The Contributions of George Beadle and Edward Tatum Bernard S. Strauss1 Department of Molecular Genetics and Cell Biology, The University of Chicago, Chicago, Illinois 60637 KEYWORDS George Beadle; Edward Tatum; Boris Ephrussi; gene action; history It will concern us particularly to take note of those cases in Genetics in the Early 1940s which men not only solved a problem but had to alter their mentality in the process, or at least discovered afterwards By the end of the 1930s, geneticists had developed a sophis- that the solution involved a change in their mental approach ticated, self-contained science. In particular, they were able (Butterfield 1962). to predict the patterns of inheritance of a variety of charac- teristics, most morphological in nature, in a variety of or- EVENTY-FIVE years ago, George Beadle and Edward ganisms although the favorites at the time were clearly Tatum published their method for producing nutritional S Drosophila andcorn(Zea mays). These characteristics were mutants in Neurospora crassa. Their study signaled the start of determined by mysterious entities known as “genes,” known a new era in experimental biology, but its significance is to be located at particular positions on the chromosomes. generally misunderstood today. The importance of the work Furthermore, a variety of peculiar patterns of inheritance is usually summarized as providing support for the “one gene– could be accounted for by alteration in chromosome struc- one enzyme” hypothesis, but its major value actually lay both ture and number with predictions as to inheritance pattern in providing a general methodology for the investigation of being quantitative and statistical. -

Michael S. Brown, MD
DISTINGUISHED PHYSICIANS AND Michael S. Brown, M.D. Sir Richard Roberts, Ph.D. Winner, 1985 Nobel Prize in Physiology or Medicine Winner, 1993 Nobel Prize in Physiology or Medicine MEDICAL SCIENTISTS MENTORING Winner, 1988 Presidential National Medal of Science A globally prominent biochemist and molecular biologist, DELEGATES HAVE INCLUDED... Dr. Brown received the world’s most prestigious medical Dr. Roberts was awarded the Nobel Prize for his prize for his work describing the regulation of the groundbreaking contribution to discovering RNA splicing. cholesterol metabolism. His work laid the foundation for Dr. Roberts is dedicating his future research to GMO crops the class of drugs now called statins taken daily by more than 20 million and food sources, and demonstrating the effect they have on humanity. — GRANDg MASTERS — people worldwide. Ferid Murad, M.D., Ph.D. Mario Capecchi, Ph.D. Boris D. Lushniak, M.D., M.P.H Winner, 1998 Nobel Prize in Physiology or Medicine Academy Science Director The Surgeon General of the United States (acting, 2013-2014) Winner, 2007 Nobel Prize in Physiology or Medicine A world-renowned pioneer in biochemistry, Dr. Murad’s Winner, 2001 National Medal of Science Rear Admiral Lushniak, M.D., M.P.H., was the United award-winning research demonstrated that nitroglycerin Winner, 2001 Lasker Award States’ leading spokesperson on matters of public health, and related drugs help patients with heart conditions by Winner, 2003 Wolf Prize in Medicine overseeing the operations of the U.S. Public Health Service releasing nitric oxide into the body, thus relaxing smooth Mario Capecchi, Ph.D., a biophysicist, is a Distinguished Commissioned Corps, which consists of approximately muscles by elevating intracellular cyclic GMP, leading to vasodilation and Professor of Human Genetics at the University of Utah School of Medicine. -

Clinical Molecular Genetics in the Uk C.1975–C.2000
CLINICAL MOLECULAR GENETICS IN THE UK c.1975–c.2000 The transcript of a Witness Seminar held by the History of Modern Biomedicine Research Group, Queen Mary, University of London, on 5 February 2013 Edited by E M Jones and E M Tansey Volume 48 2014 ©The Trustee of the Wellcome Trust, London, 2014 First published by Queen Mary, University of London, 2014 The History of Modern Biomedicine Research Group is funded by the Wellcome Trust, which is a registered charity, no. 210183. ISBN 978 0 90223 888 6 All volumes are freely available online at www.history.qmul.ac.uk/research/modbiomed/ wellcome_witnesses/ Please cite as: Jones E M, Tansey E M. (eds) (2014) Clinical Molecular Genetics in the UK c.1975–c.2000. Wellcome Witnesses to Contemporary Medicine, vol. 48. London: Queen Mary, University of London. CONTENTS What is a Witness Seminar? v Acknowledgements E M Tansey and E M Jones vii Illustrations and credits ix Abbreviations xi Ancillary guides xiii Introduction Professor Bob Williamson xv Transcript Edited by E M Jones and E M Tansey 1 Appendix 1 Photograph, with key, of delegates attending The Molecular Biology of Thalassaemia conference in Kolimbari, Crete, 1978 88 Appendix 2 Extracts from the University of Leiden postgraduate course Restriction Fragment Length Polymorphisms and Human Genetics, 1982 91 Appendix 3 Archival material of the Clinical Molecular Genetics Society 95 Biographical notes 101 References 113 Index 131 Witness Seminars: Meetings and Publications 143 WHAT IS A WITNESS SEMINAR? The Witness Seminar is a specialized form of oral history, where several individuals associated with a particular set of circumstances or events are invited to meet together to discuss, debate, and agree or disagree about their memories. -

MCDB 5220 Methods and Logics April 21 2015 Marcelo Bassalo
Cracking the Genetic Code MCDB 5220 Methods and Logics April 21 2015 Marcelo Bassalo The DNA Saga… so far Important contributions for cracking the genetic code: • The “transforming principle” (1928) Frederick Griffith The DNA Saga… so far Important contributions for cracking the genetic code: • The “transforming principle” (1928) • The nature of the transforming principle: DNA (1944 - 1952) Oswald Avery Alfred Hershey Martha Chase The DNA Saga… so far Important contributions for cracking the genetic code: • The “transforming principle” (1928) • The nature of the transforming principle: DNA (1944 - 1952) • X-ray diffraction and the structure of proteins (1951) Linus Carl Pauling The DNA Saga… so far Important contributions for cracking the genetic code: • The “transforming principle” (1928) • The nature of the transforming principle: DNA (1944 - 1952) • X-ray diffraction and the structure of proteins (1951) • The structure of DNA (1953) James Watson and Francis Crick The DNA Saga… so far Important contributions for cracking the genetic code: • The “transforming principle” (1928) • The nature of the transforming principle: DNA (1944 - 1952) • X-ray diffraction and the structure of proteins (1951) • The structure of DNA (1953) How is DNA (4 nucleotides) the genetic material while proteins (20 amino acids) are the building blocks? ? DNA Protein ? The Coding Craze ? DNA Protein What was already known? • DNA resides inside the nucleus - DNA is not the carrier • Protein synthesis occur in the cytoplasm through ribosomes {• Only RNA is associated with ribosomes (no DNA) - rRNA is not the carrier { • Ribosomal RNA (rRNA) was a homogeneous population The “messenger RNA” hypothesis François Jacob Jacques Monod The Coding Craze ? DNA RNA Protein RNA Tie Club Table from Wikipedia The Coding Craze Who won the race Marshall Nirenberg J. -

Alexander Rich (1924–2015) Biologist Who Discovered Ribosome Clusters and ‘Left-Handed’ DNA
OBITUARY COMMENT Alexander Rich (1924–2015) Biologist who discovered ribosome clusters and ‘left-handed’ DNA. first came across Alexander Rich worked out the structure of Rich in 1963. He was on the a Z-DNA fragment bound to an cover of that year’s 13 May issue RNA-editing enzyme. He and his Iof Newsweek with his PhD student colleagues also showed how the JOSIAH D. RICH Jonathan Warner. The two of them pathogenicity of the vaccinia virus, had just discovered clusters of ribo- and probably of the smallpox virus, somes called polysomes — crucial correlated with a virus-specific pro- components involved in the build- tein binding to the host’s Z-DNA. ing of proteins. Rich’s interest in the latest Rich, who died on 27 April, was discoveries across diverse disciplines born in 1924 in Hartford, Connecticut was irrepressible. During the 1970s, to immigrant parents from Russia and he worked as an adviser for NASA, Eastern Europe. He grew up during weighing in on projects exploring the Great Depression in Springfield, the possible existence of life on Mars. Massachusetts, attending a technical He also ventured into biotechnology secondary school by day, and work- and co-founded three companies: ing nights at a local rifle factory. At Repligen, Alkermes, and in his 80s, one point, his family went to live at a 3-D Matrix. local YMCA club after being evicted Rich received numerous honorary from their home. Against the odds, degrees and awards, including the US Rich made it to Harvard University National Medal of Science, presented in Cambridge, Massachusetts, grad- to him in 1995 by then US President uating with a bachelor’s degree in Bill Clinton. -

Cambridge's 92 Nobel Prize Winners Part 4 - 1996 to 2015: from Stem Cell Breakthrough to IVF
Cambridge's 92 Nobel Prize winners part 4 - 1996 to 2015: from stem cell breakthrough to IVF By Cambridge News | Posted: February 01, 2016 Some of Cambridge's most recent Nobel winners Over the last four weeks the News has been rounding up all of Cambridge's 92 Nobel Laureates, which this week comes right up to the present day. From the early giants of physics like JJ Thomson and Ernest Rutherford to the modern-day biochemists unlocking the secrets of our genome, we've covered the length and breadth of scientific discovery, as well as hugely influential figures in economics, literature and politics. What has stood out is the importance of collaboration; while outstanding individuals have always shone, Cambridge has consistently achieved where experts have come together to bounce their ideas off each other. Key figures like Max Perutz, Alan Hodgkin and Fred Sanger have not only won their own Nobels, but are regularly cited by future winners as their inspiration, as their students went on to push at the boundaries they established. In the final part of our feature we cover the last 20 years, when Cambridge has won an average of a Nobel Prize a year, and shows no sign of slowing down, with ground-breaking research still taking place in our midst today. The Gender Pay Gap Sale! Shop Online to get 13.9% off From 8 - 11 March, get 13.9% off 1,000s of items, it highlights the pay gap between men & women in the UK. Shop the Gender Pay Gap Sale – now. Promoted by Oxfam 1.1996 James Mirrlees, Trinity College: Prize in Economics, for studying behaviour in the absence of complete information As a schoolboy in Galloway, Scotland, Mirrlees was in line for a Cambridge scholarship, but was forced to change his plans when on the weekend of his interview he was rushed to hospital with peritonitis.