Genomanalyse Von Prodiamesa Olivacea
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Chironominae 8.1
CHIRONOMINAE 8.1 SUBFAMILY CHIRONOMINAE 8 DIAGNOSIS: Antennae 4-8 segmented, rarely reduced. Labrum with S I simple, palmate or plumose; S II simple, apically fringed or plumose; S III simple; S IV normal or sometimes on pedicel. Labral lamellae usually well developed, but reduced or absent in some taxa. Mentum usually with 8-16 well sclerotized teeth; sometimes central teeth or entire mentum pale or poorly sclerotized; rarely teeth fewer than 8 or modified as seta-like projections. Ventromental plates well developed and usually striate, but striae reduced or vestigial in some taxa; beard absent. Prementum without dense brushes of setae. Body usually with anterior and posterior parapods and procerci well developed; setal fringe not present, but sometimes with bifurcate pectinate setae. Penultimate segment sometimes with 1-2 pairs of ventral tubules; antepenultimate segment sometimes with lateral tubules. Anal tubules usually present, reduced in brackish water and marine taxa. NOTESTES: Usually the most abundant subfamily (in terms of individuals and taxa) found on the Coastal Plain of the Southeast. Found in fresh, brackish and salt water (at least one truly marine genus). Most larvae build silken tubes in or on substrate; some mine in plants, dead wood or sediments; some are free- living; some build transportable cases. Many larvae feed by spinning silk catch-nets, allowing them to fill with detritus, etc., and then ingesting the net; some taxa are grazers; some are predacious. Larvae of several taxa (especially Chironomus) have haemoglobin that gives them a red color and the ability to live in low oxygen conditions. With only one exception (Skutzia), at the generic level the larvae of all described (as adults) southeastern Chironominae are known. -

Mogren Etal 2012.Pdf
Science of the Total Environment 425 (2012) 60–65 Contents lists available at SciVerse ScienceDirect Science of the Total Environment journal homepage: www.elsevier.com/locate/scitotenv Survival, reproduction, and arsenic body burdens in Chironomus riparius exposed to arsenate and phosphate Christina L. Mogren a,⁎, Guntram R. von Kiparski b, David R. Parker b, John T. Trumble a a Department of Entomology, University of California, Riverside, 900 University Ave, Riverside, CA 92521, United States b Department of Environmental Science, University of California, Riverside, 900 University Ave, Riverside, CA 92521, United States article info abstract Article history: Despite the increasing awareness of arsenic (As) contamination in surface waters worldwide, little is Received 12 January 2012 known about how As alone and in the presence of other chemicals affects aquatic insects. Larvae of Received in revised form 5 March 2012 Chironomus riparius were exposed in a laboratory investigation to factorial combinations of 0, 0.13, 2.0, 5.3, Accepted 6 March 2012 − − and 13 μmol As l 1 and 0, 0.15, and 15 μmol PO l 1 throughout development from first instar to pupal Available online 29 March 2012 4 emergence. The time between male and female emergence increased from 1.8±0.17 days to 2.9± 0.34 days with exposure at higher As levels. The highest As exposure also decreased the number of eggs Keywords: Aquatic per egg mass, which may affect population maintenance. For these parameters, there was no effect from Arsenic PO4, and no interaction between As and PO4. Total As determination of larval and adult tissues was conducted Bioaccumulation using Hydride Generated Atomic Absorption Spectroscopy (HGAAS) and revealed concentrations ranging Metalloid from 2.48±0.363 to 30.5±0.473 μg/g and 1.03±0.286 to 8.97±0.662 μg/g, respectively, indicating elimina- Sublethal toxicity tion of approximately 72% of total As body burdens between the fourth instar and adult stages. -

Chironomidae Hirschkopf
Literatur Chironomidae Gesäuse U.A. zur Bestimmung und Ermittlung der Autökologie herangezogene Literatur: Albu, P. (1972): Două specii de Chironomide noi pentru ştiinţă în masivul Retezat.- St. şi Cerc. Biol., Seria Zoologie, 24: 15-20. Andersen, T.; Mendes, H.F. (2002): Neotropical and Mexican Mesosmittia Brundin, with the description of four new species (Insecta, Diptera, Chironomidae).- Spixiana, 25(2): 141-155. Andersen, T.; Sæther, O.A. (1993): Lerheimia, a new genus of Orthocladiinae from Africa (Diptera: Chironomidae).- Spixiana, 16: 105-112. Andersen, T.; Sæther, O.A.; Mendes, H.F. (2010): Neotropical Allocladius Kieffer, 1913 and Pseudosmittia Edwards, 1932 (Diptera: Chironomidae).- Zootaxa, 2472: 1-77. Baranov, V.A. (2011): New and rare species of Orthocladiinae (Diptera, Chironomidae) from the Crimea, Ukraine.- Vestnik zoologii, 45(5): 405-410. Boggero, A.; Zaupa, S.; Rossaro, B. (2014): Pseudosmittia fabioi sp. n., a new species from Sardinia (Diptera: Chironomidae, Orthocladiinae).- Journal of Entomological and Acarological Research, [S.l.],46(1): 1-5. Brundin, L. (1947): Zur Kenntnis der schwedischen Chironomiden.- Arkiv för Zoologi, 39 A(3): 1- 95. Brundin, L. (1956): Zur Systematik der Orthocladiinae (Dipt. Chironomidae).- Rep. Inst. Freshwat. Drottningholm 37: 5-185. Casas, J.J.; Laville, H. (1990): Micropsectra seguyi, n. sp. du groupe attenuata Reiss (Diptera: Chironomidae) de la Sierra Nevada (Espagne).- Annls Soc. ent. Fr. (N.S.), 26(3): 421-425. Caspers, N. (1983): Chironomiden-Emergenz zweier Lunzer Bäche, 1972.- Arch. Hydrobiol. Suppl. 65: 484-549. Caspers, N. (1987): Chaetocladius insolitus sp. n. (Diptera: Chironomidae) from Lunz, Austria. In: Saether, O.A. (Ed.): A conspectus of contemporary studies in Chironomidae (Diptera). -
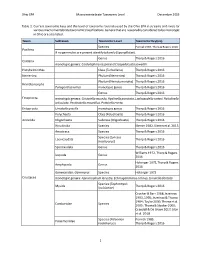
Ohio EPA Macroinvertebrate Taxonomic Level December 2019 1 Table 1. Current Taxonomic Keys and the Level of Taxonomy Routinely U
Ohio EPA Macroinvertebrate Taxonomic Level December 2019 Table 1. Current taxonomic keys and the level of taxonomy routinely used by the Ohio EPA in streams and rivers for various macroinvertebrate taxonomic classifications. Genera that are reasonably considered to be monotypic in Ohio are also listed. Taxon Subtaxon Taxonomic Level Taxonomic Key(ies) Species Pennak 1989, Thorp & Rogers 2016 Porifera If no gemmules are present identify to family (Spongillidae). Genus Thorp & Rogers 2016 Cnidaria monotypic genera: Cordylophora caspia and Craspedacusta sowerbii Platyhelminthes Class (Turbellaria) Thorp & Rogers 2016 Nemertea Phylum (Nemertea) Thorp & Rogers 2016 Phylum (Nematomorpha) Thorp & Rogers 2016 Nematomorpha Paragordius varius monotypic genus Thorp & Rogers 2016 Genus Thorp & Rogers 2016 Ectoprocta monotypic genera: Cristatella mucedo, Hyalinella punctata, Lophopodella carteri, Paludicella articulata, Pectinatella magnifica, Pottsiella erecta Entoprocta Urnatella gracilis monotypic genus Thorp & Rogers 2016 Polychaeta Class (Polychaeta) Thorp & Rogers 2016 Annelida Oligochaeta Subclass (Oligochaeta) Thorp & Rogers 2016 Hirudinida Species Klemm 1982, Klemm et al. 2015 Anostraca Species Thorp & Rogers 2016 Species (Lynceus Laevicaudata Thorp & Rogers 2016 brachyurus) Spinicaudata Genus Thorp & Rogers 2016 Williams 1972, Thorp & Rogers Isopoda Genus 2016 Holsinger 1972, Thorp & Rogers Amphipoda Genus 2016 Gammaridae: Gammarus Species Holsinger 1972 Crustacea monotypic genera: Apocorophium lacustre, Echinogammarus ischnus, Synurella dentata Species (Taphromysis Mysida Thorp & Rogers 2016 louisianae) Crocker & Barr 1968; Jezerinac 1993, 1995; Jezerinac & Thoma 1984; Taylor 2000; Thoma et al. Cambaridae Species 2005; Thoma & Stocker 2009; Crandall & De Grave 2017; Glon et al. 2018 Species (Palaemon Pennak 1989, Palaemonidae kadiakensis) Thorp & Rogers 2016 1 Ohio EPA Macroinvertebrate Taxonomic Level December 2019 Taxon Subtaxon Taxonomic Level Taxonomic Key(ies) Informal grouping of the Arachnida Hydrachnidia Smith 2001 water mites Genus Morse et al. -

DNA Barcoding
Full-time PhD studies of Ecology and Environmental Protection Piotr Gadawski Species diversity and origin of non-biting midges (Chironomidae) from a geologically young lake PhD Thesis and its old spring system Performed in Department of Invertebrate Zoology and Hydrobiology in Institute of Ecology and Environmental Protection Różnorodność gatunkowa i pochodzenie fauny Supervisor: ochotkowatych (Chironomidae) z geologicznie Prof. dr hab. Michał Grabowski młodego jeziora i starego systemu źródlisk Auxiliary supervisor: Dr. Matteo Montagna, Assoc. Prof. Łódź, 2020 Łódź, 2020 Table of contents Acknowledgements ..........................................................................................................3 Summary ...........................................................................................................................4 General introduction .........................................................................................................6 Skadar Lake ...................................................................................................................7 Chironomidae ..............................................................................................................10 Species concept and integrative taxonomy .................................................................12 DNA barcoding ...........................................................................................................14 Chapter I. First insight into the diversity and ecology of non-biting midges (Diptera: Chironomidae) -

Tanytarsini (Diptera: Chironomidae)
applyparastyle “fig//caption/p[1]” parastyle “FigCapt” This is a pre-copyedited, author-produced version of an article accepted for publication in ZOOLOGICAL JOURNAL OF THE LINNEAN SOCIETY following peer review. The version of record Zakrzewska M., Singh H., Wagner-Wysiecka E., Giłka W., Minute and diverse in fossil sticky stuff: Tanytarsini (Diptera: Chironomidae) from early Eocene Indian Cambay amber, Zoological Journal of the Linnean Society, zlz159 is available online at: DOI: 10.1093/antazolina/zlz159 Minute and diverse in fossil sticky stuff: Tanytarsini (Diptera: Chironomidae) from early Eocene Indian Cambay amber MARTA ZAKRZEWSKA1, HUKAM SINGH2, EWA WAGNER-WYSIECKA3 and WOJCIECH GIŁKA1* 1Laboratory of Systematic Zoology, Department of Invertebrate Zoology and Parasitology, Faculty of Biology, University of Gdańsk, Wita Stwosza 59, 80-308 Gdańsk, Poland 2Birbal Sahni Institute of Palaeosciences, 53 University Road, Lucknow, India 3Department of Chemistry and Technology of Functional Materials, Faculty of Chemistry, Gdańsk University of Technology, Narutowicza 11/12, 80-233 Gdańsk, Poland We here present a pioneering systematic review of fossil dipterans of the tribe Tanytarsini (family Chironomidae) discovered in Indian amber from Cambay. The specimens examined belong to five species: Gujaratomyia miripes, Stempellina stebneri sp. nov., Stempellinella pollex sp. nov., Tanytarsus forfex sp. nov. and Tanytarsus ramus sp. nov., which are described. All species belong to the oldest known Tanytarsini and come from the Cambay shale formation in Tadkeshwar, dated to the early Eocene (~54 Mya). Displaying unusual characters/structures of diagnostic and phylogenetic importance, the specimens studied are discussed against the background of the evolution and systematics of the oldest fossil (Eocene) and extant representatives in the tribe. -

Genomic Platforms and Molecular Physiology of Insect Stress Tolerance
Genomic Platforms and Molecular Physiology of Insect Stress Tolerance DISSERTATION Presented in Partial Fulfillment of the Requirements for the Degree Doctor of Philosophy in the Graduate School of The Ohio State University By Justin Peyton MS Graduate Program in Evolution, Ecology and Organismal Biology The Ohio State University 2015 Dissertation Committee: Professor David L. Denlinger Advisor Professor Zakee L. Sabree Professor Amanda A. Simcox Professor Joseph B. Williams Copyright by Justin Tyler Peyton 2015 Abstract As ectotherms with high surface area to volume ratio, insects are particularly susceptible to desiccation and low temperature stress. In this dissertation, I examine the molecular underpinnings of two facets of these stresses: rapid cold hardening and cryoprotective dehydration. Rapid cold hardening (RCH) is an insect’s ability to prepare for cold stress when that stress is preceded by an intermediate temperature for minutes to hours. In order to gain a better understanding of cold shock, recovery from cold shock, and RCH in Sarcophaga bullata I examine the transcriptome with microarray and the metabolome with gas chromatography coupled with mass spectrometry (GCMS) in response to these treatments. I found that RCH has very little effect on the transcriptome, but results in a shift from aerobic metabolism to glycolysis/gluconeogenesis during RCH and preserved metabolic homeostasis during recovery. In cryoprotective dehydration (CD), a moisture gradient is established between external ice and the moisture in the body of an insect. As temperatures decline, the external ice crystals grow, drawing in more moisture which dehydrates the insect causing its melting point to track the ambient temperature. To gain a better understanding of CD and dehydration in Belgica antarctica I explore the transcriptome with RNA sequencing ii and the metabolome with GCMS. -

Toxicity of Sediment-Bound Lufenuron to Benthic Arthropods in Laboratory Bioassays
Toxicity of sediment-bound lufenuron to benthic arthropods in laboratory bioassays Brock, T. C. M., Belgers, J. D. M., Boerwinkel, M. C., Jollie, L., Kraak, M. H. S., Papo, M. J., ... Roessink, I. This is a "Post-Print" accepted manuscript, which has been published in "Aquatic Toxicology" This version is distributed under a non-commercial no derivatives Creative Commons (CC-BY-NC-ND) user license, which permits use, distribution, and reproduction in any medium, provided the original work is properly cited and not used for commercial purposes. Further, the restriction applies that if you remix, transform, or build upon the material, you may not distribute the modified material. Please cite this publication as follows: Brock, T. C. M., Belgers, J. D. M., Boerwinkel, M. C., Jollie, L., Kraak, M. H. S., Papo, M. J., ... Roessink, I. (2018). Toxicity of sediment-bound lufenuron to benthic arthropods in laboratory bioassays. Aquatic Toxicology, 198, 118-128. DOI: 10.1016/j.aquatox.2018.03.005 You can download the published version at: https://doi.org/10.1016/j.aquatox.2018.03.005 Toxicity of sediment-bound lufenuron to benthic arthropods in laboratory bioassays T.C.M. Brocka,#, J.D.M. Belgersa, M-C. Boerwinkela, L. Jollieb, M.H.S. Kraakb, M.J. Papob, J.A. Vonkb, I. Roessinka a Wageningen Environmental Research (Alterra), Wageningen University and Research, P.O. Box 47, 6700 AA Wageningen, The Netherlands b Institute for Biodiversity and Ecosystem Dynamics (IBED), University of Amsterdam, The Netherlands # Corresponding author. E-mail address: [email protected] (T.C.M. Brock) Key words: Sediment ecotoxicology Benzoylurea insecticide Sediment-spiked laboratory toxicity tests Benthic macroinvertebrates Species Sensitivity Distributions Regulatory Acceptable Concentration 2 ABSTRACT This paper deals with species sensitivity distributions (SSDs) for the lipophilic insecticide lufenuron and benthic arthropods based on sediment-spiked laboratory toxicity tests. -

MONITORING of BIOLOGICAL VARIABLES in Chironomus Sancticaroli CULTURING AS TEST ORGANISM
MONITORING OF BIOLOGICAL VARIABLES IN Chironomus sancticaroli CULTURING AS TEST ORGANISM William Viveiros 1, Afonso Rodrigues Aquino 2 1 Companhia Ambiental do Estado de São Paulo – CETESB Setor de Ecotoxicologia Aquática Av. Professor Frederico Hermann Jr, 345 05459-900 São Paulo, SP [email protected] 2 Instituto de Pesquisas Energéticas e Nucleares – IPEN/CNEM-SP Av. Preofessor Lineu Prestes 2242 05508-000 São Paulo, SP [email protected] ABSTRACT International protocols such as USEPA, ASTM, OECD describe procedures for ecotoxicity tests with sediment samples using benthic organisms such as insect larvae of the genus Chironomus sp . The criteria for acceptability of the test are based on variables observed in control. The same protocols adopt the sensibility to the reference toxicant to ensure the quality of the test organisms before their use in testing, but do not describe methods for monitoring biological variables in cultures of these organisms. This study aims to monitor the variables: survival, mentum deformity, fecundity, fertility and hatching rate of the species Chironomus sancticaroli cultivated in laboratory under specific conditions in order to prepare a database for the future establishment of acceptance criteria for these variables. The average percentage of survival obtained was 89.3%( σ = 12.8). The average mentum deformity found was 13% ( σ = 13.9), while the fecundity (530 eggs, σ = 116) and fertility (225 larvae, σ = 135) resulted in an average hatching rate of 42% ( σ = 21.1). The variation data found although consistent with data from other author suggesting the influence of external factors such as the presence of contaminants in culture water, in addition to genetic interference caused by the inbreeding. -

Pupae of Mesozoic Oryctochlus Kalugina, 85 (Chironomidae: Podonominae)
Proceedings of the 8th International Symposium on Chironomidae - Fauna norvegica 202 Vol. 3: 5-165. ISSN: 502-4873 Pupae of Mesozoic Oryctochlus Kalugina, 1985 (Chironomidae: Podonominae), with description of two new species Elena D. Lukashevich Lukashevich ED. 2012. Pupae of Mesozoic Oryctochlus Kalugina, 1985 (Chironomidae: Podonominae), with description of two new species. Fauna norvegica 31: 159-165. The Mesozoic Chironomidae are briefly reviewed. Two new species, Oryctochlus brundini n.sp. and O. kaluginae n.sp. from the Late Jurassic of Mongolia, are described based on pupae. The species of Oryctochlus known from pupae are photographed and keyed, their chaetotaxy is discussed. doi: 10.5324/fn.v31i0.1400. Received: 2011-10-28. Accepted: 2012-06-26. Published on paper and online: 2012-10-17. Keywords: Chironomidae, Podonominae, fossil, new species, SEM 1. Borissiak Paleontological Institute RAS, Profsoyuznaya ul. 123, 117997 Moscow, Russia E-mail: [email protected] IntRODUction Diamesinae, Orthocladiinae, and possibly Buchonomyiinae. Fossil Chironomidae are abundant but relatively little studied. With exception of Manlayamyia Kalugina, 1980, Cretaceous They are known from all three periods of the Mesozoic chironomid genera are adult-based taxa. The vast majority of (Triassic, Jurassic and Cretaceous). The greater the age, the Cretaceous chironomids have been described from Lebanese lower the diversity and abundance of chironomids, and the amber and other fossil resins from Canada, Siberia and Great scantier our knowledge of them. Britain. Therefore, their descriptions follow the standards used in So far, there is no evidence that the role of chironomids in the systematics of extant midges (Evenhuis 1994; Jarzembowski Triassic and Early Jurassic ecosystems was as significant as et al. -

THE CHIRONOMIDAE of OTSEGO LAKE with KEYS to the IMMATURE STAGES of the SUBFAMILIES TANYPODINAE and DIAMESINAE (DIPTERA) Joseph
THE CHIRONOMIDAE OF OTSEGO LAKE WITH KEYS TO THE IMMATURE STAGES OF THE SUBFAMILIES TANYPODINAE AND DIAMESINAE (DIPTERA) Joseph P. Fagnani Willard N. Harman BIOLOGICAL FIELD STATION COOPERSTOWN, NEW YORK OCCASIONAL PAPER NO. 20 AUGUST, 1987 BIOLOGY DEPARTMENT STATE UNIVERSITY COLLEGE AT ONEONTA THIS MANUSCRIPT IS NOT A FORMAL PUBLICATION. The information contained herein may not be cited or reproduced without permission of the author or the S.U.N.Y. Oneonta Biology Department ABSTRACT The species of Chironomidae inhabiting Otsego Lake, New York, were studied from 1979 through 1982. This report presents the results of a variety of collecting methods used in a diversity of habitats over a considerable temporal period. The principle emphasis was on sound taxonomy and rearing to associate immatures and adults. Over 4,000 individual rearings have provided the basis for description of general morphological stages that occur during the life cycles of these species. Keys to the larvae and pupae of the 4 subfamilies and 10 tribes of Chironomidae collected in Otsego Lake were compiled. Keys are also presented for the immature stages of 12 Tanypodinae and 2 Diamesinae species found in Otsego Lake. Labeled line drawings of the majority of structures and measurements used to identify the immature stages of most species of chironomids were adapted from the literature. Extensive photomicrographs are presented along with larval and pupal characteristics, taxonomic notes, synonymies, recent literature accounts and collection records for 17 species. These include: Chironominae Paratendipes albimanus (Meigen) (Chironomini); Ortl10cladiinae Psectrocladius (Psectrocladius) simulans (Johannsen) and I!ydrobaenus johannseni (Sublette); Diamesinae - Protanypus ramosus Saether (Protanypodini) and Potthastia longimana Kieffer (Diamesini); and Tanypodinae - Clinotanypus (Clinotanypus) pinguis (Loew) (Coelotanypodini), Tanypu~ (Tanypus) punctipennis Meigen (Tanypodini), Procladius (Psilotanypus) bellus (Loew); Var. -

The Species of the Genus Diamesa (Diptera, Chironomidae) Known to Occur in Italian Alps and Apennines
Zootaxa 4193 (2): 317–331 ISSN 1175-5326 (print edition) http://www.mapress.com/j/zt/ Article ZOOTAXA Copyright © 2016 Magnolia Press ISSN 1175-5334 (online edition) http://doi.org/10.11646/zootaxa.4193.2.7 http://zoobank.org/urn:lsid:zoobank.org:pub:A1A56E2F-3A9A-4A07-B3BB-EE50B669FB50 The species of the genus Diamesa (Diptera, Chironomidae) known to occur in Italian Alps and Apennines MATTEO MONTAGNA1, SANDRA URBANELLI2 & BRUNO ROSSARO3,4 1Dipartimento di Scienze Agrarie e Ambientali—Università degli Studi di Milano, Via Celoria 2, I-20133, Milano, Italy 2Department of Environmental Biology, Università "La Sapienza" of Rome, Italy 3Dipartimento di Scienze per gli Alimenti, la Nutrizione e l’Ambiente—Università degli Studi di Milano, Via Celoria 2, I-20133, Milano, Italy 4Corresponding author. E-mail: [email protected] Abstract Some rare species from Italian Alps, belonging to the genus Diamesa Meigen, 1835 (Diptera, Chironomidae) are here re- described as adult males, because only old, incomplete descriptions are available for these taxa. Terminology of male gen- italia is reviewed, diagnostic features are illustrated in detail, and notes on biology and geographical distribution of the examined species are provided. An identification key to the known adult males is presented. Key words: Diamesinae, Diamesini, taxonomy, aquatic insects, male genitalia Introduction New conservation policies for freshwater ecosystems need to be developed as a priority due to the presence of rare species with high conservation value. Growing threats to their existence require attention, considering their importance in maintaining natural food webs (Moore et al. 2009). Effective conservation strategies require the appropriate knowledge on the morphology of the rare species as well as an update of their distribution and ecology.