Tese Jucimar Zacaria.Pdf (4.725Mb)
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

要約) Doctoral Dissertation Antibiotics Shapes Population-Level Diversity In
) Doctoral Dissertation Antibiotics shapes population-level diversity in the human gut microbiome ( ) Nishijima Suguru Acknowledgments I would like to express my sincere gratitude to my supervisor, Prof. Masahira Hattori, whose expertise, knowledge and continuous encouragement throughout my research. My sincere thanks also go to Assoc. Prof. Kenshiro Oshima (The University of Tokyo), Dr. Wataru Suda and Dr. Seok-Won Kim for their motivation, immense support and encouragement throughout my work. I am also grateful to all my collaborators, Prof. Hidetoshi Morita (Okayama University) for his fecal sample collection, DNA isolation and sincere encouragement, Prof. Kenya Honda and Dr. Koji Atarashi (Keio University) for mice experiments, Assoc. Prof. Masahiro Umezaki (the University of Tokyo) for support for dietary data analysis, Dr. Todd D. Taylor (RIKEN) for support for writing manuscript and Dr. Yuu Hirose (Toyohashi University of technology) for DNA sequencing. I also would like to thank all past and present members of our laboratory, Erica Iioka, Misa Takagi, Emi Omori, Hiromi Kuroyamagi, Naoko Yamashita, Keiko Komiya, Rina Kurokawa, Chie Shindo, Yukiko Takayama and Yasue Hattori for their great technical support and kind assistance. i Antibiotics shapes population-level diversity in the human gut microbiome () Abstract The human gut microbiome has profound influences on the host’s physiology through its interference with various intestinal functions. The development of next-generation sequencing (NGS) technologies enabled us to comprehensively explore ecological and functional features of the gut microbiomes. Recent studies using the NGS-based metagenomic approaches have suggested high ecological diversity of the microbiome across countries. However, little is known about the structure and feature of the Japanese gut microbiome, and the factor that shapes the population-level diversity in the human gut microbiome. -

Propranolol-Mediated Attenuation of MMP-9 Excretion in Infants with Hemangiomas
Supplementary Online Content Thaivalappil S, Bauman N, Saieg A, Movius E, Brown KJ, Preciado D. Propranolol-mediated attenuation of MMP-9 excretion in infants with hemangiomas. JAMA Otolaryngol Head Neck Surg. doi:10.1001/jamaoto.2013.4773 eTable. List of All of the Proteins Identified by Proteomics This supplementary material has been provided by the authors to give readers additional information about their work. © 2013 American Medical Association. All rights reserved. Downloaded From: https://jamanetwork.com/ on 10/01/2021 eTable. List of All of the Proteins Identified by Proteomics Protein Name Prop 12 mo/4 Pred 12 mo/4 Δ Prop to Pred mo mo Myeloperoxidase OS=Homo sapiens GN=MPO 26.00 143.00 ‐117.00 Lactotransferrin OS=Homo sapiens GN=LTF 114.00 205.50 ‐91.50 Matrix metalloproteinase‐9 OS=Homo sapiens GN=MMP9 5.00 36.00 ‐31.00 Neutrophil elastase OS=Homo sapiens GN=ELANE 24.00 48.00 ‐24.00 Bleomycin hydrolase OS=Homo sapiens GN=BLMH 3.00 25.00 ‐22.00 CAP7_HUMAN Azurocidin OS=Homo sapiens GN=AZU1 PE=1 SV=3 4.00 26.00 ‐22.00 S10A8_HUMAN Protein S100‐A8 OS=Homo sapiens GN=S100A8 PE=1 14.67 30.50 ‐15.83 SV=1 IL1F9_HUMAN Interleukin‐1 family member 9 OS=Homo sapiens 1.00 15.00 ‐14.00 GN=IL1F9 PE=1 SV=1 MUC5B_HUMAN Mucin‐5B OS=Homo sapiens GN=MUC5B PE=1 SV=3 2.00 14.00 ‐12.00 MUC4_HUMAN Mucin‐4 OS=Homo sapiens GN=MUC4 PE=1 SV=3 1.00 12.00 ‐11.00 HRG_HUMAN Histidine‐rich glycoprotein OS=Homo sapiens GN=HRG 1.00 12.00 ‐11.00 PE=1 SV=1 TKT_HUMAN Transketolase OS=Homo sapiens GN=TKT PE=1 SV=3 17.00 28.00 ‐11.00 CATG_HUMAN Cathepsin G OS=Homo -

Review Article RNA Degradation in Staphylococcus Aureus: Diversity of Ribonucleases and Their Impact
Hindawi Publishing Corporation International Journal of Genomics Volume 2015, Article ID 395753, 12 pages http://dx.doi.org/10.1155/2015/395753 Review Article RNA Degradation in Staphylococcus aureus: Diversity of Ribonucleases and Their Impact Rémy A. Bonnin and Philippe Bouloc Institute for Integrative Biology of the Cell (I2BC), CEA, CNRS, UniversiteParis-Sud,Universit´ eParis-Saclay,91400Orsay,France´ Correspondence should be addressed to Remy´ A. Bonnin; [email protected] Received 6 January 2015; Accepted 4 March 2015 Academic Editor: Martine A. Collart Copyright © 2015 R. A. Bonnin and P. Bouloc. This is an open access article distributed under the Creative Commons Attribution License, which permits unrestricted use, distribution, and reproduction in any medium, provided the original work is properly cited. The regulation of RNA decay is now widely recognized as having a central role in bacterial adaption to environmental stress.Here we present an overview on the diversity of ribonucleases (RNases) and their impact at the posttranscriptional level in the human pathogen Staphylococcus aureus. RNases in prokaryotes have been mainly studied in the two model organisms Escherichia coli and Bacillus subtilis. Based on identified RNases in these two models, putative orthologs have been identified in S. aureus.Themain staphylococcal RNases involved in the processing and degradation of the bulk RNA are (i) endonucleases RNase III and RNase Y and (ii) exonucleases RNase J1/J2 and PNPase, having 5 to 3 and 3 to 5 activities, respectively. The diversity and potential roles of each RNase and of Hfq and RppH are discussed in the context of recent studies, some of which are based on next-generation sequencing technology. -

Novel Missense Mutation in PTPN22 in a Chinese Pedigree With
Gong et al. BMC Endocrine Disorders (2018) 18:76 https://doi.org/10.1186/s12902-018-0305-8 RESEARCHARTICLE Open Access Novel missense mutation in PTPN22 in a Chinese pedigree with Hashimoto’s thyroiditis Licheng Gong1†, Beihong Liu2,3†, Jing Wang4, Hong Pan3, Anhui Qi2,3, Siyang Zhang2,3, Jinyi Wu1, Ping Yang1* and Binbin Wang3,4,5* Abstract Background: Hashimoto’s thyroiditis is a complex autoimmune thyroid disease, the onset of which is associated with environmental exposures and specific susceptibility genes. Its incidence in females is higher than its incidence in males. Thus far, although some susceptibility loci have been elaborated, including PTPN22, FOXP3, and CD25, the aetiology and pathogenesis of Hashimoto’s thyroiditis remains unclear. Methods: Four affected members from a Chinese family with Hashimoto’s thyroiditis were selected for whole-exome sequencing. Missense, nonsense, frameshift, or splicing-site variants shared by all affected members were identified after frequency filtering against public and internal exome databases. Segregation analysis was performed by Sanger sequencing among all members with available DNA. Results: We identified a missense mutation in PTPN22 (NM_015967.5; c. 77A > G; p.Asn26Ser) using whole-exome sequencing. PTPN22 is a known susceptibility gene associated with increased risks of multiple autoimmune diseases. Cosegregation analysis confirmed that all patients in this family, all of whom were female, carried the mutation. All public and private databases showed that the missense mutation was extremely rare. Conclusions: We found a missense mutation in PTPN22 in a Chinese HT pedigree using whole-exome sequencing. Our study, for the first time, linked a rare variant of PTPN22 to Hashimoto’s thyroiditis, providing further evidence of the disease-causing or susceptibility role of PTPN22 in autoimmune thyroid disease. -

Generated by SRI International Pathway Tools Version 25.0, Authors S
An online version of this diagram is available at BioCyc.org. Biosynthetic pathways are positioned in the left of the cytoplasm, degradative pathways on the right, and reactions not assigned to any pathway are in the far right of the cytoplasm. Transporters and membrane proteins are shown on the membrane. Periplasmic (where appropriate) and extracellular reactions and proteins may also be shown. Pathways are colored according to their cellular function. Gcf_000238675-HmpCyc: Bacillus smithii 7_3_47FAA Cellular Overview Connections between pathways are omitted for legibility. -
When the Reaction Is
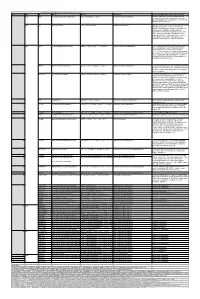
Table S3. iJL1678-ME model modification (blocked reactions) Iter. Cat. ID Name Formula Subsystem Comments (When the reaction is turned on) 1 bp2 EDD 6-phosphogluconate dehydratase 6pgc_c⇌2ddg6p_c + h2o_c Pentose Phosphate Pathway Create a major effect of steep acetate overflow elevation in high growth. Comparing to the main glycolytic pathway, it is metabolicly less efficient but proteomicly more efficient. bp1 ICL Isocitrate lyase icit_c→glx_c + succ_c Anaplerotic Reactions Bypass for the main TCA cycle pathways from turning isocitrate to succinate, when ICL is turned on, Isocitrate dehydrogenase(ICDHyr), 2-Oxogluterate dehydrogenase(AKGDH) and Succinyl-CoA synthetase (ATP-forming,SUCOAS) would reduce. Ref. (1) and (2) shows that this reaction is off in higher growth. Ref. (3) shows that this reaction is converging to being off when the dynamic of respiration using enzyme kinetics is simulated. 2 bp1 ABTA 4-aminobutyrate transaminase 4abut_c + akg_c⇌glu__L_c + sucsal_c Arginine and Proline Metabolism Another backup pathway of succinate production, from 2-Oxoglutarate (akg). Respiration would be induced when it is on, since the flux through ETC(CYTBO3_4pp and ATPS4rpp) would increase. As it requires the co-factor pyridoxal 5'-phosphate(2−) to get catalyzed(4), indicating that this reaction is regulated by the flux of other reactions(pyridoxal 5'- phosphate(2-) production, etc.). 3 GLYAT Glycine C-acetyltransferase accoa_c + gly_c⇌2aobut_c + coa_c Glycine and Serine Metabolism A reaction that back up for the respiration. Reactions fluxes in TCA cycle would drop when this reaction is turned on. It also requires pyridoxal 5'-phosphate(2−) for the regulation. 4 NADTRHD NAD transhydrogenase nad_c + nadph_c⇌nadh_c + nadp_c Oxidative Phosphorylation A reaction that would make the transition between NAD and NADP metabolically more efficient. -

Symbiotic Adaptations in the Fungal Cultivar of Leaf-Cutting Ants
ARTICLE Received 15 Apr 2014 | Accepted 24 Oct 2014 | Published 1 Dec 2014 DOI: 10.1038/ncomms6675 Symbiotic adaptations in the fungal cultivar of leaf-cutting ants Henrik H. De Fine Licht1,w, Jacobus J. Boomsma2 & Anders Tunlid1 Centuries of artificial selection have dramatically improved the yield of human agriculture; however, strong directional selection also occurs in natural symbiotic interactions. Fungus- growing attine ants cultivate basidiomycete fungi for food. One cultivar lineage has evolved inflated hyphal tips (gongylidia) that grow in bundles called staphylae, to specifically feed the ants. Here we show extensive regulation and molecular signals of adaptive evolution in gene trancripts associated with gongylidia biosynthesis, morphogenesis and enzymatic plant cell wall degradation in the leaf-cutting ant cultivar Leucoagaricus gongylophorus. Comparative analysis of staphylae growth morphology and transcriptome-wide expressional and nucleotide divergence indicate that gongylidia provide leaf-cutting ants with essential amino acids and plant-degrading enzymes, and that they may have done so for 20–25 million years without much evolutionary change. These molecular traits and signatures of selection imply that staphylae are highly advanced coevolutionary organs that play pivotal roles in the mutualism between leaf-cutting ants and their fungal cultivars. 1 Microbial Ecology Group, Department of Biology, Lund University, Ecology Building, SE-223 62 Lund, Sweden. 2 Centre for Social Evolution, Department of Biology, University of Copenhagen, Universitetsparken 15, DK-2100 Copenhagen, Denmark. w Present Address: Section for Organismal Biology, Department of Plant and Environmental Sciences, University of Copenhagen, Thorvaldsensvej 40, DK-1871 Frederiksberg, Denmark. Correspondence and requests for materials should be addressed to H.H.D.F.L. -

Open Matthew R Moreau Ph.D. Dissertation Finalfinal.Pdf
The Pennsylvania State University The Graduate School Department of Veterinary and Biomedical Sciences Pathobiology Program PATHOGENOMICS AND SOURCE DYNAMICS OF SALMONELLA ENTERICA SEROVAR ENTERITIDIS A Dissertation in Pathobiology by Matthew Raymond Moreau 2015 Matthew R. Moreau Submitted in Partial Fulfillment of the Requirements for the Degree of Doctor of Philosophy May 2015 The Dissertation of Matthew R. Moreau was reviewed and approved* by the following: Subhashinie Kariyawasam Associate Professor, Veterinary and Biomedical Sciences Dissertation Adviser Co-Chair of Committee Bhushan M. Jayarao Professor, Veterinary and Biomedical Sciences Dissertation Adviser Co-Chair of Committee Mary J. Kennett Professor, Veterinary and Biomedical Sciences Vijay Kumar Assistant Professor, Department of Nutritional Sciences Anthony Schmitt Associate Professor, Veterinary and Biomedical Sciences Head of the Pathobiology Graduate Program *Signatures are on file in the Graduate School iii ABSTRACT Salmonella enterica serovar Enteritidis (SE) is one of the most frequent common causes of morbidity and mortality in humans due to consumption of contaminated eggs and egg products. The association between egg contamination and foodborne outbreaks of SE suggests egg derived SE might be more adept to cause human illness than SE from other sources. Therefore, there is a need to understand the molecular mechanisms underlying the ability of egg- derived SE to colonize the chicken intestinal and reproductive tracts and cause disease in the human host. To this end, the present study was carried out in three objectives. The first objective was to sequence two egg-derived SE isolates belonging to the PFGE type JEGX01.0004 to identify the genes that might be involved in SE colonization and/or pathogenesis. -

A Kinase and Site-Specific Endoribonuclease
Ire1P 25 3 Ire1p: A Kinase and Site-Specific Endoribonuclease Tania N. Gonzalez and Peter Walter 1. Introduction 1.1. Ire1p in the Unfolded Protein Response The lumen of the endoplasmic reticulum (ER) is a highly specialized compartment in eukaryotic cells. Here, secretory and most membrane proteins are folded, covalently modified, and oligomerized with the assistance of specialized ER resident proteins (1). Perturbation of the ER lumen interferes with the production of many essential cellular components and can thus be highly deleterious. Indeed, in humans, defects in protein folding in the ER can lead to devastating diseases, such as cystic fibrosis, alpha1-anti- trypsin deficiency, and osteogenesis imperfecta (2). One way in which cells cope with the accumulation of unfolded proteins in the ER is by activating the unfolded protein response (UPR), an ER-to-nucleus signal transduction pathway (3–5). In the yeast Sac- charomyces cerevisiae, Ire1p is an essential component of this pathway. Ire1p is a transmembrane protein localized to ER membranes. When Ire1p was ini- tially identified as a component of the UPR, it was thought to function primarily as a serine/threonine kinase because of its homology to other kinases and its demonstrated kinase activity (6–8). Subsequent studies revealed that Ire1p functions as a site-spe- cific endoribonuclease as well (9). Both the kinase and endoribonuclease activities map to the carboxy-terminal half of Ire1p. The amino-terminal portion of Ire1p lies in the ER lumen (6,7), where it senses increases in the concentration of misfolded pro- teins. Since the ER and nuclear membranes are continuous, the carboxy-terminal portion of Ire1p is predicted to be located in either the cytoplasm and/or nucleus, where it induces downstream events in the UPR pathway. -

The Microbiota-Produced N-Formyl Peptide Fmlf Promotes Obesity-Induced Glucose
Page 1 of 230 Diabetes Title: The microbiota-produced N-formyl peptide fMLF promotes obesity-induced glucose intolerance Joshua Wollam1, Matthew Riopel1, Yong-Jiang Xu1,2, Andrew M. F. Johnson1, Jachelle M. Ofrecio1, Wei Ying1, Dalila El Ouarrat1, Luisa S. Chan3, Andrew W. Han3, Nadir A. Mahmood3, Caitlin N. Ryan3, Yun Sok Lee1, Jeramie D. Watrous1,2, Mahendra D. Chordia4, Dongfeng Pan4, Mohit Jain1,2, Jerrold M. Olefsky1 * Affiliations: 1 Division of Endocrinology & Metabolism, Department of Medicine, University of California, San Diego, La Jolla, California, USA. 2 Department of Pharmacology, University of California, San Diego, La Jolla, California, USA. 3 Second Genome, Inc., South San Francisco, California, USA. 4 Department of Radiology and Medical Imaging, University of Virginia, Charlottesville, VA, USA. * Correspondence to: 858-534-2230, [email protected] Word Count: 4749 Figures: 6 Supplemental Figures: 11 Supplemental Tables: 5 1 Diabetes Publish Ahead of Print, published online April 22, 2019 Diabetes Page 2 of 230 ABSTRACT The composition of the gastrointestinal (GI) microbiota and associated metabolites changes dramatically with diet and the development of obesity. Although many correlations have been described, specific mechanistic links between these changes and glucose homeostasis remain to be defined. Here we show that blood and intestinal levels of the microbiota-produced N-formyl peptide, formyl-methionyl-leucyl-phenylalanine (fMLF), are elevated in high fat diet (HFD)- induced obese mice. Genetic or pharmacological inhibition of the N-formyl peptide receptor Fpr1 leads to increased insulin levels and improved glucose tolerance, dependent upon glucagon- like peptide-1 (GLP-1). Obese Fpr1-knockout (Fpr1-KO) mice also display an altered microbiome, exemplifying the dynamic relationship between host metabolism and microbiota. -

Bul1, a New Protein That Binds to the Rsp5 Ubiquitin Ligase in Saccharomyces Cerevisiae
MOLECULAR AND CELLULAR BIOLOGY, July 1996, p. 3255–3263 Vol. 16, No. 7 0270-7306/96/$04.0010 Copyright q 1996, American Society for Microbiology Bul1, a New Protein That Binds to the Rsp5 Ubiquitin Ligase in Saccharomyces cerevisiae HIDEKI YASHIRODA, TOMOKO OGUCHI, YUKO YASUDA, AKIO TOH-E, AND YOSHIKO KIKUCHI* Department of Biological Sciences, Graduate School of Science, The University of Tokyo, Bunkyo-ku, Tokyo 113, Japan Received 23 October 1995/Returned for modification 27 November 1995/Accepted 26 March 1996 We characterized a temperature-sensitive mutant of Saccharomyces cerevisiae in which a mini-chromosome was unstable at a high temperature and cloned a new gene which encodes a basic and hydrophilic protein (110 kDa). The disruption of this gene caused the same temperature-sensitive growth as the original mutation. By using the two-hybrid system, we further isolated RSP5 (reverses Spt2 phenotype), which encodes a hect (homologous to E6-AP C terminus) domain, as a gene encoding a ubiquitin ligase. Thus, we named our gene BUL1 (for a protein that binds to the ubiquitin ligase). BUL1 seems to be involved in the ubiquitination pathway, since a high dose of UBI1, encoding a ubiquitin, partially suppressed the temperature sensitivity of the bul1 disruptant as well as that of a rsp5 mutant. Coexpression of RSP5 and BUL1 on a multicopy plasmid was toxic for mitotic growth of the wild-type cells. Pulse-chase experiments revealed that Bul1 in the wild-type cells remained stable, while the bands of Bul1 in the rsp5 cells were hardly detected. Since the steady-state levels of the protein were the same in the two strains as determined by immunoblotting analysis, Bul1 might be easily degraded during immunoprecipitation in the absence of intact Rsp5. -

(12) Patent Application Publication (10) Pub. No.: US 2012/0058468 A1 Mickeown (43) Pub
US 20120058468A1 (19) United States (12) Patent Application Publication (10) Pub. No.: US 2012/0058468 A1 MickeoWn (43) Pub. Date: Mar. 8, 2012 (54) ADAPTORS FOR NUCLECACID Related U.S. Application Data CONSTRUCTS IN TRANSMEMBRANE (60) Provisional application No. 61/148.737, filed on Jan. SEQUENCING 30, 2009. Publication Classification (75) Inventor: Brian Mckeown, Oxon (GB) (51) Int. Cl. CI2O I/68 (2006.01) (73) Assignee: OXFORD NANOPORE C7H 2L/00 (2006.01) TECHNOLGIES LIMITED, (52) U.S. Cl. ......................................... 435/6.1:536/23.1 Oxford (GB) (57) ABSTRACT (21) Appl. No.: 13/147,159 The invention relates to adaptors for sequencing nucleic acids. The adaptors may be used to generate single stranded constructs of nucleic acid for sequencing purposes. Such (22) PCT Fled: Jan. 29, 2010 constructs may contain both strands from a double stranded deoxyribonucleic acid (DNA) or ribonucleic acid (RNA) (86) PCT NO.: PCT/GB1O/OO160 template. The invention also relates to the constructs gener ated using the adaptors, methods of making the adaptors and S371 (c)(1), constructs, as well as methods of sequencing double stranded (2), (4) Date: Nov. 15, 2011 nucleic acids. Patent Application Publication Mar. 8, 2012 Sheet 1 of 4 US 2012/0058468 A1 Figure 5' 3 Figure 2 Figare 3 Patent Application Publication Mar. 8, 2012 Sheet 2 of 4 US 2012/0058468 A1 Figure 4 End repair Acid adapters ligate adapters Patent Application Publication Mar. 8, 2012 Sheet 3 of 4 US 2012/0058468 A1 Figure 5 Wash away type ifype foducts irrirrosilise Type| capture WashRE products away unbcure Pace É: Wash away unbound rag terts Free told fasgirre?ts aid raiser to festible Figure 6 Beatre Patent Application Publication Mar.