Departamento De Química - Física
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

WO 2012/148799 Al 1 November 2012 (01.11.2012) P O P C T
(12) INTERNATIONAL APPLICATION PUBLISHED UNDER THE PATENT COOPERATION TREATY (PCT) (19) World Intellectual Property Organization International Bureau (10) International Publication Number (43) International Publication Date WO 2012/148799 Al 1 November 2012 (01.11.2012) P O P C T (51) International Patent Classification: (81) Designated States (unless otherwise indicated, for every A61K 9/107 (2006.01) A61K 9/00 (2006.01) kind of national protection available): AE, AG, AL, AM, A 61 47/10 (2006.0V) AO, AT, AU, AZ, BA, BB, BG, BH, BR, BW, BY, BZ, CA, CH, CL, CN, CO, CR, CU, CZ, DE, DK, DM, DO, (21) International Application Number: DZ, EC, EE, EG, ES, FI, GB, GD, GE, GH, GM, GT, HN, PCT/US2012/034361 HR, HU, ID, IL, IN, IS, JP, KE, KG, KM, KN, KP, KR, (22) International Filing Date: KZ, LA, LC, LK, LR, LS, LT, LU, LY, MA, MD, ME, 20 April 2012 (20.04.2012) MG, MK, MN, MW, MX, MY, MZ, NA, NG, NI, NO, NZ, OM, PE, PG, PH, PL, PT, QA, RO, RS, RU, RW, SC, SD, (25) Filing Language: English SE, SG, SK, SL, SM, ST, SV, SY, TH, TJ, TM, TN, TR, (26) Publication Language: English TT, TZ, UA, UG, US, UZ, VC, VN, ZA, ZM, ZW. (30) Priority Data: (84) Designated States (unless otherwise indicated, for every 61/480,259 28 April 201 1 (28.04.201 1) US kind of regional protection available): ARIPO (BW, GH, GM, KE, LR, LS, MW, MZ, NA, RW, SD, SL, SZ, TZ, (71) Applicant (for all designated States except US): BOARD UG, ZM, ZW), Eurasian (AM, AZ, BY, KG, KZ, MD, RU, OF REGENTS, THE UNIVERSITY OF TEXAS SYS¬ TJ, TM), European (AL, AT, BE, BG, CH, CY, CZ, DE, TEM [US/US]; 201 West 7th St., Austin, TX 78701 (US). -

Oral Fluid Drug Test Package Insert
Marijuana (THC) 11-nor-Δ9-THC-9 COOH 4 MTD: Methadone is a synthetic analgesic drug originally used for the Oral Fluid Drug Test treatment of narcotic addiction. In addition to use as a narcotic agonist, methadone is being used more frequently as a pain management agent. The Marijuana (THC) Δ9-THC 50 Package Insert psychological effects induced by using methadone are analgesia, sedation, and respiratory depression. Based on the saliva/plasma ratio calculated over Package insert for testing of the following drugs: > 0.02 % Alcohol (ALC) Alcohol salivary pH ranges of 6.4-7.6 for therapeutic or recreational doses of Amphetamine,Cocaine,Marijuana,Methamphetamine,Opiate,Methadone, B.A.C methadone, a cut-off <50 ng/mL is suggested. Due to this recommendation, Phencyclidine,Oxycodone,Benzodiazepine,Buprenorphine,Barbiturates, the cut-off level of the methadone test was calibrated to 30 ng/mL. Cotinine,EDDP,MDMA,6-MAM,Propoxyphene ,Fentanyl,ETG and Alcohol Tramadol(TRA) 50 Tramadol PCP: Phencyclidine is a hallucinogen and, can be detected in oral fluid as a INTENDED USE & SUMMARY result of the exchange of the drug between the circulatory system and the oral cavity.5 Fentanyl(FEN) 10 The Oral Fluid Drug And Alcohol Test is intended for screening for the Norfentanyl presence of drugs and alcohol and their metabolites in oral fluid. For professional in vitro diagnostic use only. OXY: Oxycodone is a semi-synthetic opioid with a structural similarity to Ethyl codeine. The drug is manufactured by modifying thebaine, an alkaloid found in 150 The Oral Fluid Pipette Test is a lateral flow chromatographic immunoassay for Glucuronide(ETG) the opium poppy. -

Review Memorandum
510(k) SUBSTANTIAL EQUIVALENCE DETERMINATION DECISION SUMMARY ASSAY ONLY TEMPLATE A. 510(k) Number: k062165 B. Purpose for Submission: New device C. Measurand: Barbiturates D. Type of Test: Qualitative and semi-quantitative enzyme immunoassay E. Applicant: Ortho-Clinical Diagnostics, Inc. F. Proprietary and Established Names: VITROS Chemistry Products BARB Reagent VITROS Chemistry Products Calibrator 26 VITROS Chemistry Products FS Calibrator 1 VITROS Chemistry Products DAT Performance Verifiers I, II, III, IV and V G. Regulatory Information: 1. Regulation section: 21 CFR 862.3150, Barbiturates test system 21 CFR 862.3200, Clinical Toxicology Calibrator 21 CFR 862.3180, Clinical Toxicology Control 2. Classification: Class II, (reagent, calibrator) Class I, reserved (control) 3. Product code: DIS, DLJ and DIF 4. Panel: Toxicology (91) 1 H. Intended Use: 1. Intended use(s): See Indications for use. 2. Indication(s) for use: VITROS Chemistry Products BARB Reagent: For in vitro diagnostic use only. VITROS Chemistry Products BARB Reagent is used on VITROS 5,1 FS Chemistry Systems for the semi- quantitative or qualitative determination of barbiturates (BARB) in human urine using a cutoff of 200 ng/mL or 300 ng/mL. Measurements obtained with the VITROS BARB method are used in the diagnosis and treatment of barbiturates use or overdose. The VITROS Chemistry Products BARB assay is intended for use by professional laboratory personnel. It provides only a preliminary test result. A more specific alternative chemical method must be used to confirm a result with this assay. Gas Chromatograpy/Mass Spectrometry (GC/MS) is the preferred confirmatory method. Clinical consideration and professional judgment should be applied to any drug-of-abuse test result, particularly when evaluating a preliminary positive result. -

Partial Agreement in the Social and Public Health Field
COUNCIL OF EUROPE COMMITTEE OF MINISTERS (PARTIAL AGREEMENT IN THE SOCIAL AND PUBLIC HEALTH FIELD) RESOLUTION AP (88) 2 ON THE CLASSIFICATION OF MEDICINES WHICH ARE OBTAINABLE ONLY ON MEDICAL PRESCRIPTION (Adopted by the Committee of Ministers on 22 September 1988 at the 419th meeting of the Ministers' Deputies, and superseding Resolution AP (82) 2) AND APPENDIX I Alphabetical list of medicines adopted by the Public Health Committee (Partial Agreement) updated to 1 July 1988 APPENDIX II Pharmaco-therapeutic classification of medicines appearing in the alphabetical list in Appendix I updated to 1 July 1988 RESOLUTION AP (88) 2 ON THE CLASSIFICATION OF MEDICINES WHICH ARE OBTAINABLE ONLY ON MEDICAL PRESCRIPTION (superseding Resolution AP (82) 2) (Adopted by the Committee of Ministers on 22 September 1988 at the 419th meeting of the Ministers' Deputies) The Representatives on the Committee of Ministers of Belgium, France, the Federal Republic of Germany, Italy, Luxembourg, the Netherlands and the United Kingdom of Great Britain and Northern Ireland, these states being parties to the Partial Agreement in the social and public health field, and the Representatives of Austria, Denmark, Ireland, Spain and Switzerland, states which have participated in the public health activities carried out within the above-mentioned Partial Agreement since 1 October 1974, 2 April 1968, 23 September 1969, 21 April 1988 and 5 May 1964, respectively, Considering that the aim of the Council of Europe is to achieve greater unity between its members and that this -
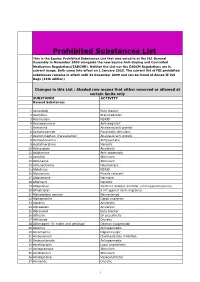
Prohibited Substances List
Prohibited Substances List This is the Equine Prohibited Substances List that was voted in at the FEI General Assembly in November 2009 alongside the new Equine Anti-Doping and Controlled Medication Regulations(EADCMR). Neither the List nor the EADCM Regulations are in current usage. Both come into effect on 1 January 2010. The current list of FEI prohibited substances remains in effect until 31 December 2009 and can be found at Annex II Vet Regs (11th edition) Changes in this List : Shaded row means that either removed or allowed at certain limits only SUBSTANCE ACTIVITY Banned Substances 1 Acebutolol Beta blocker 2 Acefylline Bronchodilator 3 Acemetacin NSAID 4 Acenocoumarol Anticoagulant 5 Acetanilid Analgesic/anti-pyretic 6 Acetohexamide Pancreatic stimulant 7 Acetominophen (Paracetamol) Analgesic/anti-pyretic 8 Acetophenazine Antipsychotic 9 Acetylmorphine Narcotic 10 Adinazolam Anxiolytic 11 Adiphenine Anti-spasmodic 12 Adrafinil Stimulant 13 Adrenaline Stimulant 14 Adrenochrome Haemostatic 15 Alclofenac NSAID 16 Alcuronium Muscle relaxant 17 Aldosterone Hormone 18 Alfentanil Narcotic 19 Allopurinol Xanthine oxidase inhibitor (anti-hyperuricaemia) 20 Almotriptan 5 HT agonist (anti-migraine) 21 Alphadolone acetate Neurosteriod 22 Alphaprodine Opiod analgesic 23 Alpidem Anxiolytic 24 Alprazolam Anxiolytic 25 Alprenolol Beta blocker 26 Althesin IV anaesthetic 27 Althiazide Diuretic 28 Altrenogest (in males and gelidngs) Oestrus suppression 29 Alverine Antispasmodic 30 Amantadine Dopaminergic 31 Ambenonium Cholinesterase inhibition 32 Ambucetamide Antispasmodic 33 Amethocaine Local anaesthetic 34 Amfepramone Stimulant 35 Amfetaminil Stimulant 36 Amidephrine Vasoconstrictor 37 Amiloride Diuretic 1 Prohibited Substances List This is the Equine Prohibited Substances List that was voted in at the FEI General Assembly in November 2009 alongside the new Equine Anti-Doping and Controlled Medication Regulations(EADCMR). -

September 25, 2020 Guangzhou Wondfo Biotech Co., Ltd. Joe Shia
September 25, 2020 Guangzhou Wondfo Biotech Co., Ltd. ℅ Joe Shia Manager LSI International 504 E Diamond Ave., Suite I Gaithersburg, MD 20877 Re: K202567 Trade/Device Name: Wondfo T-Dip® Multi-Drug Urine Test Panel Wondfo T-Dip® Multi-Drug Urine Test Panel Rx Regulation Number: 21 CFR 862.3100 Regulation Name: Amphetamine test system Regulatory Class: Class II Product Code: NFT, NGL, PTH, NFV, NFY, PTG, NGG, LCM, QBF, QAW, NFW Dated: September 2, 2020 Received: September 4, 2020 Dear Joe Shia: We have reviewed your Section 510(k) premarket notification of intent to market the device referenced above and have determined the device is substantially equivalent (for the indications for use stated in the enclosure) to legally marketed predicate devices marketed in interstate commerce prior to May 28, 1976, the enactment date of the Medical Device Amendments, or to devices that have been reclassified in accordance with the provisions of the Federal Food, Drug, and Cosmetic Act (Act) that do not require approval of a premarket approval application (PMA). You may, therefore, market the device, subject to the general controls provisions of the Act. Although this letter refers to your product as a device, please be aware that some cleared products may instead be combination products. The 510(k) Premarket Notification Database located at https://www.accessdata.fda.gov/scripts/cdrh/cfdocs/cfpmn/pmn.cfm identifies combination product submissions. The general controls provisions of the Act include requirements for annual registration, listing of devices, good manufacturing practice, labeling, and prohibitions against misbranding and adulteration. Please note: CDRH does not evaluate information related to contract liability warranties. -

(12) United States Patent (10) Patent No.: US 8,124,126 B2 Bosse Et Al
USOO81241.26B2 (12) United States Patent (10) Patent No.: US 8,124,126 B2 BOSse et al. (45) Date of Patent: Feb. 28, 2012 (54) PHARMACEUTICAL COMPOSITIONS (58) Field of Classification Search ........................ None See application file for complete search history. (75) Inventors: Paul Bosse, Charleston, SC (US); John Ameling, Cincinnati, OH (US); Bernard (56) References Cited Schachtel, Jupiter, FL (US); Ray Takigiku, Loveland, OH (US) U.S. PATENT DOCUMENTS 4,113,866 A 9, 1978 Lednicer et al. (73) Assignee: Charleston Laboratories, Inc., (Continued) Charleston, SC (US) FOREIGN PATENT DOCUMENTS (*) Notice: Subject to any disclaimer, the term of this CA 2665841 10/2007 patent is extended or adjusted under 35 U.S.C. 154(b) by 0 days. (Continued) (21) Appl. No.: 12/351,704 OTHER PUBLICATIONS Alexander, etal. Comparison ofondansetron and droperidol in reduc (22) Filed: Jan. 9, 2009 ing postoperative nausea and vomiting associated with patient-con trolled analgesia. Anaesthesia. Dec. 1995:50(12): 1086-8. (65) Prior Publication Data (Continued) US 2009/O175939 A1 Jul. 9, 2009 Primary Examiner — Susan Tran Related U.S. Application Data (74) Attorney, Agent, or Firm — Wilson Sonsini Goodrich & (60) Provisional application No. 61/020,139, filed on Jan. Rosati 9, 2008, provisional application No. 61/043,037, filed on Apr. 7, 2008, provisional application No. (57) ABSTRACT 61/060,758, filed on Jun. 11, 2008. Methods and compositions are provided which comprise effective amounts of analgesic to treat a subject, including (51) Int. Cl. reducing or eliminating an adverse effect associated with the A6 IK9/20 (2006.01) analgesic. A6 IK9/24 (2006.01) (52) U.S. -

(12) Patent Application Publication (10) Pub. No.: US 2010/0081713 A1 Sharma Et Al
US 20100081713A1 (19) United States (12) Patent Application Publication (10) Pub. No.: US 2010/0081713 A1 Sharma et al. (43) Pub. Date: Apr. 1, 2010 (54) COMPOSITIONS AND METHODS FOR (22) Filed: Mar. 18, 2009 TREATINGVIRAL INFECTIONS Related U.S. Application Data (75) Inventors: Geeta Sharma, Singapore (SG); (60) Provisional application No. 61/069,917, filed on Mar. Ralf Altmeyer, Singapore (SG); 19, 2008. Vishal Pendharker, Singapore (SG); Yu Chen, Singapore (SG); Publication Classification Michael Foley, Chestnut Hill, MA (51) Int. Cl. (US) A63L/35 (2006.01) A6II 3L/25 (2006.01) Correspondence Address: A63L/35 (2006.01) Gearhart Law LLC A6II 3/13 (2006.01) 4 Femdale Avenue A6IP3L/2 (2006.01) Chatham, NJ 07928 (US) (52) U.S. Cl. .......... 514/459; 514/529; 514/647: 514/662 (73) Assignee: CombinatoRx, (Singapore) Pte. (57) ABSTRACT Ltd. The present invention provides compositions, methods, and kits for treating or preventing a viral infection (e.g., an infec (21) Appl. No.: 12/406,716 tion caused by an influenza virus). Patent Application Publication Apr. 1, 2010 Sheet 1 of 2 US 2010/0081713 A1 ------ 80 r -0. Vehicle 0.5% HPMC g - - Sertraline-30mg/kg/day - £ 60 “A Sertraline-100mg/kg/day/kg/day i -v. Oseltamivir-30mg/kg/day ...i -0. Oseltamivir-100mg/kg/day -0. (Sertraline 30mg/kg+ . 40 Prednisolone 0.1 mg/Kg) Figure 1 Patent Application Publication Apr. 1, 2010 Sheet 2 of 2 US 2010/0081713 A1 100 468OOO 2 O Wehicle Sentraline 10 mg/kg Sentraline 30mg/kg Setraline 100mg/kg Figure 2 US 2010/008 1713 A1 Apr. -

Pharmaceutical Appendix to the Tariff Schedule 2
Harmonized Tariff Schedule of the United States (2007) (Rev. 2) Annotated for Statistical Reporting Purposes PHARMACEUTICAL APPENDIX TO THE HARMONIZED TARIFF SCHEDULE Harmonized Tariff Schedule of the United States (2007) (Rev. 2) Annotated for Statistical Reporting Purposes PHARMACEUTICAL APPENDIX TO THE TARIFF SCHEDULE 2 Table 1. This table enumerates products described by International Non-proprietary Names (INN) which shall be entered free of duty under general note 13 to the tariff schedule. The Chemical Abstracts Service (CAS) registry numbers also set forth in this table are included to assist in the identification of the products concerned. For purposes of the tariff schedule, any references to a product enumerated in this table includes such product by whatever name known. ABACAVIR 136470-78-5 ACIDUM LIDADRONICUM 63132-38-7 ABAFUNGIN 129639-79-8 ACIDUM SALCAPROZICUM 183990-46-7 ABAMECTIN 65195-55-3 ACIDUM SALCLOBUZICUM 387825-03-8 ABANOQUIL 90402-40-7 ACIFRAN 72420-38-3 ABAPERIDONUM 183849-43-6 ACIPIMOX 51037-30-0 ABARELIX 183552-38-7 ACITAZANOLAST 114607-46-4 ABATACEPTUM 332348-12-6 ACITEMATE 101197-99-3 ABCIXIMAB 143653-53-6 ACITRETIN 55079-83-9 ABECARNIL 111841-85-1 ACIVICIN 42228-92-2 ABETIMUSUM 167362-48-3 ACLANTATE 39633-62-0 ABIRATERONE 154229-19-3 ACLARUBICIN 57576-44-0 ABITESARTAN 137882-98-5 ACLATONIUM NAPADISILATE 55077-30-0 ABLUKAST 96566-25-5 ACODAZOLE 79152-85-5 ABRINEURINUM 178535-93-8 ACOLBIFENUM 182167-02-8 ABUNIDAZOLE 91017-58-2 ACONIAZIDE 13410-86-1 ACADESINE 2627-69-2 ACOTIAMIDUM 185106-16-5 ACAMPROSATE 77337-76-9 -

(12) Patent Application Publication (10) Pub. No.: US 2011/0263526 A1 SATYAM (43) Pub
US 20110263526A1 (19) United States (12) Patent Application Publication (10) Pub. No.: US 2011/0263526 A1 SATYAM (43) Pub. Date: Oct. 27, 2011 (54) NITRICOXIDE RELEASING PRODRUGS OF CD7C 69/96 (2006.01) THERAPEUTICAGENTS C07C 319/22 (2006.01) CD7C 68/02 (2006.01) (75) Inventor: Apparao SATYAM, Mumbai (IN) A6IP 29/00 (2006.01) A6IP 9/00 (2006.01) (73) Assignee: PIRAMAL LIFE SCIENCES A6IP37/08 (2006.01) LIMITED, Mumbai (IN) A6IP35/00 (2006.01) A6IP 25/24 2006.O1 (21) Appl. No.: 13/092.245 A6IP 25/08 308: A6IP3L/04 2006.O1 (22) Filed: Apr. 22, 2011 A6IP3L/2 308: Related U.S. Application Data 39t. O 308: (60) Provisional application No. 61/327,175, filed on Apr. A6IP3/10 (2006.01) 23, 2010. A6IPL/04 (2006.01) A6IP39/06 (2006.01) Publication Classification A6IP3/02 (2006.01) (51) Int. Cl. A6IP 9/06 (2006.01) A 6LX 3L/7072 (2006.01) 39t. W 308: A6 IK3I/58 (2006.01) A6IP II/08 (2006.015 A6 IK3I/55 (2006.01) A63/62 (2006.015 A6 IK 3/495 (2006.01) A6 IK 3/4439 (2006.01) (52) U.S. Cl. ........... 514/50: 514/166; 514/172: 514/217; A6 IK 3L/455 (2006.01) 514/255.04: 514/338; 514/356; 514/412; A6 IK 3/403 (2006.01) 514/420; 514/423: 514/510,536/28.53:540/67; A6 IK 3/404 (2006.01) 540/591; 544/396; 546/273.7: 546/318: 548/452: A6 IK 3/40 (2006.01) 548/500: 548/537; 549/464; 558/275 A6 IK3I/265 (2006.01) C7H 9/06 (2006.01) (57) ABSTRACT CO7I 71/00 (2006.01) CO7D 22.3/26 (2006.01) The present invention relates to nitric oxide releasing pro C07D 295/14 (2006.01) drugs of known drugs or therapeutic agents which are repre CO7D 40/12 (2006.01) sented herein as compounds of formula (I) wherein the drugs CO7D 213/80 (2006.01) or therapeutic agents contain one or more functional groups C07D 209/52 (2006.01) independently selected from a carboxylic acid, an amino, a CO7D 209/26 (2006.01) hydroxyl and a sulfhydryl group. -

(19) 11 Patent Number: 6165500
USOO6165500A United States Patent (19) 11 Patent Number: 6,165,500 Cevc (45) Date of Patent: *Dec. 26, 2000 54 PREPARATION FOR THE APPLICATION OF WO 88/07362 10/1988 WIPO. AGENTS IN MINI-DROPLETS OTHER PUBLICATIONS 75 Inventor: Gregor Cevc, Heimstetten, Germany V.M. Knepp et al., “Controlled Drug Release from a Novel Liposomal Delivery System. II. Transdermal Delivery Char 73 Assignee: Idea AG, Munich, Germany acteristics” on Journal of Controlled Release 12(1990) Mar., No. 1, Amsterdam, NL, pp. 25–30. (Exhibit A). * Notice: This patent issued on a continued pros- C.E. Price, “A Review of the Factors Influencing the Pen ecution application filed under 37 CFR etration of Pesticides Through Plant Leaves” on I.C.I. Ltd., 1.53(d), and is subject to the twenty year Plant Protection Division, Jealott's Hill Research Station, patent term provisions of 35 U.S.C. Bracknell, Berkshire RG12 6EY, U.K., pp. 237-252. 154(a)(2). (Exhibit B). K. Karzel and R.K. Liedtke, “Mechanismen Transkutaner This patent is Subject to a terminal dis- Resorption” on Grandlagen/Basics, pp. 1487–1491. (Exhibit claimer. C). Michael Mezei, “Liposomes as a Skin Drug Delivery Sys 21 Appl. No.: 07/844,664 tem” 1985 Elsevier Science Publishers B.V. (Biomedical Division), pp 345-358. (Exhibit E). 22 Filed: Apr. 8, 1992 Adrienn Gesztes and Michael Mazei, “Topical Anesthesia of 30 Foreign Application Priority Data the Skin by Liposome-Encapsulated Tetracaine” on Anesth Analg 1988; 67: pp 1079–81. (Exhibit F). Aug. 24, 1990 DE) Germany ............................... 40 26834 Harish M. Patel, "Liposomes as a Controlled-Release Sys Aug. -

Pharmacopoeial Reference Standards and Their Current Lot Numbers EP, EPISA, ICRS, BP July 2019
Pharmacopoeial reference standards and their current lot numbers EP, EPISA, ICRS, BP July 2019 Mikromol Follow LGC on LinkedIn Dear user, This catalogue includes all pharmacopoeial reference materials from EP, EPISA, ICRS and BP available at LGC, as well as their current lot numbers. We update the catalogue on a monthly basis so that you can use the lot numbers to easily track the expiry dates of your stocked reference materials. We took the lot information from actual data from pharmacopoeias, correct at the time of production. We retained all spelling and information as provided by the pharmacopoeias. The list was compiled with caution, however, errors in this list may be possible, and LGC is not responsible for any consequences as a result of these errors, may it be errors in the original Pharmacopoeial data, or from LGC during compilation of this list. For any questions or orders please contact your local LGC office. You will find all contact details on the last page of this list. Alternatively, you can browse and buy pharmaceutical reference materials online at lgcstandards.com/mikromol. Stay connected via LinkedIn (LGC Mikromol) or Twitter @LGCMikromol. Kind regards Your LGC Standards Team LGC, Queens Road, Teddington, Middlesex, TW11 0LY, United Kingdom Tel: +44 (0)20 8943 8480 Fax: +44 (0)20 8943 7554 E-mail: [email protected] European Pharmacopoeia (EP) Available Current Unit Cat. No. Name What is new? since Batch No. Quantity EPY0001552 Abacavir for peak identification 1 10 mg EPY0001551 Abacavir for system suitability 1