Sorbonne Université́
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-
Comparative Gene Expression Profiling of Stromal Cell Matrices
ell Res C ea m rc te h S & f o T h l Journal of Tiwari et al., J Stem Cell Res Ther 2013, 3:4 e a r n a r p u DOI: 10.4172/2157-7633.1000152 y o J ISSN: 2157-7633 Stem Cell Research & Therapy Research Article Open Access Comparative Gene Expression Profiling of Stromal Cell Matrices that Support Expansion of Hematopoietic Stem/Progenitor Cells Abhilasha Tiwari1,2, Christophe Lefevre2, Mark A Kirkland2*, Kevin Nicholas2 and Gopal Pande1* 1CSIR-Centre for Cellular and Molecular Biology (CCMB), Hyderabad, India 2Deakin University, Waurn Ponds, Geelong, VIC, Australia Abstract The bone marrow microenvironment maintains a stable balance between self-renewal and differentiation of hematopoietic stem/progenitor cells (HSPCs). This microenvironment, also termed the “hematopoietic niche”, is primarily composed of stromal cells and their extracellular matrices (ECM) that jointly regulate HSPC functions. Previously, we have demonstrated that umbilical cord blood derived HSPCs can be maintained and expanded on stromal cell derived acellular matrices that mimic the complexity of the hematopoietic niche. The results indicated that matrices prepared at 20% O2 with osteogenic medium (OGM) were best suited for expanding committed HSPCs, whereas, matrices prepared at 5% O2 without OGM were better for primitive progenitors. Based upon these results we proposed that individual constituents of these matrices could be responsible for regulation of specific HSPC functions. To explore this hypothesis, we have performed comparative transcriptome profiling of these matrix producing cells, which identified differential expression of both known niche regulators, such as Wnt4, Angpt2, Vcam and Cxcl12, as well as genes not previously associated with HSPC regulation, such as Depp. -

Analysis of the Indacaterol-Regulated Transcriptome in Human Airway
Supplemental material to this article can be found at: http://jpet.aspetjournals.org/content/suppl/2018/04/13/jpet.118.249292.DC1 1521-0103/366/1/220–236$35.00 https://doi.org/10.1124/jpet.118.249292 THE JOURNAL OF PHARMACOLOGY AND EXPERIMENTAL THERAPEUTICS J Pharmacol Exp Ther 366:220–236, July 2018 Copyright ª 2018 by The American Society for Pharmacology and Experimental Therapeutics Analysis of the Indacaterol-Regulated Transcriptome in Human Airway Epithelial Cells Implicates Gene Expression Changes in the s Adverse and Therapeutic Effects of b2-Adrenoceptor Agonists Dong Yan, Omar Hamed, Taruna Joshi,1 Mahmoud M. Mostafa, Kyla C. Jamieson, Radhika Joshi, Robert Newton, and Mark A. Giembycz Departments of Physiology and Pharmacology (D.Y., O.H., T.J., K.C.J., R.J., M.A.G.) and Cell Biology and Anatomy (M.M.M., R.N.), Snyder Institute for Chronic Diseases, Cumming School of Medicine, University of Calgary, Calgary, Alberta, Canada Received March 22, 2018; accepted April 11, 2018 Downloaded from ABSTRACT The contribution of gene expression changes to the adverse and activity, and positive regulation of neutrophil chemotaxis. The therapeutic effects of b2-adrenoceptor agonists in asthma was general enriched GO term extracellular space was also associ- investigated using human airway epithelial cells as a therapeu- ated with indacaterol-induced genes, and many of those, in- tically relevant target. Operational model-fitting established that cluding CRISPLD2, DMBT1, GAS1, and SOCS3, have putative jpet.aspetjournals.org the long-acting b2-adrenoceptor agonists (LABA) indacaterol, anti-inflammatory, antibacterial, and/or antiviral activity. Numer- salmeterol, formoterol, and picumeterol were full agonists on ous indacaterol-regulated genes were also induced or repressed BEAS-2B cells transfected with a cAMP-response element in BEAS-2B cells and human primary bronchial epithelial cells by reporter but differed in efficacy (indacaterol $ formoterol . -
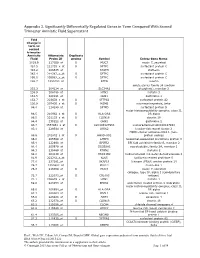
Appendix 2. Significantly Differentially Regulated Genes in Term Compared with Second Trimester Amniotic Fluid Supernatant
Appendix 2. Significantly Differentially Regulated Genes in Term Compared With Second Trimester Amniotic Fluid Supernatant Fold Change in term vs second trimester Amniotic Affymetrix Duplicate Fluid Probe ID probes Symbol Entrez Gene Name 1019.9 217059_at D MUC7 mucin 7, secreted 424.5 211735_x_at D SFTPC surfactant protein C 416.2 206835_at STATH statherin 363.4 214387_x_at D SFTPC surfactant protein C 295.5 205982_x_at D SFTPC surfactant protein C 288.7 1553454_at RPTN repetin solute carrier family 34 (sodium 251.3 204124_at SLC34A2 phosphate), member 2 238.9 206786_at HTN3 histatin 3 161.5 220191_at GKN1 gastrokine 1 152.7 223678_s_at D SFTPA2 surfactant protein A2 130.9 207430_s_at D MSMB microseminoprotein, beta- 99.0 214199_at SFTPD surfactant protein D major histocompatibility complex, class II, 96.5 210982_s_at D HLA-DRA DR alpha 96.5 221133_s_at D CLDN18 claudin 18 94.4 238222_at GKN2 gastrokine 2 93.7 1557961_s_at D LOC100127983 uncharacterized LOC100127983 93.1 229584_at LRRK2 leucine-rich repeat kinase 2 HOXD cluster antisense RNA 1 (non- 88.6 242042_s_at D HOXD-AS1 protein coding) 86.0 205569_at LAMP3 lysosomal-associated membrane protein 3 85.4 232698_at BPIFB2 BPI fold containing family B, member 2 84.4 205979_at SCGB2A1 secretoglobin, family 2A, member 1 84.3 230469_at RTKN2 rhotekin 2 82.2 204130_at HSD11B2 hydroxysteroid (11-beta) dehydrogenase 2 81.9 222242_s_at KLK5 kallikrein-related peptidase 5 77.0 237281_at AKAP14 A kinase (PRKA) anchor protein 14 76.7 1553602_at MUCL1 mucin-like 1 76.3 216359_at D MUC7 mucin 7, -

Tissue-Specific Transcriptome for Poeciliopsis Prolifica Reveals
INVESTIGATION Tissue-Specific Transcriptome for Poeciliopsis prolifica Reveals Evidence for Genetic Adaptation Related to the Evolution of a Placental Fish Nathaniel K. Jue,*,1 Robert J. Foley,* David N. Reznick,† Rachel J. O’Neill,* and Michael J. O’Neill*,2 *Institute for Systems Genomics and Department of Molecular and Cell Biology, University of Connecticut, Storrs, CT 06269 and †Department of Biology, University of California, Riverside, CA 92521 ABSTRACT The evolution of the placenta is an excellent model to examine the evolutionary processes KEYWORDS underlying adaptive complexity due to the recent, independent derivation of placentation in divergent transcriptome animal lineages. In fishes, the family Poeciliidae offers the opportunity to study placental evolution with positive selection respect to variation in degree of post-fertilization maternal provisioning among closely related sister gene expression species. In this study, we present a detailed examination of a new reference transcriptome sequence for the placenta live-bearing, matrotrophic fish, Poeciliopsis prolifica, from multiple-tissue RNA-seq data. We describe the fish genetic components active in liver, brain, late-stage embryo, and the maternal placental/ovarian complex, as well as associated patterns of positive selection in a suite of orthologous genes found in fishes. Results indicate the expression of many signaling transcripts, “non-coding” sequences and repetitive elements in the maternal placental/ovarian complex. Moreover, patterns of positive selection in protein sequence evolution were found associated with live-bearing fishes, generally, and the placental P. prolifica, specifi- cally, that appear independent of the general live-bearer lifestyle. Much of the observed patterns of gene expression and positive selection are congruent with the evolution of placentation in fish functionally converging with mammalian placental evolution and with the patterns of rapid evolution facilitated by the teleost-specific whole genome duplication event. -

TET2 Binds the Androgen Receptor and Loss Is Associated with Prostate Cancer
Oncogene (2016), 1–12 © 2016 Macmillan Publishers Limited, part of Springer Nature. All rights reserved 0950-9232/16 www.nature.com/onc ORIGINAL ARTICLE TET2 binds the androgen receptor and loss is associated with prostate cancer ML Nickerson1,14, S Das2,KMIm3, S Turan1, SI Berndt4,HLi1,5,HLou1,5, SA Brodie4,6, JN Billaud7, T Zhang8, AJ Bouk4,6, D Butcher9, Z Wang4, L Sun10, K Misner1,WTan1,5, A Esnakula11, D Esposito12, WY Huang4, RN Hoover4, MA Tucker4, JR Keller10, J Boland4,6, K Brown8, SK Anderson1, LE Moore4, WB Isaacs13, SJ Chanock4, M Yeager4,6, M Dean1,14 and T Andresson2 Genetic alterations associated with prostate cancer (PCa) may be identified by sequencing metastatic tumour genomes to identify molecular markers at this lethal stage of disease. Previously, we characterized somatic alterations in metastatic tumours in the methylcytosine dioxygenase ten-eleven translocation 2 (TET2), which is altered in 5–15% of myeloid, kidney, colon and PCas. Genome-wide association studies previously identified non-coding risk variants associated with PCa and melanoma. We perform fine-mapping of PCa risk across TET2 using genotypes from the PEGASUS case-control cohort and identify six new risk variants in introns 1 and 2. Oligonucleotides containing two risk variants are bound by the transcription factor octamer-binding protein 1 (Oct1/POU2F1) and TET2 and Oct1 expression are positively correlated in prostate tumours. TET2 is expressed in normal prostate tissue and reduced in a subset of tumours from the Cancer Genome Atlas (TCGA). Small interfering RNA-mediated TET2 knockdown (KD) increases LNCaP cell proliferation, migration and wound healing, verifying loss drives a cancer phenotype. -
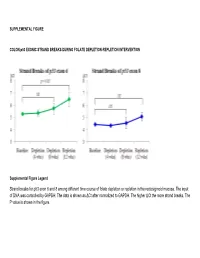
Strand Breaks for P53 Exon 6 and 8 Among Different Time Course of Folate Depletion Or Repletion in the Rectosigmoid Mucosa
SUPPLEMENTAL FIGURE COLON p53 EXONIC STRAND BREAKS DURING FOLATE DEPLETION-REPLETION INTERVENTION Supplemental Figure Legend Strand breaks for p53 exon 6 and 8 among different time course of folate depletion or repletion in the rectosigmoid mucosa. The input of DNA was controlled by GAPDH. The data is shown as ΔCt after normalized to GAPDH. The higher ΔCt the more strand breaks. The P value is shown in the figure. SUPPLEMENT S1 Genes that were significantly UPREGULATED after folate intervention (by unadjusted paired t-test), list is sorted by P value Gene Symbol Nucleotide P VALUE Description OLFM4 NM_006418 0.0000 Homo sapiens differentially expressed in hematopoietic lineages (GW112) mRNA. FMR1NB NM_152578 0.0000 Homo sapiens hypothetical protein FLJ25736 (FLJ25736) mRNA. IFI6 NM_002038 0.0001 Homo sapiens interferon alpha-inducible protein (clone IFI-6-16) (G1P3) transcript variant 1 mRNA. Homo sapiens UDP-N-acetyl-alpha-D-galactosamine:polypeptide N-acetylgalactosaminyltransferase 15 GALNTL5 NM_145292 0.0001 (GALNT15) mRNA. STIM2 NM_020860 0.0001 Homo sapiens stromal interaction molecule 2 (STIM2) mRNA. ZNF645 NM_152577 0.0002 Homo sapiens hypothetical protein FLJ25735 (FLJ25735) mRNA. ATP12A NM_001676 0.0002 Homo sapiens ATPase H+/K+ transporting nongastric alpha polypeptide (ATP12A) mRNA. U1SNRNPBP NM_007020 0.0003 Homo sapiens U1-snRNP binding protein homolog (U1SNRNPBP) transcript variant 1 mRNA. RNF125 NM_017831 0.0004 Homo sapiens ring finger protein 125 (RNF125) mRNA. FMNL1 NM_005892 0.0004 Homo sapiens formin-like (FMNL) mRNA. ISG15 NM_005101 0.0005 Homo sapiens interferon alpha-inducible protein (clone IFI-15K) (G1P2) mRNA. SLC6A14 NM_007231 0.0005 Homo sapiens solute carrier family 6 (neurotransmitter transporter) member 14 (SLC6A14) mRNA. -

C10ORF10/DEPP, a Transcriptional Target of FOXO3, Regulates ROS
Salcher et al. Molecular Cancer 2014, 13:224 http://www.molecular-cancer.com/content/13/1/224 RESEARCH Open Access C10ORF10/DEPP, a transcriptional target of FOXO3, regulates ROS-sensitivity in human neuroblastoma Stefan Salcher2,4, Judith Hagenbuchner2,4, Kathrin Geiger4, Maximilian A Seiter1,4, Johannes Rainer3,4, Reinhard Kofler3,4, Martin Hermann5, Ursula Kiechl-Kohlendorfer2, Michael J Ausserlechner1,4* and Petra Obexer2,4* Abstract Background: FOXO transcription factors control cellular levels of reactive oxygen species (ROS) which critically contribute to cell survival and cell death in neuroblastoma. In the present study we investigated the regulation of C10orf10/DEPP by the transcription factor FOXO3. As a physiological function of C10orf10/DEPP has not been described so far we analyzed its effects on cellular ROS detoxification and death sensitization in human neuroblastoma cells. Methods: The effect of DEPP on cellular ROS was measured by catalase activity assay and live cell fluorescence microscopy using the ROS-sensitive dye reduced MitoTracker Red CM-H2XROS. The cellular localization of DEPP was determined by confocal microscopy of EYFP-tagged DEPP, fluorescent peroxisomal- and mitochondrial probes and co-immunoprecipitation of the PEX7 receptor. Results: We report for the first time that DEPP regulates ROS detoxification and localizes to peroxisomes and mitochondria in neuroblastoma cells. FOXO3-mediated apoptosis involves a biphasic ROS accumulation. Knockdown of DEPP prevented the primary and secondary ROS wave during FOXO3 activation and attenuated FOXO3- and H2O2-induced apoptosis. Conditional overexpression of DEPP elevates cellular ROS levels and sensitizes to H2O2 and etoposide-induced cell death. In neuronal cells, cellular ROS are mainly detoxified in peroxisomes by the enzyme CAT/catalase. -

Biochimica Et Biophysica Acta 1828 (2013) 118–133
View metadata, citation and similar papers at core.ac.uk brought to you by CORE provided by Elsevier - Publisher Connector Biochimica et Biophysica Acta 1828 (2013) 118–133 Contents lists available at SciVerse ScienceDirect Biochimica et Biophysica Acta journal homepage: www.elsevier.com/locate/bbamem Review Regulation of connexin expression by transcription factors and epigenetic mechanisms☆ Masahito Oyamada a,⁎, Kumiko Takebe a, Yumiko Oyamada b a Department of Food Science and Human Nutrition, Faculty of Human Life Sciences, Fuji Women's University, Ishikarishi, Japan b Department of Surgical Pathology, Tonan Hospital, Sapporo, Japan article info abstract Article history: Gap junctions are specialized cell–cell junctions that directly link the cytoplasm of neighboring cells. They Received 17 September 2011 mediate the direct transfer of metabolites and ions from one cell to another. Discoveries of human genetic Received in revised form 17 December 2011 disorders due to mutations in gap junction protein (connexin [Cx]) genes and experimental data on connexin Accepted 27 December 2011 knockout mice provide direct evidence that gap junctional intercellular communication is essential for tissue Available online 4 January 2012 functions and organ development, and that its dysfunction causes diseases. Connexin-related signaling also involves extracellular signaling (hemichannels) and non-channel intracellular signaling. Thus far, 21 Keywords: fi Connexin human genes and 20 mouse genes for connexins have been identi ed. Each connexin shows tissue- or cell- Epigenetic regulation type-specific expression, and most organs and many cell types express more than one connexin. Connexin Gap junction expression can be regulated at many of the steps in the pathway from DNA to RNA to protein. -

81964717.Pdf
Developmental Biology 393 (2014) 195–208 Contents lists available at ScienceDirect Developmental Biology journal homepage: www.elsevier.com/locate/developmentalbiology Review The role of homeobox genes in retinal development and disease Jamie L. Zagozewski a,1, Qi Zhang b,1, Vanessa I. Pinto c, Jeffrey T. Wigle c,d, David D. Eisenstat a,c,e,n a Department of Medical Genetics, University of Alberta, Edmonton, AB, Canada T6G 2H7 b Department of Human Anatomy and Cell Science, University of Manitoba, Winnipeg, MB, Canada R3E 0J9 c Department of Biochemistry and Medical Genetics, University of Manitoba, Winnipeg, MB, Canada R3E 0J9 d Institute of Cardiovascular Sciences, St. Boniface Hospital Research Institute, Winnipeg, MB, Canada R2H 2A6 e Department of Pediatrics, University of Alberta, Edmonton, AB, Canada T6G 1C9 article info abstract Article history: Homeobox genes are an evolutionarily conserved class of transcription factors that are critical for Received 24 March 2014 development of many organ systems, including the brain and eye. During retinogenesis, homeodomain- Received in revised form containing transcription factors, which are encoded by homeobox genes, play essential roles in the 2 July 2014 regionalization and patterning of the optic neuroepithelium, specification of retinal progenitors and Accepted 8 July 2014 differentiation of all seven of the retinal cell classes that derive from a common progenitor. Home- Available online 15 July 2014 odomain transcription factors control retinal cell fate by regulating the expression of target genes Keywords: required for retinal progenitor cell fate decisions and for terminal differentiation of specific retinal cell Retina types. The essential role of homeobox genes during retinal development is demonstrated by the number Vertebrate of human eye diseases, including colobomas and anophthalmia, which are attributed to homeobox gene Homeobox mutations. -

The Homeobox Gene Chx10/Vsx2 Regulates Rdcvf Promoter Activity in the Inner Retina
THE HOMEOBOX GENE CHX10/VSX2 REGULATES RDCVF PROMOTER ACTIVITY IN THE INNER RETINA 1 2 1 1 Sacha ReichmanP ,P Ravi Kiran Reddy KalathurP ,P Sophie LambardP ,P Najate Ait-Ali , 3 2 2 2 1,3 Yanjiang Yang , Aurélie LardenoisP ,P Raymond RippP P Olivier PochP ,P Donald J. ZackP ,P 1 1 José-Alain SahelP P and Thierry Léveillard*P .P 1 Department of Genetics, Institut de la Vision, INSERM T Université Pierre et Marie Curie- 2 Paris6, UMR-S 968, Paris, F-75012 France,TP Institut de Génétique et de Biologie Moléculaire 3 et Cellulaire T (IGBMC),T 67404T Illkirch CEDEXT TFrance,P Wilmer Ophthalmological Institute, Johns Hopkins University School of Medicine, 600 N. Wolfe St. T Baltimore,T T MD 21287 USA. *Corresponding author: Thierry Léveillard, Department of Genetics, Institut de la Vision, INSERM, UPMC Univ Paris 06, UMR-S 968, CNRS 7210, Paris, F-75012 France. Tel : 33 1 53 46 25 48, Fax : 33 1 53 46 25 02, Email : [email protected] ABSTRACT Rod-derived Cone Viability Factor (RdCVF) is a trophic factor with therapeutic potential for the treatment of retinitis pigmentosa, a retinal disease that commonly results in blindness. RdCVF is encoded by Nucleoredoxin-like 1 (Nxnl1), a gene homologous with the family of thioredoxins that participate in the defense against oxidative stress. RdCVF expression is lost after rod degeneration in the first phase of retinitis pigmentosa, and this loss has been implicated in the more clinically significant secondary cone degeneration that often occurs. Here we describe a study of the Nxnl1 promoter using an approach that combines promoter and transcriptomic analysis. -

A Meta-Analysis of the Effects of High-LET Ionizing Radiations in Human Gene Expression
Supplementary Materials A Meta-Analysis of the Effects of High-LET Ionizing Radiations in Human Gene Expression Table S1. Statistically significant DEGs (Adj. p-value < 0.01) derived from meta-analysis for samples irradiated with high doses of HZE particles, collected 6-24 h post-IR not common with any other meta- analysis group. This meta-analysis group consists of 3 DEG lists obtained from DGEA, using a total of 11 control and 11 irradiated samples [Data Series: E-MTAB-5761 and E-MTAB-5754]. Ensembl ID Gene Symbol Gene Description Up-Regulated Genes ↑ (2425) ENSG00000000938 FGR FGR proto-oncogene, Src family tyrosine kinase ENSG00000001036 FUCA2 alpha-L-fucosidase 2 ENSG00000001084 GCLC glutamate-cysteine ligase catalytic subunit ENSG00000001631 KRIT1 KRIT1 ankyrin repeat containing ENSG00000002079 MYH16 myosin heavy chain 16 pseudogene ENSG00000002587 HS3ST1 heparan sulfate-glucosamine 3-sulfotransferase 1 ENSG00000003056 M6PR mannose-6-phosphate receptor, cation dependent ENSG00000004059 ARF5 ADP ribosylation factor 5 ENSG00000004777 ARHGAP33 Rho GTPase activating protein 33 ENSG00000004799 PDK4 pyruvate dehydrogenase kinase 4 ENSG00000004848 ARX aristaless related homeobox ENSG00000005022 SLC25A5 solute carrier family 25 member 5 ENSG00000005108 THSD7A thrombospondin type 1 domain containing 7A ENSG00000005194 CIAPIN1 cytokine induced apoptosis inhibitor 1 ENSG00000005381 MPO myeloperoxidase ENSG00000005486 RHBDD2 rhomboid domain containing 2 ENSG00000005884 ITGA3 integrin subunit alpha 3 ENSG00000006016 CRLF1 cytokine receptor like -

Supplemental Data
Supplementary Table 1. Gene sets from Figure 6. Lists of genes from each individual gene set defined in Figure 6, including the fold-change in expression of each gene in treatment group pair-wise comparisons. ENSEMBL: Ensembl gene identifier; Symbol: official gene symbol; logFC: log fold change; p value: significance of fold-change in a pair-wise comparison, P<0.05 cut-off; FDR: false discovery rate, expected proportion of false positives among the differentially expressed genes in a pair-wise comparison (FDR<0.25 cut-off). Sup. Table 1 SET I CP versus Sal CP versus CP+DCA DCA versus Sal ENSEMBL Symbol logFC PValue FDR logFC PValue FDR logFC PValue FDR Desc ENSMUSG00000020326 Ccng1 2.64 0.00 0.00 -0.06 0.13 0.96 0.40 0.00 0.23 cyclin G1 [Source:MGI Symbol;Acc:MGI:102890] ENSMUSG00000031886 Ces2e 3.97 0.00 0.00 -0.24 0.02 0.28 0.01 1.00 1.00 carboxylesterase 2E [Source:MGI Symbol;Acc:MGI:2443170] ENSMUSG00000041959 S100a10 2.31 0.00 0.00 -0.21 0.02 0.23 -0.11 0.53 1.00 S100 calcium binding protein A10 (calpactin) [Source:MGI Symbol;Acc:MGI:1339468] ENSMUSG00000092341 Malat1 1.09 0.00 0.00 -0.11 0.20 1.00 0.66 0.00 0.00 metastasis associated lung adenocarcinoma transcript 1 (non-coding RNA) [Source:MGI Symbol;Acc:MGI:1919539] ENSMUSG00000072949 Acot1 1.73 0.00 0.00 -0.22 0.01 0.12 -0.44 0.01 1.00 acyl-CoA thioesterase 1 [Source:MGI Symbol;Acc:MGI:1349396] ENSMUSG00000064339 mt-Rnr2 1.09 0.00 0.00 -0.08 0.17 1.00 0.67 0.00 0.07 mitochondrially encoded 16S rRNA [Source:MGI Symbol;Acc:MGI:102492] ENSMUSG00000025934 Gsta3 1.86 0.00 0.00 -0.28