Tonin Marianaferreira D.Pdf
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-
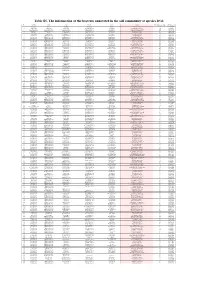
Table S5. the Information of the Bacteria Annotated in the Soil Community at Species Level
Table S5. The information of the bacteria annotated in the soil community at species level No. Phylum Class Order Family Genus Species The number of contigs Abundance(%) 1 Firmicutes Bacilli Bacillales Bacillaceae Bacillus Bacillus cereus 1749 5.145782459 2 Bacteroidetes Cytophagia Cytophagales Hymenobacteraceae Hymenobacter Hymenobacter sedentarius 1538 4.52499338 3 Gemmatimonadetes Gemmatimonadetes Gemmatimonadales Gemmatimonadaceae Gemmatirosa Gemmatirosa kalamazoonesis 1020 3.000970902 4 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas indica 797 2.344876284 5 Firmicutes Bacilli Lactobacillales Streptococcaceae Lactococcus Lactococcus piscium 542 1.594633558 6 Actinobacteria Thermoleophilia Solirubrobacterales Conexibacteraceae Conexibacter Conexibacter woesei 471 1.385742446 7 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas taxi 430 1.265115184 8 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas wittichii 388 1.141545794 9 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas sp. FARSPH 298 0.876754244 10 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sorangium cellulosum 260 0.764953367 11 Proteobacteria Deltaproteobacteria Myxococcales Polyangiaceae Sorangium Sphingomonas sp. Cra20 260 0.764953367 12 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas panacis 252 0.741416341 -

Genome Mining Reveals the Genus Xanthomonas to Be A
Royer et al. BMC Genomics 2013, 14:658 http://www.biomedcentral.com/1471-2164/14/658 RESEARCH ARTICLE Open Access Genome mining reveals the genus Xanthomonas to be a promising reservoir for new bioactive non-ribosomally synthesized peptides Monique Royer1, Ralf Koebnik2, Mélanie Marguerettaz1, Valérie Barbe3, Guillaume P Robin2, Chrystelle Brin4, Sébastien Carrere5, Camila Gomez1, Manuela Hügelland6, Ginka H Völler6, Julie Noëll1, Isabelle Pieretti1, Saskia Rausch6, Valérie Verdier2, Stéphane Poussier7, Philippe Rott1, Roderich D Süssmuth6 and Stéphane Cociancich1* Abstract Background: Various bacteria can use non-ribosomal peptide synthesis (NRPS) to produce peptides or other small molecules. Conserved features within the NRPS machinery allow the type, and sometimes even the structure, of the synthesized polypeptide to be predicted. Thus, bacterial genome mining via in silico analyses of NRPS genes offers an attractive opportunity to uncover new bioactive non-ribosomally synthesized peptides. Xanthomonas is a large genus of Gram-negative bacteria that cause disease in hundreds of plant species. To date, the only known small molecule synthesized by NRPS in this genus is albicidin produced by Xanthomonas albilineans. This study aims to estimate the biosynthetic potential of Xanthomonas spp. by in silico analyses of NRPS genes with unknown function recently identified in the sequenced genomes of X. albilineans and related species of Xanthomonas. Results: We performed in silico analyses of NRPS genes present in all published genome sequences of Xanthomonas spp., as well as in unpublished draft genome sequences of Xanthomonas oryzae pv. oryzae strain BAI3 and Xanthomonas spp. strain XaS3. These two latter strains, together with X. albilineans strain GPE PC73 and X. -

Rivadalve Coelho Gonçalves Etiologia Da Mancha
RIVADALVE COELHO GONÇALVES ETIOLOGIA DA MANCHA BACTERIANA DO EUCALIPTO NO BRASIL Tese apresentada à Universidade Federal de Viçosa, como parte das exigências do Programa de Pós- Graduação em Fitopatologia, para obtenção do título de Doctor Scientiae. VIÇOSA MINAS GERAIS – BRASIL 2003 Ficha catalográfica preparada pela Seção de Catalogação e Classificação da Biblioteca Central da UFV T Gonçalves, Rivadalve Coelho, 1970- G635e Etiologia da mancha bacteriana do eucalipto no Brasil / 2003 Rivadalve Coelho Gonçalves. – Viçosa : UFV, 2003. xiii, 79f. : il. ; 29cm. Inclui apêndice. Orientador: Acelino Couto Alfenas. Tese (doutorado) - Universidade Federal de Viçosa. Inclui bibliografia. 1. Mancha bacteriana - Etiologia. 2. Xanthomonas. 3. Eucalipto - Doenças e pragas. I. Universidade Federal de Viçosa. II.Título. CDD 20.ed. 632.32 RIVADALVE COELHO GONÇALVES ETIOLOGIA DA MANCHA BACTERIANA DO EUCALIPTO NO BRASIL Tese apresentada à Universidade Federal de Viçosa, como parte das exigências do Programa de Pós- Graduação em Fitopatologia, para obtenção do título de Doctor Scientiae. APROVADA: 6 de novembro de 2003. _______________________________ _______________________________ Prof. Luiz Antonio Maffia Prof. José Rogério de Oliveira (Conselheiro) (Conselheiro) _______________________________ _______________________________ Prof. Júlio Cézar Mattos Cascardo Dr. Miguel Angel Dita Rodríguez _______________________________ Prof. Acelino Couto Alfenas (Orientador) Ao professor Acelino Couto Alfenas DEDICO ii AGRADECIMENTOS A Deus, provedor de vida, inteligência -

Bacteria-Killing Type IV Secretion Systems
fmicb-10-01078 May 18, 2019 Time: 16:6 # 1 REVIEW published: 21 May 2019 doi: 10.3389/fmicb.2019.01078 Bacteria-Killing Type IV Secretion Systems Germán G. Sgro1†, Gabriel U. Oka1†, Diorge P. Souza1‡, William Cenens1, Ethel Bayer-Santos1‡, Bruno Y. Matsuyama1, Natalia F. Bueno1, Thiago Rodrigo dos Santos1, Cristina E. Alvarez-Martinez2, Roberto K. Salinas1 and Chuck S. Farah1* 1 Departamento de Bioquímica, Instituto de Química, Universidade de São Paulo, São Paulo, Brazil, 2 Departamento de Genética, Evolução, Microbiologia e Imunologia, Instituto de Biologia, University of Campinas (UNICAMP), Edited by: Campinas, Brazil Ignacio Arechaga, University of Cantabria, Spain Reviewed by: Bacteria have been constantly competing for nutrients and space for billions of years. Elisabeth Grohmann, During this time, they have evolved many different molecular mechanisms by which Beuth Hochschule für Technik Berlin, to secrete proteinaceous effectors in order to manipulate and often kill rival bacterial Germany Xiancai Rao, and eukaryotic cells. These processes often employ large multimeric transmembrane Army Medical University, China nanomachines that have been classified as types I–IX secretion systems. One of the *Correspondence: most evolutionarily versatile are the Type IV secretion systems (T4SSs), which have Chuck S. Farah [email protected] been shown to be able to secrete macromolecules directly into both eukaryotic and †These authors have contributed prokaryotic cells. Until recently, examples of T4SS-mediated macromolecule transfer equally to this work from one bacterium to another was restricted to protein-DNA complexes during ‡ Present address: bacterial conjugation. This view changed when it was shown by our group that many Diorge P. -

000468384900002.Pdf
UNIVERSIDADE ESTADUAL DE CAMPINAS SISTEMA DE BIBLIOTECAS DA UNICAMP REPOSITÓRIO DA PRODUÇÃO CIENTIFICA E INTELECTUAL DA UNICAMP Versão do arquivo anexado / Version of attached file: Versão do Editor / Published Version Mais informações no site da editora / Further information on publisher's website: https://www.frontiersin.org/articles/10.3389/fmicb.2019.01078/full DOI: 10.3389/fmicb.2019.01078 Direitos autorais / Publisher's copyright statement: ©2019 by Frontiers Research Foundation. All rights reserved. DIRETORIA DE TRATAMENTO DA INFORMAÇÃO Cidade Universitária Zeferino Vaz Barão Geraldo CEP 13083-970 – Campinas SP Fone: (19) 3521-6493 http://www.repositorio.unicamp.br fmicb-10-01078 May 18, 2019 Time: 16:6 # 1 REVIEW published: 21 May 2019 doi: 10.3389/fmicb.2019.01078 Bacteria-Killing Type IV Secretion Systems Germán G. Sgro1†, Gabriel U. Oka1†, Diorge P. Souza1‡, William Cenens1, Ethel Bayer-Santos1‡, Bruno Y. Matsuyama1, Natalia F. Bueno1, Thiago Rodrigo dos Santos1, Cristina E. Alvarez-Martinez2, Roberto K. Salinas1 and Chuck S. Farah1* 1 Departamento de Bioquímica, Instituto de Química, Universidade de São Paulo, São Paulo, Brazil, 2 Departamento de Genética, Evolução, Microbiologia e Imunologia, Instituto de Biologia, University of Campinas (UNICAMP), Edited by: Campinas, Brazil Ignacio Arechaga, University of Cantabria, Spain Reviewed by: Bacteria have been constantly competing for nutrients and space for billions of years. Elisabeth Grohmann, During this time, they have evolved many different molecular mechanisms by which Beuth Hochschule für Technik Berlin, to secrete proteinaceous effectors in order to manipulate and often kill rival bacterial Germany Xiancai Rao, and eukaryotic cells. These processes often employ large multimeric transmembrane Army Medical University, China nanomachines that have been classified as types I–IX secretion systems. -

DEEPT Genomics) Reveals Misclassification of Xanthomonas Species Complexes Into Xylella, Stenotrophomonas and Pseudoxanthomonas
bioRxiv preprint doi: https://doi.org/10.1101/2020.02.04.933507; this version posted February 5, 2020. The copyright holder for this preprint (which was not certified by peer review) is the author/funder. All rights reserved. No reuse allowed without permission. Deep phylo-taxono-genomics (DEEPT genomics) reveals misclassification of Xanthomonas species complexes into Xylella, Stenotrophomonas and Pseudoxanthomonas Kanika Bansal1,^, Sanjeet Kumar1,$,^, Amandeep Kaur1, Shikha Sharma1, Prashant Patil1,#, Prabhu B. Patil1,* 1Bacterial Genomics and Evolution Laboratory, CSIR-Institute of Microbial Technology, Chandigarh. $Present address: Department of Archaeogenetics, Max Planck Institute for the Science of Human History, Jena, Germany. #Present address: Department of Microbiology, School of Medicine, University of Washington, Seattle, WA, USA. ^Equal Contribution *Corresponding author Running Title: Comprehensive phylo-taxono-genomics of Xanthomonas and its relatives. Correspondence: Prabhu B. Patil Email: [email protected] Principal Scientist CSIR- Institute of Microbial Technology Sector 39-A, Chandigarh, India- 160036 bioRxiv preprint doi: https://doi.org/10.1101/2020.02.04.933507; this version posted February 5, 2020. The copyright holder for this preprint (which was not certified by peer review) is the author/funder. All rights reserved. No reuse allowed without permission. Abstract Genus Xanthomonas encompasses specialized group of phytopathogenic bacteria with genera Xylella, Stenotrophomonas and Pseudoxanthomonas being its closest relatives. While species of genera Xanthomonas and Xylella are known as serious phytopathogens, members of other two genera are found in diverse habitats with metabolic versatility of biotechnological importance. Few species of Stenotrophomonas are multidrug resistant opportunistic nosocomial pathogens. In the present study, we report genomic resource of genus Pseudoxanthomonas and further in-depth comparative studies with publically available genome resources of other three genera. -

1 a Horizontally Acquired Expansin Gene Increases Virulence of the Emerging Plant
bioRxiv preprint doi: https://doi.org/10.1101/681643; this version posted July 2, 2019. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY-NC-ND 4.0 International license. 1 1 A horizontally acquired expansin gene increases virulence of the emerging plant 2 pathogen Erwinia tracheiphila 3 Jorge Rochaa,b*#, Lori R. Shapiroa,c*, Roberto Koltera 4 5 #Address correspondence to Jorge Rocha, [email protected] 6 a Department of Microbiology, Harvard Medical School, Boston MA. 7 b Present Address: Conacyt-Centro de Investigación y Desarrollo en Agrobiotecnología 8 Alimentaria, San Agustin Tlaxiaca, Mexico 9 c Present Address: Department of Applied Ecology, North Carolina State University, 10 Raleigh, NC 11 12 * JR and LRS contributed equally to this work. 13 Word count: Abstract 219, Main Text (Introduction, Results, Discussion) 4354 14 15 Running title: An expansin increases Erwinia tracheiphila virulence 16 #Address correspondence to Jorge Rocha, [email protected] 17 Keywords: expansin, virulence, glycoside hydrolase, Cucurbita, Erwinia, squash, plant 18 cell wall, cellulose, pectin, horizontal gene transfer, plant pathogen, xylem 19 20 Author Contributions: JR and LRS conceived of the study. JR designed and conducted 21 molecular protocols and lab experiments. LRS conducted computational analyses and 22 performed experiments. JR, LRS and RK interpreted experimental data. JR and LRS 23 wrote the first draft of the manuscript, and JR, LRS and RK added critical revisions. bioRxiv preprint doi: https://doi.org/10.1101/681643; this version posted July 2, 2019. -

The Type VI Secretion System of Xanthomonas Phaseoli Pv
Montenegro Benavides et al. BMC Microbiology (2021) 21:14 https://doi.org/10.1186/s12866-020-02066-1 RESEARCH ARTICLE Open Access The type VI secretion system of Xanthomonas phaseoli pv. manihotis is involved in virulence and in vitro motility Nathaly Andrea Montenegro Benavides1, Alejandro Alvarez B.1, Mario L. Arrieta-Ortiz2, Luis Miguel Rodriguez-R3, David Botero1, Javier Felipe Tabima4, Luisa Castiblanco1, Cesar Trujillo1, Silvia Restrepo1 and Adriana Bernal1* Abstract Background: The type VI protein secretion system (T6SS) is important in diverse cellular processes in Gram- negative bacteria, including interactions with other bacteria and with eukaryotic hosts. In this study we analyze the evolution of the T6SS in the genus Xanthomonas and evaluate its importance of the T6SS for virulence and in vitro motility in Xanthomonas phaseoli pv. manihotis (Xpm), the causal agent of bacterial blight in cassava (Manihot esculenta). We delineate the organization of the T6SS gene clusters in Xanthomonas and then characterize proteins of this secretion system in Xpm strain CIO151. Results: We describe the presence of three different clusters in the genus Xanthomonas that vary in their organization and degree of synteny between species. Using a gene knockout strategy, we also found that vgrG and hcp are required for maximal aggressiveness of Xpm on cassava plants while clpV is important for both motility and maximal aggressiveness. Conclusion: We characterized the T6SS in 15 different strains in Xanthomonas and our phylogenetic analyses suggest that the T6SS might have been acquired by a very ancient event of horizontal gene transfer and maintained through evolution, hinting at their importance for the adaptation of Xanthomonas to their hosts. -

Genome Mining Reveals the Genus
Genome mining reveals the genus Xanthomonas to be a promising reservoir for new bioactive non-ribosomally synthesized peptides Monique Royer, Ralf Koebnik, Melanie Marguerettaz, Valerie Barbe, Guillaume P. Robin, Chrystelle Brin, Sebastien Carrere, Camila Gomez, Manuela Huegelland, Ginka H. Voeller, et al. To cite this version: Monique Royer, Ralf Koebnik, Melanie Marguerettaz, Valerie Barbe, Guillaume P. Robin, et al.. Genome mining reveals the genus Xanthomonas to be a promising reservoir for new bioactive non- ribosomally synthesized peptides. BMC Genomics, BioMed Central, 2013, 14, 10.1186/1471-2164-14- 658. hal-01209938 HAL Id: hal-01209938 https://hal.archives-ouvertes.fr/hal-01209938 Submitted on 29 May 2020 HAL is a multi-disciplinary open access L’archive ouverte pluridisciplinaire HAL, est archive for the deposit and dissemination of sci- destinée au dépôt et à la diffusion de documents entific research documents, whether they are pub- scientifiques de niveau recherche, publiés ou non, lished or not. The documents may come from émanant des établissements d’enseignement et de teaching and research institutions in France or recherche français ou étrangers, des laboratoires abroad, or from public or private research centers. publics ou privés. Royer et al. BMC Genomics 2013, 14:658 http://www.biomedcentral.com/1471-2164/14/658 RESEARCH ARTICLE Open Access Genome mining reveals the genus Xanthomonas to be a promising reservoir for new bioactive non-ribosomally synthesized peptides Monique Royer1, Ralf Koebnik2, Mélanie Marguerettaz1, Valérie Barbe3, Guillaume P Robin2, Chrystelle Brin4, Sébastien Carrere5, Camila Gomez1, Manuela Hügelland6, Ginka H Völler6, Julie Noëll1, Isabelle Pieretti1, Saskia Rausch6, Valérie Verdier2, Stéphane Poussier7, Philippe Rott1, Roderich D Süssmuth6 and Stéphane Cociancich1* Abstract Background: Various bacteria can use non-ribosomal peptide synthesis (NRPS) to produce peptides or other small molecules. -

Eukaryota Fungi Bacteria
Streptomyces-antibioticus Streptomyces-griseus Streptomyces-lincolnensis Streptomyces-phaeochromogenes Streptomyces-scabiei Streptomyces-violaceoruber Streptomyces-viridochromogenes Streptomyces-griseoruber Streptomyces-sclerotialus Streptomyces-olivochromogenes Streptomyces-cellulosae Streptomyces-chartreusis Streptomyces-cattleya Streptomyces-galbus Streptomyces-caelestis Streptomyces-subrutilus Streptomyces-acidiscabies Streptomyces-diastatochromogenes Streptomyces-neyagawaensis Streptomyces-canus Streptomyces-yerevanensis Streptomyces-afghaniensis Streptomyces-bicolor Streptomyces-cellostaticus Streptomyces-griseorubiginosus Streptomyces-phaeopurpureus Streptomyces-prasinus Streptomyces-prunicolor Streptomyces-pseudovenezuelae Streptomyces-resistomycificus Streptomyces Streptomyces-iakyrus Streptomyces-katrae Streptomyces-mirabilis Streptomyces-peruviensis Streptomyces-regalis Streptomyces-torulosus Streptomyces-turgidiscabies Streptomyces-ipomoeae Streptomyces-phaeoluteigriseus Streptomyces-curacoi Streptomyces-azureus Streptomyces-europaeiscabiei Streptomyces-stelliscabiei Streptomyces-puniciscabiei Streptomyces-aureus Streptomyces-ossamyceticus Streptomyces-variegatus Streptomyces-roseochromogenus Streptomyces-xylophagus Streptomyces-sviceus Streptomyces-fulvoviolaceus Streptomyces-bungoensis Streptomyces-erythrochromogenes Streptomycetaceae Streptomyces-davaonensis Streptomyces-olindensis Streptomyces-rubellomurinus Streptomyces-indicus Streptomyces-caeruleatus Streptomyces-hokutonensis Streptomyces-humi Streptomyces-jeddahensis -

Molecular Characterisation of the Causal Agent of Bacterial Leaf Streak of Maize
Molecular characterisation of the causal agent of bacterial leaf streak of maize NJJ Niemann 21114900 Dissertation submitted in fulfilment of the requirements for the degree Magister Scientiae in Environmental Sciences at the Potchefstroom Campus of the North-West University Supervisor: Prof CC Bezuidenhout Co-supervisor: Prof BC Flett May 2015 Declaration I declare that this dissertation submitted for the degree of Master of Science in Environmental Sciences at the North-West University, Potchefstroom Campus, has not been previously submitted by me for a degree at this or any other university, that it is my own work in design and execution, and that all material contained herein has been duly acknowledged. __________________________ __________________ NJJ Niemann Date ii Acknowledgements Thank you God for giving me the strength and will to complete this dissertation. I would like to thank the following people: My father, mother and brother for all their contributions and encouragement. My family and friends for their constant words of motivation. My supervisors for their support and providing me with the platform to work independently. Stefan Barnard for his input and patience with the construction of maps. Dr Gupta for his technical assistance. Thanks to the following organisations: The Maize Trust, the ARC and the NRF for their financial support of this research. iii Abstract All members of the genus Xanthomonas are considered to be plant pathogenic, with specific pathovars infecting several high value agricultural crops. One of these pathovars, X. campestris pv. zeae (as this is only a proposed name it will further on be referred to as Xanthomonas BLSD) the causal agent of bacterial leaf steak of maize, has established itself as a widespread significant maize pathogen within South Africa. -

Diversité Des Populations De Xanthomonas Phaseoli Pv
Diversité des populations de Xanthomonas phaseoli pv. manihotis au Mali et recherche de sources de résistance durables chez le manioc Moussa Kante To cite this version: Moussa Kante. Diversité des populations de Xanthomonas phaseoli pv. manihotis au Mali et recherche de sources de résistance durables chez le manioc. Sciences agricoles. Université Montpellier; Université de Bamako. Institut Supérieur de Formation et de Recherche Appliquée, 2020. Français. NNT : 2020MONTG028. tel-03157340 HAL Id: tel-03157340 https://tel.archives-ouvertes.fr/tel-03157340 Submitted on 3 Mar 2021 HAL is a multi-disciplinary open access L’archive ouverte pluridisciplinaire HAL, est archive for the deposit and dissemination of sci- destinée au dépôt et à la diffusion de documents entific research documents, whether they are pub- scientifiques de niveau recherche, publiés ou non, lished or not. The documents may come from émanant des établissements d’enseignement et de teaching and research institutions in France or recherche français ou étrangers, des laboratoires abroad, or from public or private research centers. publics ou privés. THÈSE POUR OBTENIR LE GRADE DE DOCTEUR DE L’UNIVERSITÉ DE M ONTPELLIER En Mécanisme des Interactions Parasitaires École doctorale GAIA Unité de recherche IPME En partenariat international avec IPU Ex-ISFRA & Université de Ségou, MALI Diversité des populations de Xanthomonas phaseoli pv manihotis (Xpm ) au Mali et recherche de source de résistance durable chez le manioc. Présentée par Moussa KANTE Rapport de gestion Le 17 Décembre 2020 Sous la direction de Boris SZUREK, Directeur de thèse et Ousmane KOITA, Co-Directeur de thèse 2015 Devant le jury composé de Dr Haby SANOU, Directeur de Recherche, Institut d’Economie Rurale/IER -Bamako Présidente Prof.