C Tin O M Y C E T O L O G Ic
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Within-Arctic Horizontal Gene Transfer As a Driver of Convergent Evolution in Distantly Related 2 Microalgae
bioRxiv preprint doi: https://doi.org/10.1101/2021.07.31.454568; this version posted August 2, 2021. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY-NC-ND 4.0 International license. 1 Within-Arctic horizontal gene transfer as a driver of convergent evolution in distantly related 2 microalgae 3 Richard G. Dorrell*+1,2, Alan Kuo3*, Zoltan Füssy4, Elisabeth Richardson5,6, Asaf Salamov3, Nikola 4 Zarevski,1,2,7 Nastasia J. Freyria8, Federico M. Ibarbalz1,2,9, Jerry Jenkins3,10, Juan Jose Pierella 5 Karlusich1,2, Andrei Stecca Steindorff3, Robyn E. Edgar8, Lori Handley10, Kathleen Lail3, Anna Lipzen3, 6 Vincent Lombard11, John McFarlane5, Charlotte Nef1,2, Anna M.G. Novák Vanclová1,2, Yi Peng3, Chris 7 Plott10, Marianne Potvin8, Fabio Rocha Jimenez Vieira1,2, Kerrie Barry3, Joel B. Dacks5, Colomban de 8 Vargas2,12, Bernard Henrissat11,13, Eric Pelletier2,14, Jeremy Schmutz3,10, Patrick Wincker2,14, Chris 9 Bowler1,2, Igor V. Grigoriev3,15, and Connie Lovejoy+8 10 11 1 Institut de Biologie de l'ENS (IBENS), Département de Biologie, École Normale Supérieure, CNRS, 12 INSERM, Université PSL, 75005 Paris, France 13 2CNRS Research Federation for the study of Global Ocean Systems Ecology and Evolution, 14 FR2022/Tara Oceans GOSEE, 3 rue Michel-Ange, 75016 Paris, France 15 3 US Department of Energy Joint Genome Institute, Lawrence Berkeley National Laboratory, 1 16 Cyclotron Road, Berkeley, -
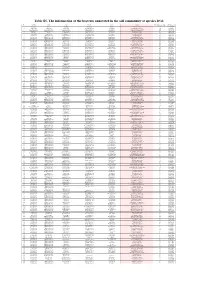
Table S5. the Information of the Bacteria Annotated in the Soil Community at Species Level
Table S5. The information of the bacteria annotated in the soil community at species level No. Phylum Class Order Family Genus Species The number of contigs Abundance(%) 1 Firmicutes Bacilli Bacillales Bacillaceae Bacillus Bacillus cereus 1749 5.145782459 2 Bacteroidetes Cytophagia Cytophagales Hymenobacteraceae Hymenobacter Hymenobacter sedentarius 1538 4.52499338 3 Gemmatimonadetes Gemmatimonadetes Gemmatimonadales Gemmatimonadaceae Gemmatirosa Gemmatirosa kalamazoonesis 1020 3.000970902 4 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas indica 797 2.344876284 5 Firmicutes Bacilli Lactobacillales Streptococcaceae Lactococcus Lactococcus piscium 542 1.594633558 6 Actinobacteria Thermoleophilia Solirubrobacterales Conexibacteraceae Conexibacter Conexibacter woesei 471 1.385742446 7 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas taxi 430 1.265115184 8 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas wittichii 388 1.141545794 9 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas sp. FARSPH 298 0.876754244 10 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sorangium cellulosum 260 0.764953367 11 Proteobacteria Deltaproteobacteria Myxococcales Polyangiaceae Sorangium Sphingomonas sp. Cra20 260 0.764953367 12 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas panacis 252 0.741416341 -

Within-Arctic Horizontal Gene Transfer As a Driver of Convergent Evolution in Distantly Related 1 Microalgae 2 Richard G. Do
bioRxiv preprint doi: https://doi.org/10.1101/2021.07.31.454568; this version posted August 2, 2021. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY-NC-ND 4.0 International license. 1 Within-Arctic horizontal gene transfer as a driver of convergent evolution in distantly related 2 microalgae 3 Richard G. Dorrell*+1,2, Alan Kuo3*, Zoltan Füssy4, Elisabeth Richardson5,6, Asaf Salamov3, Nikola 4 Zarevski,1,2,7 Nastasia J. Freyria8, Federico M. Ibarbalz1,2,9, Jerry Jenkins3,10, Juan Jose Pierella 5 Karlusich1,2, Andrei Stecca Steindorff3, Robyn E. Edgar8, Lori Handley10, Kathleen Lail3, Anna Lipzen3, 6 Vincent Lombard11, John McFarlane5, Charlotte Nef1,2, Anna M.G. Novák Vanclová1,2, Yi Peng3, Chris 7 Plott10, Marianne Potvin8, Fabio Rocha Jimenez Vieira1,2, Kerrie Barry3, Joel B. Dacks5, Colomban de 8 Vargas2,12, Bernard Henrissat11,13, Eric Pelletier2,14, Jeremy Schmutz3,10, Patrick Wincker2,14, Chris 9 Bowler1,2, Igor V. Grigoriev3,15, and Connie Lovejoy+8 10 11 1 Institut de Biologie de l'ENS (IBENS), Département de Biologie, École Normale Supérieure, CNRS, 12 INSERM, Université PSL, 75005 Paris, France 13 2CNRS Research Federation for the study of Global Ocean Systems Ecology and Evolution, 14 FR2022/Tara Oceans GOSEE, 3 rue Michel-Ange, 75016 Paris, France 15 3 US Department of Energy Joint Genome Institute, Lawrence Berkeley National Laboratory, 1 16 Cyclotron Road, Berkeley, -

Studies on Antibacterial Activity and Diversity of Cultivable Actinobacteria Isolated from Mangrove Soil in Futian and Maoweihai of China
Hindawi Evidence-Based Complementary and Alternative Medicine Volume 2019, Article ID 3476567, 11 pages https://doi.org/10.1155/2019/3476567 Research Article Studies on Antibacterial Activity and Diversity of Cultivable Actinobacteria Isolated from Mangrove Soil in Futian and Maoweihai of China Feina Li,1 Shaowei Liu,1 Qinpei Lu,1 Hongyun Zheng,1,2 Ilya A. Osterman,3,4 Dmitry A. Lukyanov,3 Petr V. Sergiev,3,4 Olga A. Dontsova,3,4,5 Shuangshuang Liu,6 Jingjing Ye,1,2 Dalin Huang ,2 and Chenghang Sun 1 1 Institute of Medicinal Biotechnology, Chinese Academy of Medical Sciences & Peking Union Medical College, Beijing 100050, China 2College of Basic Medical Sciences, Guilin Medical University, Guilin 541004, China 3Center of Life Sciences, Skolkovo Institute of Science and Technology, Moscow 143025, Russia 4Department of Chemistry, A.N. Belozersky Institute of Physico-Chemical Biology, Lomonosov Moscow State University, Moscow 119992, Russia 5Shemyakin-Ovchinnikov Institute of Bioorganic Chemistry, Te Russian Academy of Sciences, Moscow 117997, Russia 6China Pharmaceutical University, Nanjing 210009, China Correspondence should be addressed to Dalin Huang; [email protected] and Chenghang Sun; [email protected] Received 29 March 2019; Accepted 21 May 2019; Published 9 June 2019 Guest Editor: Jayanta Kumar Patra Copyright © 2019 Feina Li et al. Tis is an open access article distributed under the Creative Commons Attribution License, which permits unrestricted use, distribution, and reproduction in any medium, provided the original work is properly cited. Mangrove is a rich and underexploited ecosystem with great microbial diversity for discovery of novel and chemically diverse antimicrobial compounds. Te goal of the study was to explore the pharmaceutical actinobacterial resources from mangrove soil and gain insight into the diversity and novelty of cultivable actinobacteria. -

The Role of the Surface Smear Microbiome in the Development of Defective Smear on Surface-Ripened Red-Smear Cheese
AIMS Microbiology, 4(4): 622–641. DOI: 10.3934/microbiol.2018.4.622 Received: 29 June 2018 Accepted: 20 September 2018 Published: 25 October 2018 http://www.aimspress.com/journal/microbiology Research article The role of the surface smear microbiome in the development of defective smear on surface-ripened red-smear cheese Jasmine S. Ritschard1,†, Lea Amato2,†, Yadhu Kumar3, Britta Müller3, Leo Meile2 and Markus Schuppler1,* 1 Laboratory of Food Microbiology, Institute of Food, Nutrition and Health, ETH Zurich, Schmelzbergstrasse 7, 8092 Zurich, Switzerland 2 Laboratory of Food Biotechnology, Institute of Food, Nutrition and Health, ETH Zurich, Schmelzbergstrasse 7, 8092 Zurich, Switzerland 3 Eurofins GATC Biotech AG, Jakob-Stadler-Platz 7, 78467 Konstanz, Germany * Correspondence: Email: [email protected]; Tel: +41446327862; Fax: +41446321266. † These two authors contributed equally. Abstract: The complex smear microbiota colonizing the surface of red-smear cheese fundamentally impacts the ripening process, appearance and shelf life of cheese. To decipher the prokaryotic composition of the cheese smear microbiome, the surface of a semi-hard surface ripened cheese was studied post-ripening by culture-based and culture-independent molecular approaches. The aim was to detect potential bacterial alterations in the composition of the cheese smear microbiota resulting from cheese storage in vacuum film-prepackaging, which is often accompanied by the development of a surface smear defect. Next-generation sequencing of amplified 16S rRNA gene fragments revealed an unexpected high diversity of a total of 132 different genera from the domains Bacteria and Archaea on the cheese surface. Beside typical smear organisms, our study revealed the presence of several microorganisms so far not associated with cheese, but related to milk, farm and cheese dairy environments. -

Unrecorded Bacterial Species Belonging to the Phylum Actinobacteria Originated from Republic of Korea
Journal of Species Research 6(1):25-41, 2017 Unrecorded bacterial species belonging to the phylum Actinobacteria originated from Republic of Korea Mi-Sun Kim1, Ji-Hee Lee1, Seung-Bum Kim2, Jang-Cheon Cho3, Soon Dong Lee4, Ki-seong Joh5, Chang-Jun Cha6, Wan-Taek Im7, Jin-Woo Bae8, Kwangyeop Jahng9, Hana Yi10 and Chi-Nam Seong1,* 1Department of Biology, Sunchon National University, Suncheon 57922, Republic of Korea 2Department of Microbiology, Chungnam National University, Daejeon 34134, Republic of Korea 3Department of Biological Sciences, Inha University, Incheon 22212, Republic of Korea 4Department of Science Education, Jeju National University, Jeju 63243, Republic of Korea 5Department of Bioscience and Biotechnology, Hankuk University of Foreign Studies, Gyeonggi 17035, Republic of Korea 6Department of Biotechnology, Chung-Ang University, Anseong 17546, Republic of Korea 7Department of Biotechnology, Hankyong National University, Anseong 17579, Republic of Korea 8Department of Biology, Kyung Hee University, Seoul 02447, Republic of Korea 9Department of Life Sciences, Chonbuk National University, Jeonju 54896, Republic of Korea 10Department of Public Health Science & Guro Hospital, Korea University, Seoul 02841, Republic of Korea *Correspondent: [email protected] As a subset study for the collection of Korean indigenous prokaryotic species, 62 bacterial strains belonging to the phylum Actinobacteria were isolated from various sources. Each strain showed higher 16S rRNA gene sequence similarity (>98.75%) and formed a robust phylogenetic clade with closest species of the phylum Actinobacteria which were defined with valid names, already. There is no official description on these 62 actinobacterial species in Korea. Consequently, unrecorded 62 species of 25 genera in the 14 families belonging to the order Actinomycetales of the phylum Actinobacteria were found in Korea. -

Humibacter Aquilariae
Humibacter aquilariae Definition of the noun Humibacter. What does Humibacter mean as a name of something? Humibacter is a genus of Microbacteriaceae, described by Vaz-Moreira & al. in 2008. Online dictionaries and encyclopedias with entries for Humibacter. Click on a label to prioritize search results according to that topic: Scrabble value of H4U1M3I1B3A1C3T1E1R1. Filimonas aquilariae sp. nov., isolated from agarwood chips. Int J Syst Evol Microbiol. 2017 Sep; 67(9):3219-3225. Humibacter aquilariae sp. nov., an actinobacterium isolated from an agarwood chip. Int J Syst Evol Microbiol. 2017 May; 67(5):1468-1472. Humibacter. Quite the same Wikipedia. Just better. Humibacter is a Gram-positive, mesophilic, strictly aerobic, chemoorganotrophic and motile genus of bacteria from the family of Microbacteriaceae.[2][1][3][4] Humibacter occur in sewage sludge.[4]. References. ^ a b c d e f g h Parte, A.C. "Humibacter". www.bacterio.net. ^ Falkiewicz-Dulik, Michalina; Janda, Katarzyna; Wypych, George (2015). Handbook of Material Biodegradation, Biodeterioration, and Biostablization. Humibacter aquilariae sp. nov., an actinobacterium isolated from an agarwood chip. Shih-Yao Lin, Asif Abdul Hameed, +4 authors C. Young. International journal of systematic and⦠2017. A polyphasic taxonomic approach was used to characterize a presumably novel bacterium, designated strain CC-YTH161T, isolated from an agarwood sample. Cells of strain CC-YTH161T were⦠(More). Scientific name i. Humibacter. Taxonomy navigation. ⺠Microbacteriaceae. Choose one > Humibacter albus > Humibacter antri > Humibacter aquilariae > Humibacter ginsengisoli > Humibacter ginsengiterrae > Humibacter soli > Humibacter sp. BT305 > Humibacter sp. O406 > Humibacter sp. Po15c > Humibacter sp. SO3. All lower taxonomy nodes (11). Genus Humibacter. -

Of Bergey's Manual
BERGEY’S MANUAL® OF Systematic Bacteriology Second Edition Volume Five The Actinobacteria, Part A and B BERGEY’S MANUAL® OF Systematic Bacteriology Second Edition Volume Five The Actinobacteria, Part A and B Michael Goodfellow, Peter Kämpfer, Hans-Jürgen Busse, Martha E. Trujillo, Ken-ichiro Suzuki, Wolfgang Ludwig and William B. Whitman EDITORS, VOLUME FIVE William B. Whitman DIRECTOR OF THE EDITORIAL OFFICE Aidan C. Parte MANAGING EDITOR EDITORIAL BOARD Fred A. Rainey, Chairman, Peter Kämpfer, Vice Chairman, Paul De Vos, Jongsik Chun, Martha E. Trujillo and William B. Whitman WITH CONTRIBUTIONS FROM 116 COLLEAGUES William B. Whitman Bergey’s Manual Trust Department of Microbiology 527 Biological Sciences Building University of Georgia Athens, GA 30602-2605 USA ISBN 978-0-387-95043-3 ISBN 978-0-387-68233-4 (eBook) DOI 10.1007/978-0-387-68233-4 Springer New York Dordrecht Heidelberg London Library of Congress Control Number: 2012930836 © 2012, 1984–1989 Bergey’s Manual Trust Bergey’s Manual is a registered trademark of Bergey’s Manual Trust. All rights reserved. This work may not be translated or copied in whole or in part without the written permission of the publisher (Springer Science+Business Media, LLC, 233 Spring Street, New York, NY 10013, USA), except for brief excerpts in connection with reviews or scholarly analysis. Use in connection with any form of information storage and retrieval, electronic adaptation, computer software, or by similar or dissimilar methodology now known or hereafter developed is forbidden. The use in this publication of trade names, trademarks, service marks, and similar terms, even if they are not identified as such, is not to be taken as an expression of opinion as to whether or not they are subject to proprietary rights. -

Humibacter Albus Gen. Nov., Sp. Nov., Isolated from Sewage Sludge Compost
Humibacter albus gen. nov., sp. nov., isolated from sewage sludge compost Ivone Vaz-Moreira,1,2 M. Fernanda Nobre,3 Anto´nio C. Silva Ferreira,1 Peter Schumann,4 Olga C. Nunes2 and Ce´lia M. Manaia1 Correspondence 1Escola Superior de Biotecnologia, Universidade Cato´lica Portuguesa, 4200-072 Porto, Portugal Ce´lia M. Manaia 2LEPAE – Departamento de Engenharia Quı´mica, Faculdade de Engenharia, Universidade do Porto, [email protected] 4200-465 Porto, Portugal 3Departamento de Zoologia, Universidade de Coimbra, 3004-517 Coimbra, Portugal 4DSMZ-Deutsche Sammlung von Mikroorganismen und Zellkulturen GmbH, Inhoffenstraße 7B, D-38124 Braunschweig, Germany A bacterial strain isolated from sewage sludge compost, strain SC-083T, was characterized. The isolate was a motile, Gram-positive, short rod, forming coryneform V-shaped cells during the early stages of growth. The organism was strictly aerobic and able to grow between 22 and 36 6C and between pH 5.5 and 8.0. The predominant fatty acids were cyclohexyl-C17 : 0, anteiso-C17 : 0 and iso-C16 : 0, the major respiratory quinones were menaquinone 11 (MK-11) and 12 (MK-12), and the genomic DNA G+C content was 68 mol%. The peptidoglycan contained the diagnostic diamino acids ornithine and 2,4-diaminobutyric acid and was of acetyl type. The 16S rRNA gene sequence analysis indicated that this isolate belongs to the family Microbacteriaceae with the type strains of the species Leifsonia xyli (96 % gene sequence similarity), Leifsonia shinshuensis (96 %), Leifsonia naganoensis (95 %), Leifsonia aquatica (95 %), Agromyces ramosus (95 %) and Curtobacterium citreum (95 %) among the closest phylogenetic neighbours. The phylogenetic analysis and phenetic characteristics support the proposal of a new genus and a novel species, with the name Humibacter albus gen. -

Contents Topic 1. Introduction to Microbiology. the Subject and Tasks
Contents Topic 1. Introduction to microbiology. The subject and tasks of microbiology. A short historical essay………………………………………………………………5 Topic 2. Systematics and nomenclature of microorganisms……………………. 10 Topic 3. General characteristics of prokaryotic cells. Gram’s method ………...45 Topic 4. Principles of health protection and safety rules in the microbiological laboratory. Design, equipment, and working regimen of a microbiological laboratory………………………………………………………………………….162 Topic 5. Physiology of bacteria, fungi, viruses, mycoplasmas, rickettsia……...185 TOPIC 1. INTRODUCTION TO MICROBIOLOGY. THE SUBJECT AND TASKS OF MICROBIOLOGY. A SHORT HISTORICAL ESSAY. Contents 1. Subject, tasks and achievements of modern microbiology. 2. The role of microorganisms in human life. 3. Differentiation of microbiology in the industry. 4. Communication of microbiology with other sciences. 5. Periods in the development of microbiology. 6. The contribution of domestic scientists in the development of microbiology. 7. The value of microbiology in the system of training veterinarians. 8. Methods of studying microorganisms. Microbiology is a science, which study most shallow living creatures - microorganisms. Before inventing of microscope humanity was in dark about their existence. But during the centuries people could make use of processes vital activity of microbes for its needs. They could prepare a koumiss, alcohol, wine, vinegar, bread, and other products. During many centuries the nature of fermentations remained incomprehensible. Microbiology learns morphology, physiology, genetics and microorganisms systematization, their ecology and the other life forms. Specific Classes of Microorganisms Algae Protozoa Fungi (yeasts and molds) Bacteria Rickettsiae Viruses Prions The Microorganisms are extraordinarily widely spread in nature. They literally ubiquitous forward us from birth to our death. Daily, hourly we eat up thousands and thousands of microbes together with air, water, food. -

Metabolic Roles of Uncultivated Bacterioplankton Lineages in the Northern Gulf of Mexico 2 “Dead Zone” 3 4 J
bioRxiv preprint doi: https://doi.org/10.1101/095471; this version posted June 12, 2017. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY-NC 4.0 International license. 1 Metabolic roles of uncultivated bacterioplankton lineages in the northern Gulf of Mexico 2 “Dead Zone” 3 4 J. Cameron Thrash1*, Kiley W. Seitz2, Brett J. Baker2*, Ben Temperton3, Lauren E. Gillies4, 5 Nancy N. Rabalais5,6, Bernard Henrissat7,8,9, and Olivia U. Mason4 6 7 8 1. Department of Biological Sciences, Louisiana State University, Baton Rouge, LA, USA 9 2. Department of Marine Science, Marine Science Institute, University of Texas at Austin, Port 10 Aransas, TX, USA 11 3. School of Biosciences, University of Exeter, Exeter, UK 12 4. Department of Earth, Ocean, and Atmospheric Science, Florida State University, Tallahassee, 13 FL, USA 14 5. Department of Oceanography and Coastal Sciences, Louisiana State University, Baton Rouge, 15 LA, USA 16 6. Louisiana Universities Marine Consortium, Chauvin, LA USA 17 7. Architecture et Fonction des Macromolécules Biologiques, CNRS, Aix-Marseille Université, 18 13288 Marseille, France 19 8. INRA, USC 1408 AFMB, F-13288 Marseille, France 20 9. Department of Biological Sciences, King Abdulaziz University, Jeddah, Saudi Arabia 21 22 *Correspondence: 23 JCT [email protected] 24 BJB [email protected] 25 26 27 28 Running title: Decoding microbes of the Dead Zone 29 30 31 Abstract word count: 250 32 Text word count: XXXX 33 34 Page 1 of 31 bioRxiv preprint doi: https://doi.org/10.1101/095471; this version posted June 12, 2017. -

Aerobic Hydrocarbon-Degrading Microbial Communities in Oilsands Tailings Ponds
University of Calgary PRISM: University of Calgary's Digital Repository Graduate Studies The Vault: Electronic Theses and Dissertations 2016 Aerobic Hydrocarbon-degrading Microbial Communities in Oilsands Tailings Ponds Rochman, Fauziah Rochman, F. (2016). Aerobic Hydrocarbon-degrading Microbial Communities in Oilsands Tailings Ponds (Unpublished doctoral thesis). University of Calgary, Calgary, AB. doi:10.11575/PRISM/24733 http://hdl.handle.net/11023/3508 doctoral thesis University of Calgary graduate students retain copyright ownership and moral rights for their thesis. You may use this material in any way that is permitted by the Copyright Act or through licensing that has been assigned to the document. For uses that are not allowable under copyright legislation or licensing, you are required to seek permission. Downloaded from PRISM: https://prism.ucalgary.ca UNIVERSITY OF CALGARY Aerobic Hydrocarbon-degrading Microbial Communities in Oilsands Tailings Ponds by Fauziah Fakhrunnisa Rochman A THESIS SUBMITTED TO THE FACULTY OF GRADUATE STUDIES IN PARTIAL FULFILMENT OF THE REQUIREMENTS FOR THE DEGREE OF DOCTOR OF PHILOSOPHY GRADUATE PROGRAM IN BIOLOGICAL SCIENCES CALGARY, ALBERTA DECEMBER, 2016 © Fauziah Fakhrunnisa Rochman 2016 Abstract Oilsands process-affected water (OSPW), produced by the surface-mining oilsands industry in Alberta, Canada, is alkaline and contains salts, various metals, and hydrocarbon compounds. In this thesis, aerobic communities involved in several key biogeochemical processes in OSPW were studied. Degradation of several key hydrocarbons was analyzed in depth. Benzene and naphthalene were used as models for aromatic hydrocarbons, in which their oxidation rates, degrading communities, and degradation pathways in OSPW were researched. The potential oxidation rates were 36.7 μmol L-1 day-1 for benzene and 85.4 μmol L-1 day-1 for naphthalene.