A Framework for Gene Co-Expression Network Analysis of Lung Cancer
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Inhibition of Breast and Brain Cancer Cell Growth by Bccipα, An
Oncogene (2001) 20, 336 ± 345 ã 2001 Nature Publishing Group All rights reserved 0950 ± 9232/01 $15.00 www.nature.com/onc Inhibition of breast and brain cancer cell growth by BCCIPa,an evolutionarily conserved nuclear protein that interacts with BRCA2 Jingmei Liu1, Yuan Yuan1,2, Juan Huan2 and Zhiyuan Shen*,1 1Department of Molecular Genetics and Microbiology, University of New Mexico Health Sciences Center; 915 Camino de Salud, NE. Albuquerque, New Mexico, NM 87131, USA; 2Graduate Program of Molecular Genetics, College of Medicine, University of Illinois at Chicago, 900 S. Ashland Ave. Chicago, Illinois, IL 60607, USA BRCA2 is a tumor suppressor gene involved in mammary mouse BRCA2. It is expected that important functions tumorigenesis. Although important functions have been of BRCA2 reside in these conserved domains. Based on assigned to a few conserved domains of BRCA2, little is the functional analysis of the conserved BRCA2 known about the longest internal conserved domain domains, several models have been proposed for the encoded by exons 14 ± 24. We identi®ed a novel protein, role of BRCA2 in tumor suppression. designated BCCIPa, that interacts with part of the An N-terminus conserved domain in exon 3 (amino internal conserved region of human BRCA2. Human acids 48 ± 105) has been implicated in transcriptional BCCIP represents a family of proteins that are regulation of gene expression (Milner et al., 1997; evolutionarily conserved, and contain three distinct Nordling et al., 1998). Deletion of this region has been domains: an N-terminus acidic domain (NAD) of 30 ± identi®ed in breast cancers (Nordling et al., 1998). -

Analysis of Trans Esnps Infers Regulatory Network Architecture
Analysis of trans eSNPs infers regulatory network architecture Anat Kreimer Submitted in partial fulfillment of the requirements for the degree of Doctor of Philosophy in the Graduate School of Arts and Sciences COLUMBIA UNIVERSITY 2014 © 2014 Anat Kreimer All rights reserved ABSTRACT Analysis of trans eSNPs infers regulatory network architecture Anat Kreimer eSNPs are genetic variants associated with transcript expression levels. The characteristics of such variants highlight their importance and present a unique opportunity for studying gene regulation. eSNPs affect most genes and their cell type specificity can shed light on different processes that are activated in each cell. They can identify functional variants by connecting SNPs that are implicated in disease to a molecular mechanism. Examining eSNPs that are associated with distal genes can provide insights regarding the inference of regulatory networks but also presents challenges due to the high statistical burden of multiple testing. Such association studies allow: simultaneous investigation of many gene expression phenotypes without assuming any prior knowledge and identification of unknown regulators of gene expression while uncovering directionality. This thesis will focus on such distal eSNPs to map regulatory interactions between different loci and expose the architecture of the regulatory network defined by such interactions. We develop novel computational approaches and apply them to genetics-genomics data in human. We go beyond pairwise interactions to define network motifs, including regulatory modules and bi-fan structures, showing them to be prevalent in real data and exposing distinct attributes of such arrangements. We project eSNP associations onto a protein-protein interaction network to expose topological properties of eSNPs and their targets and highlight different modes of distal regulation. -

Statistical Methods for High-Dimensional Networked Data Analysis
Statistical Methods for High-Dimensional Networked Data Analysis by Yan Zhou A dissertation submitted in partial fulfillment of the requirements for the degree of Doctor of Philosophy (Biostatistics) in The University of Michigan 2015 Doctoral Committee: Professor Peter X. K. Song, Chair Professor Matthias Kretzler Assistant Professor Xiaoquan William Wen Professor Ji Zhu c Yan Zhou 2015 All Rights Reserved To my family ii ACKNOWLEDGEMENTS I would like to acknowledge many people for their help throughout my graduate study. I would first like to express my deepest gratitude to my advisor Dr. Peter X.- K. Song, for sharing his knowledge, encouraging me to work hard and constantly try to improve my work, and giving me the freedom to explore a diverse set of projects. He is the one who first sparked my interest in networked data analysis. I am not only respectful for his immense knowledge, motivation, and enthusiasm towards research, but also appreciate his invaluable guidance and enthusiastic encouragement during many difficult moments in my research. I could not have imagined having a better advisor and mentor for my Ph.D. study. I would also like to thank my other thesis committee members Dr. Ji Zhu, Dr. Xiaoquan Willian Wen and Dr. Matthias Kretzler for their help and advices that made me more productive than what I could achieve on my own. All enlightening discussions I had with them have lead to better contents of this dissertation. In addition, I would like to acknowledge my collaborators, Dr. Pei Wang, Dr. Betsy Lozoff and Dr. Fengji Geng, who offered me excellent opportunities and con- structive suggestions to apply my statistical knowledge to solve real-world problems, which greatly strengthened the scientific background of my dissertation research. -
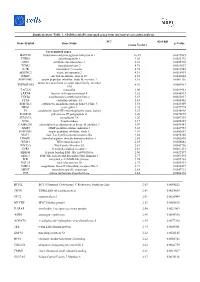
Supplementary Table 1. All Differentially Expressed Genes from Microarray Screening Analysis
Supplementary Table 1. All differentially expressed genes from microarray screening analysis. FCa (Gal-KD Gene Symbol Gene Name p-Value versus Vector) Up-regulated genes HAPLN1 hyaluronan and proteoglycan link protein 1 10.49 0.0027085 THBS1 thrombospondin 1 9.20 0.0022192 ODC1 ornithine decarboxylase 1 6.38 0.0055776 TGM2 transglutaminase 2 4.76 0.0015627 IL7R interleukin 7 receptor 4.75 0.0017245 SERINC2 serine incorporator 2 4.51 0.0014919 ITM2C integral membrane protein 2C 4.32 0.0044644 SERPINB7 serpin peptidase inhibitor, clade B, member 7 4.18 0.0081136 tumor necrosis factor receptor superfamily, member TNFRSF10D 4.01 0.0085561 10d TAGLN transgelin 3.90 0.0099963 LRRN4 leucine rich repeat neuronal 4 3.82 0.0046513 TGFB2 transforming growth factor beta 2 3.51 0.0035017 CPA4 carboxypeptidase A4 3.43 0.0008452 EPB41L3 erythrocyte membrane protein band 4.1-like 3 3.34 0.0025309 NRG1 neuregulin 1 3.28 0.0079724 F3 coagulation factor III (thromboplastin, tissue factor) 3.27 0.0038968 POLR3G polymerase III polypeptide G 3.26 0.0070675 SEMA7A semaphorin 7A 3.20 0.0087335 NT5E 5-nucleotidase 3.17 0.0036353 CAMK2N1 calmodulin-dependent protein kinase II inhibitor 1 3.07 0.0090141 TIMP3 TIMP metallopeptidase inhibitor 3 3.03 0.0047953 SERPINE1 serpin peptidase inhibitor, clade E 2.97 0.0053652 MALL mal, T-cell differentiation protein-like 2.88 0.0078205 DDAH1 dimethylarginine dimethylaminohydrolase 1 2.86 0.0002895 WDR3 WD repeat domain 3 2.85 0.0058842 WNT5A Wnt Family Member 5A 2.81 0.0043796 GPR1 G protein-coupled receptor 1 2.81 0.0021313 -

Efficient Zygotic Genome Editing Via RAD51-Enhanced Interhomolog Repair 2 3 Jonathan J
bioRxiv preprint doi: https://doi.org/10.1101/263699; this version posted August 6, 2018. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY-NC-ND 4.0 International license. 1 Efficient Zygotic Genome Editing via RAD51-Enhanced Interhomolog Repair 2 3 Jonathan J. Wilde1,3, Tomomi Aida1,3, Martin Wienisch1, Qiangge Zhang1, Peimin Qi1, and 4 Guoping Feng1,2* 5 6 Affiliations: 7 1McGovern Institute for Brain Research, Department of Brain and Cognitive Sciences, 8 Massachusetts Institute of Technology (MIT), Cambridge, Massachusetts 02139, USA 9 10 2Stanley Center for Psychiatric Research, Broad Institute of MIT and Harvard, Cambridge, 11 Massachusetts 02142, USA 12 13 3These authors contributed equally to this work. 14 15 *E-mail: [email protected] 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 bioRxiv preprint doi: https://doi.org/10.1101/263699; this version posted August 6, 2018. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY-NC-ND 4.0 International license. 47 Recent advances in genome editing have greatly improved knock-in (KI) efficiency1- 48 9. Searching for factors to further improve KI efficiency for therapeutic use and generation 49 of non-human primate (NHP) models, we found that the strand exchange protein RAD51 50 can significantly increase homozygous KI using CRISPR/Cas9 in mouse embryos through 51 an interhomolog repair (IHR) mechanism. -

Conservation of the BRCA1 Gene
Rochester Institute of Technology RIT Scholar Works Theses 1-2007 Conservation of the BRCA1 gene Matthew Gary Wronkowski Follow this and additional works at: https://scholarworks.rit.edu/theses Recommended Citation Wronkowski, Matthew Gary, "Conservation of the BRCA1 gene" (2007). Thesis. Rochester Institute of Technology. Accessed from This Thesis is brought to you for free and open access by RIT Scholar Works. It has been accepted for inclusion in Theses by an authorized administrator of RIT Scholar Works. For more information, please contact [email protected]. Conservation of the BRCAI Gene Approved: G.R.Skuse Gary R. Skuse, Ph.D. Director of Bioinformatics Richard L. Doolittle Richard L. Doolittle, Ph.D. Head, Department of Biological Sciences Submitted in partial fulfillment of the requirements for the Master of Science degree in Bioinformatics at the Rochester Institute of Technology. Matthew Wronkowski April 2007 Committee Members Anne Haake, Ph.D. is professor of information technology and bioinformatics courses at the Rochester Institute of Technology. Dina Newman, Ph.D. is a professor of human genomics and biology courses in the College of Science at the Rochester Institute of Technology. Maureen Ferran, Ph.D. is a professor of biology and virology in the College of Science at the Rochester Institute of Technology. Gary Skuse, Ph.D. is director of the Bioinformatics program at the Rochester Institute of Technology. Copyright Release Form Title of thesis: Conservation of the BRCAI Gene Name of author: Matthew Gary Wronkowski Degree: Masters of Science Program: Bioinformatics College: Science I understand that I must submit a print copy of my thesis to the RIT Archives, per current RIT guidelines for the completion of my degree. -

Downloaded the “Top Edge” Version
bioRxiv preprint doi: https://doi.org/10.1101/855338; this version posted December 6, 2019. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY 4.0 International license. 1 Drosophila models of pathogenic copy-number variant genes show global and 2 non-neuronal defects during development 3 Short title: Non-neuronal defects of fly homologs of CNV genes 4 Tanzeen Yusuff1,4, Matthew Jensen1,4, Sneha Yennawar1,4, Lucilla Pizzo1, Siddharth 5 Karthikeyan1, Dagny J. Gould1, Avik Sarker1, Yurika Matsui1,2, Janani Iyer1, Zhi-Chun Lai1,2, 6 and Santhosh Girirajan1,3* 7 8 1. Department of Biochemistry and Molecular Biology, Pennsylvania State University, 9 University Park, PA 16802 10 2. Department of Biology, Pennsylvania State University, University Park, PA 16802 11 3. Department of Anthropology, Pennsylvania State University, University Park, PA 16802 12 4 contributed equally to work 13 14 *Correspondence: 15 Santhosh Girirajan, MBBS, PhD 16 205A Life Sciences Building 17 Pennsylvania State University 18 University Park, PA 16802 19 E-mail: [email protected] 20 Phone: 814-865-0674 21 1 bioRxiv preprint doi: https://doi.org/10.1101/855338; this version posted December 6, 2019. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY 4.0 International license. 22 ABSTRACT 23 While rare pathogenic copy-number variants (CNVs) are associated with both neuronal and non- 24 neuronal phenotypes, functional studies evaluating these regions have focused on the molecular 25 basis of neuronal defects. -

Transcriptomic and Proteomic Profiling Provides Insight Into
BASIC RESEARCH www.jasn.org Transcriptomic and Proteomic Profiling Provides Insight into Mesangial Cell Function in IgA Nephropathy † † ‡ Peidi Liu,* Emelie Lassén,* Viji Nair, Celine C. Berthier, Miyuki Suguro, Carina Sihlbom,§ † | † Matthias Kretzler, Christer Betsholtz, ¶ Börje Haraldsson,* Wenjun Ju, Kerstin Ebefors,* and Jenny Nyström* *Department of Physiology, Institute of Neuroscience and Physiology, §Proteomics Core Facility at University of Gothenburg, University of Gothenburg, Gothenburg, Sweden; †Division of Nephrology, Department of Internal Medicine and Department of Computational Medicine and Bioinformatics, University of Michigan, Ann Arbor, Michigan; ‡Division of Molecular Medicine, Aichi Cancer Center Research Institute, Nagoya, Japan; |Department of Immunology, Genetics and Pathology, Uppsala University, Uppsala, Sweden; and ¶Integrated Cardio Metabolic Centre, Karolinska Institutet Novum, Huddinge, Sweden ABSTRACT IgA nephropathy (IgAN), the most common GN worldwide, is characterized by circulating galactose-deficient IgA (gd-IgA) that forms immune complexes. The immune complexes are deposited in the glomerular mesangium, leading to inflammation and loss of renal function, but the complete pathophysiology of the disease is not understood. Using an integrated global transcriptomic and proteomic profiling approach, we investigated the role of the mesangium in the onset and progression of IgAN. Global gene expression was investigated by microarray analysis of the glomerular compartment of renal biopsy specimens from patients with IgAN (n=19) and controls (n=22). Using curated glomerular cell type–specific genes from the published literature, we found differential expression of a much higher percentage of mesangial cell–positive standard genes than podocyte-positive standard genes in IgAN. Principal coordinate analysis of expression data revealed clear separation of patient and control samples on the basis of mesangial but not podocyte cell–positive standard genes. -

YY1/BCCIP Coordinately Regulates P53-Responsive Element (P53re)-Mediated Transactivation of P21waf1/Cip1
International Journal of Molecular Sciences Article YY1/BCCIP Coordinately Regulates P53-Responsive Element (p53RE)-Mediated Transactivation of p21Waf1/Cip1 Yi Sui 1, Tingting Wu 1, Fuqiang Li 2, Fei Wang 1, Yong Cai 1,3,4,* and Jingji Jin 1,3,4,* 1 Department of Biochemistry and Molecular Biology, School of Life Sciences, Jilin University, Changchun 130012, China; [email protected] (Y.S.); [email protected] (T.W.); [email protected] (F.W.) 2 School of Pharmacy, Changchun University of Chinese Medicine, Changchun 130117, China; [email protected] 3 National Engineering Laboratory for AIDS Vaccine, School of Life Sciences, Jilin University, Changchun 130012, China 4 Key Laboratory for Molecular Enzymology and Engineering, the Ministry of Education, Jilin University, Changchun 130012, China * Correspondence: [email protected] (Y.C.); [email protected] (J.J.); Tel.: +86-431-8515-5132 (Y.C.); +86-431-8515-5475 (J.J.) Received: 1 April 2019; Accepted: 26 April 2019; Published: 28 April 2019 Abstract: Transactivation of p21 (cyclin-dependent kinase inhibitor 1A, CDKN1A) is closely related to the recruitment of transcription cofactors at the p53 responsive elements (p53REs) in its promoter region. Human chromatin remodeling enzyme INO80 can be recruited to the p53REs of p21 promoter and negatively regulates p21. As one of the key subunits of the INO80 complex, YY1 has also been confirmed to bind to the p53RE sites of p21 promoter. Importantly, YY1 was recently reported to be bound and stabilized by BCCIP (BRCA2 and CDKN1A-interacting protein). Therefore, we hypothesized that the YY1/BCCIP complex plays an important role in regulating the transactivation of p21. -

Genetic Alterations of Protein Tyrosine Phosphatases in Human Cancers
Oncogene (2015) 34, 3885–3894 © 2015 Macmillan Publishers Limited All rights reserved 0950-9232/15 www.nature.com/onc REVIEW Genetic alterations of protein tyrosine phosphatases in human cancers S Zhao1,2,3, D Sedwick3,4 and Z Wang2,3 Protein tyrosine phosphatases (PTPs) are enzymes that remove phosphate from tyrosine residues in proteins. Recent whole-exome sequencing of human cancer genomes reveals that many PTPs are frequently mutated in a variety of cancers. Among these mutated PTPs, PTP receptor T (PTPRT) appears to be the most frequently mutated PTP in human cancers. Beside PTPN11, which functions as an oncogene in leukemia, genetic and functional studies indicate that most of mutant PTPs are tumor suppressor genes. Identification of the substrates and corresponding kinases of the mutant PTPs may provide novel therapeutic targets for cancers harboring these mutant PTPs. Oncogene (2015) 34, 3885–3894; doi:10.1038/onc.2014.326; published online 29 September 2014 INTRODUCTION tyrosine/threonine-specific phosphatases. (4) Class IV PTPs include Protein tyrosine phosphorylation has a critical role in virtually all four Drosophila Eya homologs (Eya1, Eya2, Eya3 and Eya4), which human cellular processes that are involved in oncogenesis.1 can dephosphorylate both tyrosine and serine residues. Protein tyrosine phosphorylation is coordinately regulated by protein tyrosine kinases (PTKs) and protein tyrosine phosphatases 1 THE THREE-DIMENSIONAL STRUCTURE AND CATALYTIC (PTPs). Although PTKs add phosphate to tyrosine residues in MECHANISM OF PTPS proteins, PTPs remove it. Many PTKs are well-documented oncogenes.1 Recent cancer genomic studies provided compelling The three-dimensional structures of the catalytic domains of evidence that many PTPs function as tumor suppressor genes, classical PTPs (RPTPs and non-RPTPs) are extremely well because a majority of PTP mutations that have been identified in conserved.5 Even the catalytic domain structures of the dual- human cancers are loss-of-function mutations. -

Biological Models of Colorectal Cancer Metastasis and Tumor Suppression
BIOLOGICAL MODELS OF COLORECTAL CANCER METASTASIS AND TUMOR SUPPRESSION PROVIDE MECHANISTIC INSIGHTS TO GUIDE PERSONALIZED CARE OF THE COLORECTAL CANCER PATIENT By Jesse Joshua Smith Dissertation Submitted to the Faculty of the Graduate School of Vanderbilt University In partial fulfillment of the requirements For the degree of DOCTOR OF PHILOSOPHY In Cell and Developmental Biology May, 2010 Nashville, Tennessee Approved: Professor R. Daniel Beauchamp Professor Robert J. Coffey Professor Mark deCaestecker Professor Ethan Lee Professor Steven K. Hanks Copyright 2010 by Jesse Joshua Smith All Rights Reserved To my grandparents, Gladys and A.L. Lyth and Juanda Ruth and J.E. Smith, fully supportive and never in doubt. To my amazing and enduring parents, Rebecca Lyth and Jesse E. Smith, Jr., always there for me. .my sure foundation. To Jeannine, Bill and Reagan for encouragement, patience, love, trust and a solid backing. To Granny George and Shawn for loving support and care. And To my beautiful wife, Kelly, My heart, soul and great love, Infinitely supportive, patient and graceful. ii ACKNOWLEDGEMENTS This work would not have been possible without the financial support of the Vanderbilt Medical Scientist Training Program through the Clinical and Translational Science Award (Clinical Investigator Track), the Society of University Surgeons-Ethicon Scholarship Fund and the Surgical Oncology T32 grant and the Vanderbilt Medical Center Section of Surgical Sciences and the Department of Surgical Oncology. I am especially indebted to Drs. R. Daniel Beauchamp, Chairman of the Section of Surgical Sciences, Dr. James R. Goldenring, Vice Chairman of Research of the Department of Surgery, Dr. Naji N. -

Primepcr™Assay Validation Report
PrimePCR™Assay Validation Report Gene Information Gene Name thymosin beta 15a Gene Symbol TMSB15A Organism Human Gene Summary Description Not Available Gene Aliases TMSB15, TMSB15B, TMSL8, TMSNB, Tb15, TbNB RefSeq Accession No. NC_000023.10, NT_011651.17 UniGene ID Hs.56145 Ensembl Gene ID ENSG00000158164 Entrez Gene ID 11013 Assay Information Unique Assay ID qHsaCED0045803 Assay Type SYBR® Green Detected Coding Transcript(s) ENST00000289373, ENST00000601407 Amplicon Context Sequence TTAAAACAGGCTTATCCAGTATAGGATGCAAGAACTACATACACAAAGGTCATCA ATGGTGAGATTATTGACAGCATCTGCCATCTGGAACATATGCACCAATGGTAAGT TTAAAAATGAATT Amplicon Length (bp) 93 Chromosome Location X:101768728-101768850 Assay Design Exonic Purification Desalted Validation Results Efficiency (%) 95 R2 0.9994 cDNA Cq 21.97 cDNA Tm (Celsius) 79 gDNA Cq 25.12 Specificity (%) 100 Information to assist with data interpretation is provided at the end of this report. Page 1/4 PrimePCR™Assay Validation Report TMSB15A, Human Amplification Plot Amplification of cDNA generated from 25 ng of universal reference RNA Melt Peak Melt curve analysis of above amplification Standard Curve Standard curve generated using 20 million copies of template diluted 10-fold to 20 copies Page 2/4 PrimePCR™Assay Validation Report Products used to generate validation data Real-Time PCR Instrument CFX384 Real-Time PCR Detection System Reverse Transcription Reagent iScript™ Advanced cDNA Synthesis Kit for RT-qPCR Real-Time PCR Supermix SsoAdvanced™ SYBR® Green Supermix Experimental Sample qPCR Human Reference Total RNA Data Interpretation Unique Assay ID This is a unique identifier that can be used to identify the assay in the literature and online. Detected Coding Transcript(s) This is a list of the Ensembl transcript ID(s) that this assay will detect. Details for each transcript can be found on the Ensembl website at www.ensembl.org.