Investigation of Cap-Independent Translation in Neuronal Differentiation
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Sequence Analysis and Comparative Study of the Protein Subunits of Archaeal Rnase P
biomolecules Review Sequence Analysis and Comparative Study of the Protein Subunits of Archaeal RNase P Manoj P. Samanta 1,*,†, Stella M. Lai 2,3,†, Charles J. Daniels 3,4 and Venkat Gopalan 2,3,* 1 Systemix Institute, Redmond, WA 98053, USA 2 Department of Chemistry & Biochemistry, The Ohio State University, Columbus, OH 43210, USA; [email protected] 3 Center for RNA Biology, The Ohio State University, Columbus, OH 43210, USA; [email protected] 4 Department of Microbiology, The Ohio State University, Columbus, OH 43210, USA * Correspondence: [email protected] (M.P.S.); [email protected] (V.G.); Tel.: +1-425-898-8187 (M.P.S.); +1-614-292-1332 (V.G.) † These authors contributed equally to this work. Academic Editor: Denis Drainas Received: 1 March 2016; Accepted: 8 April 2016; Published: 20 April 2016 Abstract: RNase P, a ribozyme-based ribonucleoprotein (RNP) complex that catalyzes tRNA 51-maturation, is ubiquitous in all domains of life, but the evolution of its protein components (RNase P proteins, RPPs) is not well understood. Archaeal RPPs may provide clues on how the complex evolved from an ancient ribozyme to an RNP with multiple archaeal and eukaryotic (homologous) RPPs, which are unrelated to the single bacterial RPP. Here, we analyzed the sequence and structure of archaeal RPPs from over 600 available genomes. All five RPPs are found in eight archaeal phyla, suggesting that these RPPs arose early in archaeal evolutionary history. The putative ancestral genomic loci of archaeal RPPs include genes encoding several members of ribosome, exosome, and proteasome complexes, which may indicate coevolution/coordinate regulation of RNase P with other core cellular machineries. -

Download on 20
bioRxiv preprint doi: https://doi.org/10.1101/850776; this version posted January 19, 2020. The copyright holder for this preprint (which was not certified by peer review) is the author/funder. All rights reserved. No reuse allowed without permission. Intramembrane protease RHBDL4 cleaves oligosaccharyltransferase subunits to target them for ER-associated degradation Julia D. Knopf1, Nina Landscheidt1, Cassandra L. Pegg2, Benjamin L. Schulz2, Nathalie Kühnle1, Chao-Wei Chao1, Simon Huck1 and Marius K. Lemberg1, # 1Centre for Molecular Biology of Heidelberg University (ZMBH), DKFZ-ZMBH Alliance, 69120 Heidelberg, Germany. 2School of Chemistry and Molecular Biosciences, ARC Training Centre for Biopharmaceutical Innovation, The University of Queensland, St Lucia QLD 4072, Australia. #Corresponding author: [email protected] Running title: RHBDL4 triggers ERAD of OST subunits Key words: Rhomboid serine protease, Rhbdd1, ubiquitin-dependent proteolysis, post- translational protein abundance control, N-linked glycosylation. Abbreviations ERAD, ER-associated degradation; OST, oligosacharyltransferase; TM, transmembrane; UIM, ubiquitin-interacting motif. Abstract The Endoplasmic Reticulum (ER)-resident intramembrane rhomboid protease RHBDL4 generates metastable protein fragments and together with the ER-associated degradation (ERAD) machinery provides a clearance mechanism for aberrant and surplus proteins. However, the endogenous substrate spectrum and with that the role of RHBDL4 in physiological ERAD is mainly unknown. Here, we use a substrate trapping approach in combination with quantitative proteomics to identify physiological RHBDL4 substrates. This revealed oligosacharyltransferase (OST) complex subunits such as the catalytic active subunit STT3A as substrates for the RHBDL4-dependent ERAD pathway. RHBDL4-catalyzed cleavage inactivates OST subunits by triggering dislocation into the cytoplasm and subsequent proteasomal degradation. -

Differential Expression Profile Prioritization of Positional Candidate Glaucoma Genes the GLC1C Locus
LABORATORY SCIENCES Differential Expression Profile Prioritization of Positional Candidate Glaucoma Genes The GLC1C Locus Frank W. Rozsa, PhD; Kathleen M. Scott, BS; Hemant Pawar, PhD; John R. Samples, MD; Mary K. Wirtz, PhD; Julia E. Richards, PhD Objectives: To develop and apply a model for priori- est because of moderate expression and changes in tization of candidate glaucoma genes. expression. Transcription factor ZBTB38 emerges as an interesting candidate gene because of the overall expres- Methods: This Affymetrix GeneChip (Affymetrix, Santa sion level, differential expression, and function. Clara, Calif) study of gene expression in primary cul- ture human trabecular meshwork cells uses a positional Conclusions: Only1geneintheGLC1C interval fits our differential expression profile model for prioritization of model for differential expression under multiple glau- candidate genes within the GLC1C genetic inclusion in- coma risk conditions. The use of multiple prioritization terval. models resulted in filtering 7 candidate genes of higher interest out of the 41 known genes in the region. Results: Sixteen genes were expressed under all condi- tions within the GLC1C interval. TMEM22 was the only Clinical Relevance: This study identified a small sub- gene within the interval with differential expression in set of genes that are most likely to harbor mutations that the same direction under both conditions tested. Two cause glaucoma linked to GLC1C. genes, ATP1B3 and COPB2, are of interest in the con- text of a protein-misfolding model for candidate selec- tion. SLC25A36, PCCB, and FNDC6 are of lesser inter- Arch Ophthalmol. 2007;125:117-127 IGH PREVALENCE AND PO- identification of additional GLC1C fami- tential for severe out- lies7,18-20 who provide optimal samples for come combine to make screening candidate genes for muta- adult-onset primary tions.7,18,20 The existence of 2 distinct open-angle glaucoma GLC1C haplotypes suggests that muta- (POAG) a significant public health prob- tions will not be limited to rare descen- H1 lem. -

Evidence for Differential Alternative Splicing in Blood of Young Boys With
Stamova et al. Molecular Autism 2013, 4:30 http://www.molecularautism.com/content/4/1/30 RESEARCH Open Access Evidence for differential alternative splicing in blood of young boys with autism spectrum disorders Boryana S Stamova1,2,5*, Yingfang Tian1,2,4, Christine W Nordahl1,3, Mark D Shen1,3, Sally Rogers1,3, David G Amaral1,3 and Frank R Sharp1,2 Abstract Background: Since RNA expression differences have been reported in autism spectrum disorder (ASD) for blood and brain, and differential alternative splicing (DAS) has been reported in ASD brains, we determined if there was DAS in blood mRNA of ASD subjects compared to typically developing (TD) controls, as well as in ASD subgroups related to cerebral volume. Methods: RNA from blood was processed on whole genome exon arrays for 2-4–year-old ASD and TD boys. An ANCOVA with age and batch as covariates was used to predict DAS for ALL ASD (n=30), ASD with normal total cerebral volumes (NTCV), and ASD with large total cerebral volumes (LTCV) compared to TD controls (n=20). Results: A total of 53 genes were predicted to have DAS for ALL ASD versus TD, 169 genes for ASD_NTCV versus TD, 1 gene for ASD_LTCV versus TD, and 27 genes for ASD_LTCV versus ASD_NTCV. These differences were significant at P <0.05 after false discovery rate corrections for multiple comparisons (FDR <5% false positives). A number of the genes predicted to have DAS in ASD are known to regulate DAS (SFPQ, SRPK1, SRSF11, SRSF2IP, FUS, LSM14A). In addition, a number of genes with predicted DAS are involved in pathways implicated in previous ASD studies, such as ROS monocyte/macrophage, Natural Killer Cell, mTOR, and NGF signaling. -

DHX29 Antibody A
Revision 1 C 0 2 - t DHX29 Antibody a e r o t S Orders: 877-616-CELL (2355) [email protected] Support: 877-678-TECH (8324) 9 5 Web: [email protected] 1 www.cellsignal.com 4 # 3 Trask Lane Danvers Massachusetts 01923 USA For Research Use Only. Not For Use In Diagnostic Procedures. Applications: Reactivity: Sensitivity: MW (kDa): Source: UniProt ID: Entrez-Gene Id: WB, IP H M R Endogenous 155 Rabbit Q7Z478 54505 Product Usage Information Application Dilution Western Blotting 1:1000 Immunoprecipitation 1:50 Storage Supplied in 10 mM sodium HEPES (pH 7.5), 150 mM NaCl, 100 µg/ml BSA and 50% glycerol. Store at –20°C. Do not aliquot the antibody. Specificity / Sensitivity DHX29 Antibody detects endogenous levels of total DHX29 protein. Species Reactivity: Human, Mouse, Rat Source / Purification Polyclonal antibodies are produced by immunizing animals with a synthetic peptide corresponding to residues at the C-terminus of human DHX29. Antibodies were purified by protein A and peptide affinity chromatography. Background DHX29 is an ATP-dependent RNA helicase that belongs to the DEAD-box helicase family (DEAH subfamily). DHX29 contains one central helicase and one helicase at the carboxy-terminal domain (1). Its function has not been fully established but DHX29 was recently shown to facilitate translation initiation on mRNAs with structured 5' untranslated regions (2). DHX29 binds 40S subunits and hydrolyzes ATP, GTP, UTP, and CTP. Hydrolysis of nucleotide triphosphates by DHX29 is strongly stimulated by 43S complexes and is required for DHX29 activity in promoting 48S complex formation (2). 1. de la Cruz, J. -

Plenary and Platform Abstracts
American Society of Human Genetics 68th Annual Meeting PLENARY AND PLATFORM ABSTRACTS Abstract #'s Tuesday, October 16, 5:30-6:50 pm: 4. Featured Plenary Abstract Session I Hall C #1-#4 Wednesday, October 17, 9:00-10:00 am, Concurrent Platform Session A: 6. Variant Insights from Large Population Datasets Ballroom 20A #5-#8 7. GWAS in Combined Cancer Phenotypes Ballroom 20BC #9-#12 8. Genome-wide Epigenomics and Non-coding Variants Ballroom 20D #13-#16 9. Clonal Mosaicism in Cancer, Alzheimer's Disease, and Healthy Room 6A #17-#20 Tissue 10. Genetics of Behavioral Traits and Diseases Room 6B #21-#24 11. New Frontiers in Computational Genomics Room 6C #25-#28 12. Bone and Muscle: Identifying Causal Genes Room 6D #29-#32 13. Precision Medicine Initiatives: Outcomes and Lessons Learned Room 6E #33-#36 14. Environmental Exposures in Human Traits Room 6F #37-#40 Wednesday, October 17, 4:15-5:45 pm, Concurrent Platform Session B: 24. Variant Interpretation Practices and Resources Ballroom 20A #41-#46 25. Integrated Variant Analysis in Cancer Genomics Ballroom 20BC #47-#52 26. Gene Discovery and Functional Models of Neurological Disorders Ballroom 20D #53-#58 27. Whole Exome and Whole Genome Associations Room 6A #59-#64 28. Sequencing-based Diagnostics for Newborns and Infants Room 6B #65-#70 29. Omics Studies in Alzheimer's Disease Room 6C #71-#76 30. Cardiac, Valvular, and Vascular Disorders Room 6D #77-#82 31. Natural Selection and Human Phenotypes Room 6E #83-#88 32. Genetics of Cardiometabolic Traits Room 6F #89-#94 Wednesday, October 17, 6:00-7:00 pm, Concurrent Platform Session C: 33. -

'Next- Generation' Sequencing Data Analysis
Novel Algorithm Development for ‘Next- Generation’ Sequencing Data Analysis Agne Antanaviciute Submitted in accordance with the requirements for the degree of Doctor of Philosophy University of Leeds School of Medicine Leeds Institute of Biomedical and Clinical Sciences 12/2017 ii The candidate confirms that the work submitted is her own, except where work which has formed part of jointly-authored publications has been included. The contribution of the candidate and the other authors to this work has been explicitly given within the thesis where reference has been made to the work of others. This copy has been supplied on the understanding that it is copyright material and that no quotation from the thesis may be published without proper acknowledgement ©2017 The University of Leeds and Agne Antanaviciute The right of Agne Antanaviciute to be identified as Author of this work has been asserted by her in accordance with the Copyright, Designs and Patents Act 1988. Acknowledgements I would like to thank all the people who have contributed to this work. First and foremost, my supervisors Dr Ian Carr, Professor David Bonthron and Dr Christopher Watson, who have provided guidance, support and motivation. I could not have asked for a better supervisory team. I would also like to thank my collaborators Dr Belinda Baquero and Professor Adrian Whitehouse for opening new, interesting research avenues. A special thanks to Dr Belinda Baquero for all the hard wet lab work without which at least half of this thesis would not exist. Thanks to everyone at the NGS Facility – Carolina Lascelles, Catherine Daley, Sally Harrison, Ummey Hany and Laura Crinnion – for the generation of NGS data used in this work and creating a supportive and stimulating work environment. -

Association of Gene Ontology Categories with Decay Rate for Hepg2 Experiments These Tables Show Details for All Gene Ontology Categories
Supplementary Table 1: Association of Gene Ontology Categories with Decay Rate for HepG2 Experiments These tables show details for all Gene Ontology categories. Inferences for manual classification scheme shown at the bottom. Those categories used in Figure 1A are highlighted in bold. Standard Deviations are shown in parentheses. P-values less than 1E-20 are indicated with a "0". Rate r (hour^-1) Half-life < 2hr. Decay % GO Number Category Name Probe Sets Group Non-Group Distribution p-value In-Group Non-Group Representation p-value GO:0006350 transcription 1523 0.221 (0.009) 0.127 (0.002) FASTER 0 13.1 (0.4) 4.5 (0.1) OVER 0 GO:0006351 transcription, DNA-dependent 1498 0.220 (0.009) 0.127 (0.002) FASTER 0 13.0 (0.4) 4.5 (0.1) OVER 0 GO:0006355 regulation of transcription, DNA-dependent 1163 0.230 (0.011) 0.128 (0.002) FASTER 5.00E-21 14.2 (0.5) 4.6 (0.1) OVER 0 GO:0006366 transcription from Pol II promoter 845 0.225 (0.012) 0.130 (0.002) FASTER 1.88E-14 13.0 (0.5) 4.8 (0.1) OVER 0 GO:0006139 nucleobase, nucleoside, nucleotide and nucleic acid metabolism3004 0.173 (0.006) 0.127 (0.002) FASTER 1.28E-12 8.4 (0.2) 4.5 (0.1) OVER 0 GO:0006357 regulation of transcription from Pol II promoter 487 0.231 (0.016) 0.132 (0.002) FASTER 6.05E-10 13.5 (0.6) 4.9 (0.1) OVER 0 GO:0008283 cell proliferation 625 0.189 (0.014) 0.132 (0.002) FASTER 1.95E-05 10.1 (0.6) 5.0 (0.1) OVER 1.50E-20 GO:0006513 monoubiquitination 36 0.305 (0.049) 0.134 (0.002) FASTER 2.69E-04 25.4 (4.4) 5.1 (0.1) OVER 2.04E-06 GO:0007050 cell cycle arrest 57 0.311 (0.054) 0.133 (0.002) -

Noelia Díaz Blanco
Effects of environmental factors on the gonadal transcriptome of European sea bass (Dicentrarchus labrax), juvenile growth and sex ratios Noelia Díaz Blanco Ph.D. thesis 2014 Submitted in partial fulfillment of the requirements for the Ph.D. degree from the Universitat Pompeu Fabra (UPF). This work has been carried out at the Group of Biology of Reproduction (GBR), at the Department of Renewable Marine Resources of the Institute of Marine Sciences (ICM-CSIC). Thesis supervisor: Dr. Francesc Piferrer Professor d’Investigació Institut de Ciències del Mar (ICM-CSIC) i ii A mis padres A Xavi iii iv Acknowledgements This thesis has been made possible by the support of many people who in one way or another, many times unknowingly, gave me the strength to overcome this "long and winding road". First of all, I would like to thank my supervisor, Dr. Francesc Piferrer, for his patience, guidance and wise advice throughout all this Ph.D. experience. But above all, for the trust he placed on me almost seven years ago when he offered me the opportunity to be part of his team. Thanks also for teaching me how to question always everything, for sharing with me your enthusiasm for science and for giving me the opportunity of learning from you by participating in many projects, collaborations and scientific meetings. I am also thankful to my colleagues (former and present Group of Biology of Reproduction members) for your support and encouragement throughout this journey. To the “exGBRs”, thanks for helping me with my first steps into this world. Working as an undergrad with you Dr. -

CHARACTERIZATION, EPIGENETIC DRUG EFFECT, and GENE DELIVERY to BREAST CANCER CELLS a Dissertation Presented to the Graduate Facu
CHARACTERIZATION, EPIGENETIC DRUG EFFECT, AND GENE DELIVERY TO BREAST CANCER CELLS A Dissertation Presented to The Graduate Faculty of The University of Akron In Partial Fulfillment of Requirements for the Degree Doctor of Philosophy Shan Lu December, 2015 CHARACTERIZATION, EPIGENETIC DRUG EFFECT, AND GENE DELIVERY TO BREAST CANCER CELLS Shan Lu Dissertation Approved: Accepted: Advisor Department Chair Dr. Vinod Labhasetwar Dr. Stephen Weeks Committee Chair Dean of the College Dr. Coleen Pugh Dr. John Green Committee Member Dean of Graduate School Dr. Abraham Joy Dr. Chand Midha Committee Member Dr. Ali Dhinojwala Committee Member Dr. Anand Ramamurthi Committee Member Dr. Peter Niewiarowski ii ABSTRACT Cancer relapse is strongly associated with the presence of cancer stem cells (CSCs), which drive the development of metastasis and drug resistance. In human breast cancer, CSCs are identified by the CD44+/CD24- phenotype and characterized by drug resistance, high tumorigenicity and metastatic potential. In this study, I found that MCF-7/Adr cells that are breast cancer cells resistant to doxorubicin (Dox) uniformly displayed CSC surface markers, possessed CSC proteins, formed in vitro mammospheres, yet retained low migratory rate. They were also able to self-renew and differentiate under floating culture condition and are responsive to epigenetic drug treatment. High degree of DNA methylation (modifications of the cytosine residues of DNA) and histone deacetylation are major epigenetic landmarks of CSCs. In this work, I showed that MCF-7/Adr cells are sensitive to histone deacetylation inhibitor suberoylanilide hydroxamic acid (SAHA). Through RNA-sequencing technology, I also found that decitabine (DAC) and SAHA similarly affected a large number of the examined pathways, including drug and nanoparticle cellular uptake and transport, lipid metabolism, carcinogenesis and nuclear transport pathways. -

Large-Scale Functional Prediction of Individual M6a RNA Methylation Sites from an RNA Co-Methylation Network
Wu et al. BMC Bioinformatics (2019) 20:223 https://doi.org/10.1186/s12859-019-2840-3 RESEARCHARTICLE Open Access m6Acomet: large-scale functional prediction of individual m6A RNA methylation sites from an RNA co- methylation network Xiangyu Wu1,2, Zhen Wei1,2, Kunqi Chen1,2, Qing Zhang1,4, Jionglong Su5, Hui Liu6, Lin Zhang6* and Jia Meng1,3* Abstract Background: Over one hundred different types of post-transcriptional RNA modifications have been identified in human. Researchers discovered that RNA modifications can regulate various biological processes, and RNA methylation, especially N6-methyladenosine, has become one of the most researched topics in epigenetics. Results: To date, the study of epitranscriptome layer gene regulation is mostly focused on the function of mediator proteins of RNA methylation, i.e., the readers, writers and erasers. There is limited investigation of the functional relevance of individual m6A RNA methylation site. To address this, we annotated human m6A sites in large-scale based on the guilt-by-association principle from an RNA co-methylation network. It is constructed based on public human MeRIP-Seq datasets profiling the m6A epitranscriptome under 32 independent experimental conditions. By systematically examining the network characteristics obtained from the RNA methylation profiles, a total of 339,158 putative gene ontology functions associated with 1446 human m6A sites were identified. These are biological functions that may be regulated at epitranscriptome layer via reversible m6A RNA methylation. The results were further validated on a soft benchmark by comparing to a random predictor. Conclusions: An online web server m6Acomet was constructed to support direct query for the predicted biological functions of m6A sites as well as the sites exhibiting co-methylated patterns at the epitranscriptome layer. -
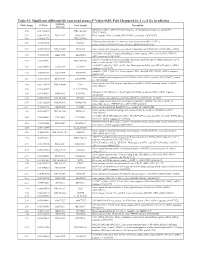
(P -Value<0.05, Fold Change≥1.4), 4 Vs. 0 Gy Irradiation
Table S1: Significant differentially expressed genes (P -Value<0.05, Fold Change≥1.4), 4 vs. 0 Gy irradiation Genbank Fold Change P -Value Gene Symbol Description Accession Q9F8M7_CARHY (Q9F8M7) DTDP-glucose 4,6-dehydratase (Fragment), partial (9%) 6.70 0.017399678 THC2699065 [THC2719287] 5.53 0.003379195 BC013657 BC013657 Homo sapiens cDNA clone IMAGE:4152983, partial cds. [BC013657] 5.10 0.024641735 THC2750781 Ciliary dynein heavy chain 5 (Axonemal beta dynein heavy chain 5) (HL1). 4.07 0.04353262 DNAH5 [Source:Uniprot/SWISSPROT;Acc:Q8TE73] [ENST00000382416] 3.81 0.002855909 NM_145263 SPATA18 Homo sapiens spermatogenesis associated 18 homolog (rat) (SPATA18), mRNA [NM_145263] AA418814 zw01a02.s1 Soares_NhHMPu_S1 Homo sapiens cDNA clone IMAGE:767978 3', 3.69 0.03203913 AA418814 AA418814 mRNA sequence [AA418814] AL356953 leucine-rich repeat-containing G protein-coupled receptor 6 {Homo sapiens} (exp=0; 3.63 0.0277936 THC2705989 wgp=1; cg=0), partial (4%) [THC2752981] AA484677 ne64a07.s1 NCI_CGAP_Alv1 Homo sapiens cDNA clone IMAGE:909012, mRNA 3.63 0.027098073 AA484677 AA484677 sequence [AA484677] oe06h09.s1 NCI_CGAP_Ov2 Homo sapiens cDNA clone IMAGE:1385153, mRNA sequence 3.48 0.04468495 AA837799 AA837799 [AA837799] Homo sapiens hypothetical protein LOC340109, mRNA (cDNA clone IMAGE:5578073), partial 3.27 0.031178378 BC039509 LOC643401 cds. [BC039509] Homo sapiens Fas (TNF receptor superfamily, member 6) (FAS), transcript variant 1, mRNA 3.24 0.022156298 NM_000043 FAS [NM_000043] 3.20 0.021043295 A_32_P125056 BF803942 CM2-CI0135-021100-477-g08 CI0135 Homo sapiens cDNA, mRNA sequence 3.04 0.043389246 BF803942 BF803942 [BF803942] 3.03 0.002430239 NM_015920 RPS27L Homo sapiens ribosomal protein S27-like (RPS27L), mRNA [NM_015920] Homo sapiens tumor necrosis factor receptor superfamily, member 10c, decoy without an 2.98 0.021202829 NM_003841 TNFRSF10C intracellular domain (TNFRSF10C), mRNA [NM_003841] 2.97 0.03243901 AB002384 C6orf32 Homo sapiens mRNA for KIAA0386 gene, partial cds.