Einfluss Von ADAM10, ADAM17, ADAM22 Und ADAM23 Auf Neurale Differenzierungsvorgänge
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Human ADAM12 Quantikine ELISA
Quantikine® ELISA Human ADAM12 Immunoassay Catalog Number DAD120 For the quantitative determination of A Disintegrin And Metalloproteinase domain- containing protein 12 (ADAM12) concentrations in cell culture supernates, serum, plasma, and urine. This package insert must be read in its entirety before using this product. For research use only. Not for use in diagnostic procedures. TABLE OF CONTENTS SECTION PAGE INTRODUCTION .....................................................................................................................................................................1 PRINCIPLE OF THE ASSAY ...................................................................................................................................................2 LIMITATIONS OF THE PROCEDURE .................................................................................................................................2 TECHNICAL HINTS .................................................................................................................................................................2 MATERIALS PROVIDED & STORAGE CONDITIONS ...................................................................................................3 OTHER SUPPLIES REQUIRED .............................................................................................................................................3 PRECAUTIONS .........................................................................................................................................................................4 -

Supplementary Table 1: Adhesion Genes Data Set
Supplementary Table 1: Adhesion genes data set PROBE Entrez Gene ID Celera Gene ID Gene_Symbol Gene_Name 160832 1 hCG201364.3 A1BG alpha-1-B glycoprotein 223658 1 hCG201364.3 A1BG alpha-1-B glycoprotein 212988 102 hCG40040.3 ADAM10 ADAM metallopeptidase domain 10 133411 4185 hCG28232.2 ADAM11 ADAM metallopeptidase domain 11 110695 8038 hCG40937.4 ADAM12 ADAM metallopeptidase domain 12 (meltrin alpha) 195222 8038 hCG40937.4 ADAM12 ADAM metallopeptidase domain 12 (meltrin alpha) 165344 8751 hCG20021.3 ADAM15 ADAM metallopeptidase domain 15 (metargidin) 189065 6868 null ADAM17 ADAM metallopeptidase domain 17 (tumor necrosis factor, alpha, converting enzyme) 108119 8728 hCG15398.4 ADAM19 ADAM metallopeptidase domain 19 (meltrin beta) 117763 8748 hCG20675.3 ADAM20 ADAM metallopeptidase domain 20 126448 8747 hCG1785634.2 ADAM21 ADAM metallopeptidase domain 21 208981 8747 hCG1785634.2|hCG2042897 ADAM21 ADAM metallopeptidase domain 21 180903 53616 hCG17212.4 ADAM22 ADAM metallopeptidase domain 22 177272 8745 hCG1811623.1 ADAM23 ADAM metallopeptidase domain 23 102384 10863 hCG1818505.1 ADAM28 ADAM metallopeptidase domain 28 119968 11086 hCG1786734.2 ADAM29 ADAM metallopeptidase domain 29 205542 11085 hCG1997196.1 ADAM30 ADAM metallopeptidase domain 30 148417 80332 hCG39255.4 ADAM33 ADAM metallopeptidase domain 33 140492 8756 hCG1789002.2 ADAM7 ADAM metallopeptidase domain 7 122603 101 hCG1816947.1 ADAM8 ADAM metallopeptidase domain 8 183965 8754 hCG1996391 ADAM9 ADAM metallopeptidase domain 9 (meltrin gamma) 129974 27299 hCG15447.3 ADAMDEC1 ADAM-like, -

Supplementary Table S4. FGA Co-Expressed Gene List in LUAD
Supplementary Table S4. FGA co-expressed gene list in LUAD tumors Symbol R Locus Description FGG 0.919 4q28 fibrinogen gamma chain FGL1 0.635 8p22 fibrinogen-like 1 SLC7A2 0.536 8p22 solute carrier family 7 (cationic amino acid transporter, y+ system), member 2 DUSP4 0.521 8p12-p11 dual specificity phosphatase 4 HAL 0.51 12q22-q24.1histidine ammonia-lyase PDE4D 0.499 5q12 phosphodiesterase 4D, cAMP-specific FURIN 0.497 15q26.1 furin (paired basic amino acid cleaving enzyme) CPS1 0.49 2q35 carbamoyl-phosphate synthase 1, mitochondrial TESC 0.478 12q24.22 tescalcin INHA 0.465 2q35 inhibin, alpha S100P 0.461 4p16 S100 calcium binding protein P VPS37A 0.447 8p22 vacuolar protein sorting 37 homolog A (S. cerevisiae) SLC16A14 0.447 2q36.3 solute carrier family 16, member 14 PPARGC1A 0.443 4p15.1 peroxisome proliferator-activated receptor gamma, coactivator 1 alpha SIK1 0.435 21q22.3 salt-inducible kinase 1 IRS2 0.434 13q34 insulin receptor substrate 2 RND1 0.433 12q12 Rho family GTPase 1 HGD 0.433 3q13.33 homogentisate 1,2-dioxygenase PTP4A1 0.432 6q12 protein tyrosine phosphatase type IVA, member 1 C8orf4 0.428 8p11.2 chromosome 8 open reading frame 4 DDC 0.427 7p12.2 dopa decarboxylase (aromatic L-amino acid decarboxylase) TACC2 0.427 10q26 transforming, acidic coiled-coil containing protein 2 MUC13 0.422 3q21.2 mucin 13, cell surface associated C5 0.412 9q33-q34 complement component 5 NR4A2 0.412 2q22-q23 nuclear receptor subfamily 4, group A, member 2 EYS 0.411 6q12 eyes shut homolog (Drosophila) GPX2 0.406 14q24.1 glutathione peroxidase -

Differential Gene Expression and Functional Analysis Implicate Novel Mechanisms in Enteric Nervous System Precursor Migration and Neuritogenesis
View metadata, citation and similar papers at core.ac.uk brought to you by CORE provided by Elsevier - Publisher Connector Developmental Biology 298 (2006) 259–271 www.elsevier.com/locate/ydbio Differential gene expression and functional analysis implicate novel mechanisms in enteric nervous system precursor migration and neuritogenesis Bhupinder P.S. Vohra a, Keiji Tsuji b,c, Mayumi Nagashimada b,d, Toshihiro Uesaka b, ⁎ ⁎ Daniel Wind a, Ming Fu a, Jennifer Armon a, Hideki Enomoto b, , Robert O. Heuckeroth a, a Department of Pediatrics and Department of Molecular Biology and Pharmacology, Washington University School of Medicine, 660 S. Euclid Avenue, Box 8208, St. Louis, MO 63110, USA b Laboratory for Neuronal Differentiation and Regeneration, RIKEN Center for Developmental Biology, 2-2-3 Minatojima-minamimachi, Chuo-ku, Kobe, Hyogo 650-0047, Japan c Department of Pediatrics, Kyoto Prefectural University of Medicine, Graduate School of Medical Science, Kamigyo-ku, Kyoto 602-8566, Japan d Osaka University Graduate School of Medicine, 2-2 Yamadaoka, Suita, Osaka 565-0871, Japan Received for publication 17 April 2006; revised 17 May 2006; accepted 22 June 2006 Available online 27 June 2006 Abstract Enteric nervous system (ENS) development requires complex interactions between migrating neural-crest-derived cells and the intestinal microenvironment. Although some molecules influencing ENS development are known, many aspects remain poorly understood. To identify additional molecules critical for ENS development, we used DNA microarray, quantitative real-time PCR and in situ hybridization to compare gene expression in E14 and P0 aganglionic or wild type mouse intestine. Eighty-three genes were identified with at least two-fold higher expression in wild type than aganglionic bowel. -
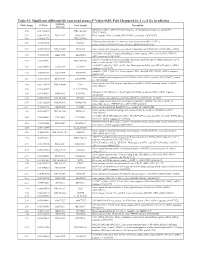
(P -Value<0.05, Fold Change≥1.4), 4 Vs. 0 Gy Irradiation
Table S1: Significant differentially expressed genes (P -Value<0.05, Fold Change≥1.4), 4 vs. 0 Gy irradiation Genbank Fold Change P -Value Gene Symbol Description Accession Q9F8M7_CARHY (Q9F8M7) DTDP-glucose 4,6-dehydratase (Fragment), partial (9%) 6.70 0.017399678 THC2699065 [THC2719287] 5.53 0.003379195 BC013657 BC013657 Homo sapiens cDNA clone IMAGE:4152983, partial cds. [BC013657] 5.10 0.024641735 THC2750781 Ciliary dynein heavy chain 5 (Axonemal beta dynein heavy chain 5) (HL1). 4.07 0.04353262 DNAH5 [Source:Uniprot/SWISSPROT;Acc:Q8TE73] [ENST00000382416] 3.81 0.002855909 NM_145263 SPATA18 Homo sapiens spermatogenesis associated 18 homolog (rat) (SPATA18), mRNA [NM_145263] AA418814 zw01a02.s1 Soares_NhHMPu_S1 Homo sapiens cDNA clone IMAGE:767978 3', 3.69 0.03203913 AA418814 AA418814 mRNA sequence [AA418814] AL356953 leucine-rich repeat-containing G protein-coupled receptor 6 {Homo sapiens} (exp=0; 3.63 0.0277936 THC2705989 wgp=1; cg=0), partial (4%) [THC2752981] AA484677 ne64a07.s1 NCI_CGAP_Alv1 Homo sapiens cDNA clone IMAGE:909012, mRNA 3.63 0.027098073 AA484677 AA484677 sequence [AA484677] oe06h09.s1 NCI_CGAP_Ov2 Homo sapiens cDNA clone IMAGE:1385153, mRNA sequence 3.48 0.04468495 AA837799 AA837799 [AA837799] Homo sapiens hypothetical protein LOC340109, mRNA (cDNA clone IMAGE:5578073), partial 3.27 0.031178378 BC039509 LOC643401 cds. [BC039509] Homo sapiens Fas (TNF receptor superfamily, member 6) (FAS), transcript variant 1, mRNA 3.24 0.022156298 NM_000043 FAS [NM_000043] 3.20 0.021043295 A_32_P125056 BF803942 CM2-CI0135-021100-477-g08 CI0135 Homo sapiens cDNA, mRNA sequence 3.04 0.043389246 BF803942 BF803942 [BF803942] 3.03 0.002430239 NM_015920 RPS27L Homo sapiens ribosomal protein S27-like (RPS27L), mRNA [NM_015920] Homo sapiens tumor necrosis factor receptor superfamily, member 10c, decoy without an 2.98 0.021202829 NM_003841 TNFRSF10C intracellular domain (TNFRSF10C), mRNA [NM_003841] 2.97 0.03243901 AB002384 C6orf32 Homo sapiens mRNA for KIAA0386 gene, partial cds. -

Comparative Transcriptome Analyses Reveal Genes Associated with SARS-Cov-2 Infection of Human Lung Epithelial Cells
bioRxiv preprint doi: https://doi.org/10.1101/2020.06.24.169268; this version posted June 24, 2020. The copyright holder for this preprint (which was not certified by peer review) is the author/funder. All rights reserved. No reuse allowed without permission. Comparative transcriptome analyses reveal genes associated with SARS-CoV-2 infection of human lung epithelial cells Darshan S. Chandrashekar1, *, Upender Manne1,2,#, Sooryanarayana Varambally1,2,3,#* 1Department of Pathology, University of Alabama at Birmingham, Birmingham, AL 2Comprehensive Cancer Center, University of Alabama at Birmingham, Birmingham, AL 3Institute of Informatics, University of Alabama at Birmingham, Birmingham, AL # Share Senior Authorship (UM Email: [email protected]) *Correspondence to: Sooryanarayana Varambally, Ph.D., Molecular and Cellular Pathology, Department of Pathology, Wallace Tumor Institute, 4th floor, 20B, University of Alabama at Birmingham, Birmingham, AL 35233, USA Phone: (205) 996-1654 Email: [email protected] And Darshan S. Chandrashekar Ph.D., Department of Pathology, University of Alabama at Birmingham, Birmingham, AL Email: [email protected] Running Title: SARS-CoV-2 gene signature in infected lung epithelial cells Disclosure of Potential Conflicts of Interest: No potential conflicts of interest were disclosed. Page | 1 bioRxiv preprint doi: https://doi.org/10.1101/2020.06.24.169268; this version posted June 24, 2020. The copyright holder for this preprint (which was not certified by peer review) is the author/funder. All rights reserved. No reuse allowed without permission. Abstract: Understanding the molecular mechanism of SARS-CoV-2 infection (the cause of COVID-19) is a scientific priority for 2020. Various research groups are working toward development of vaccines and drugs, and many have published genomic and transcriptomic data related to this viral infection. -
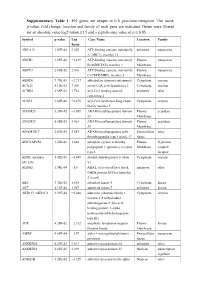
Supplementary Table 1
Supplementary Table 1. 492 genes are unique to 0 h post-heat timepoint. The name, p-value, fold change, location and family of each gene are indicated. Genes were filtered for an absolute value log2 ration 1.5 and a significance value of p ≤ 0.05. Symbol p-value Log Gene Name Location Family Ratio ABCA13 1.87E-02 3.292 ATP-binding cassette, sub-family unknown transporter A (ABC1), member 13 ABCB1 1.93E-02 −1.819 ATP-binding cassette, sub-family Plasma transporter B (MDR/TAP), member 1 Membrane ABCC3 2.83E-02 2.016 ATP-binding cassette, sub-family Plasma transporter C (CFTR/MRP), member 3 Membrane ABHD6 7.79E-03 −2.717 abhydrolase domain containing 6 Cytoplasm enzyme ACAT1 4.10E-02 3.009 acetyl-CoA acetyltransferase 1 Cytoplasm enzyme ACBD4 2.66E-03 1.722 acyl-CoA binding domain unknown other containing 4 ACSL5 1.86E-02 −2.876 acyl-CoA synthetase long-chain Cytoplasm enzyme family member 5 ADAM23 3.33E-02 −3.008 ADAM metallopeptidase domain Plasma peptidase 23 Membrane ADAM29 5.58E-03 3.463 ADAM metallopeptidase domain Plasma peptidase 29 Membrane ADAMTS17 2.67E-04 3.051 ADAM metallopeptidase with Extracellular other thrombospondin type 1 motif, 17 Space ADCYAP1R1 1.20E-02 1.848 adenylate cyclase activating Plasma G-protein polypeptide 1 (pituitary) receptor Membrane coupled type I receptor ADH6 (includes 4.02E-02 −1.845 alcohol dehydrogenase 6 (class Cytoplasm enzyme EG:130) V) AHSA2 1.54E-04 −1.6 AHA1, activator of heat shock unknown other 90kDa protein ATPase homolog 2 (yeast) AK5 3.32E-02 1.658 adenylate kinase 5 Cytoplasm kinase AK7 -

LGI1–ADAM22–MAGUK Configures Transsynaptic Nanoalignment for Synaptic Transmission and Epilepsy Prevention
LGI1–ADAM22–MAGUK configures transsynaptic nanoalignment for synaptic transmission and epilepsy prevention Yuko Fukataa,b,1, Xiumin Chenc,d,1, Satomi Chikenb,e, Yoko Hiranoa,f, Atsushi Yamagatag, Hiroki Inahashia, Makoto Sanboh, Hiromi Sanob,e, Teppei Gotoh, Masumi Hirabayashib,h, Hans-Christian Kornaui,j, Harald Prüssi,k, Atsushi Nambub,e, Shuya Fukail, Roger A. Nicollc,d,2, and Masaki Fukataa,b,2 aDivision of Membrane Physiology, Department of Molecular and Cellular Physiology, National Institute for Physiological Sciences, National Institutes of Natural Sciences, Aichi 444-8787, Japan; bDepartment of Physiological Sciences, School of Life Science, SOKENDAI (The Graduate University for Advanced Studies), Aichi 444-8585, Japan; cDepartment of Cellular and Molecular Pharmacology, University of California, San Francisco, CA 94158; dDepartment of Physiology, University of California, San Francisco, CA 94158; eDivision of System Neurophysiology, Department of System Neuroscience, National Institute for Physiological Sciences, National Institutes of Natural Sciences, Aichi 444-8585, Japan; fDepartment of Pediatrics, Graduate School of Medicine, The University of Tokyo, Tokyo 113-8655, Japan; gLaboratory for Protein Functional and Structural Biology, RIKEN Center for Biosystems Dynamics Research, Kanagawa 230-0045, Japan; hCenter for Genetic Analysis of Behavior, National Institute for Physiological Sciences, National Institutes of Natural Sciences, Okazaki 444-8787, Japan; iGerman Center for Neurodegenerative Diseases (DZNE), 10117 Berlin, Germany; jNeuroscience Research Center, Cluster NeuroCure, Charité-Universitätsmedizin Berlin, 10117 Berlin, Germany; kDepartment of Neurology and Experimental Neurology, Charité-Universitätsmedizin Berlin, 10117 Berlin, Germany; and lDepartment of Chemistry, Graduate School of Science, Kyoto University, 606-8502 Kyoto, Japan Contributed by Roger A. Nicoll, December 1, 2020 (sent for review October 29, 2020; reviewed by David S. -

Mutation Analysis of Candidate Genes Within the 2Q33.3 Linkage Area for Familial Early-Onset Generalised Osteoarthritis
European Journal of Human Genetics (2007) 15, 791–799 & 2007 Nature Publishing Group All rights reserved 1018-4813/07 $30.00 www.nature.com/ejhg ARTICLE Mutation analysis of candidate genes within the 2q33.3 linkage area for familial early-onset generalised osteoarthritis Josine L Min1, Ingrid Meulenbelt*,1, Margreet Kloppenburg2, Cornelia M van Duijn3 and P Eline Slagboom1 1Molecular Epidemiology, Leiden University Medical Centre, Leiden, The Netherlands; 2Department of Rheumatology, Leiden University Medical Centre, Leiden, The Netherlands; 3Department of Epidemiology & Biostatistics, Erasmus Medical Centre, Rotterdam, The Netherlands In a genome-wide linkage scan of seven families with familial early-onset osteoarthritis (FOA), we mapped a FOA locus to a 5 cM region on chromosome 2q33.3–2q34 with a maximum LOD score of 6.05. To identify causal variants, 17 positional candidate genes and FRZB were sequenced for coding, splice sites, and 50 and 30 untranslated regions. The pathogenicity of possible disease-causing variants was evaluated using predicted effects on protein structure and function, splicing enhancers, degree of conservation and frequency in 790 unrelated subjects from the population-based Rotterdam study scored for the presence of radiographic signs of OA (ROA). Nine novel variants, identified in NRP2, XM_371590, ADAM23, IDH1, PIP5K3 and PTHR2, cosegregated with FOA, of which two were promising. IDH1 Y183C cosegregated in one family, involved a conserved amino-acid change and showed a damaging effect predicted by PolyPhen and SIFT. In the Rotterdam sample, carriers of IDH1 Y183C (0.02) had an increased but insignificant risk for generalised ROA. The second variant, NRP2 c.1938-21T4C cosegregated in three families. -

Expression of ADAMTS13 and PCNA in the Placentas of Gestational Diabetic Mothers
Int. J. Morphol., 39(1):38-44, 2021. Expression of ADAMTS13 and PCNA in the Placentas of Gestational Diabetic Mothers Expresión de ADAMTS13 y PCNA en las Placentas de Madres Diabéticas Gestacionales Süleyman Cemil Oglak1 & Mehmet Obut1 OGLAK, S. C. & OBUT, M. Expression of ADAMTS13 and PCNA in the placentas of gestational diabetic mothers. Int. J. Morphol., 39(1):38-44, 2021. SUMMARY: GDM is linked with overexpression of inflammatory cytokines and increased oxidative stress, leading to endothelial dysfunction and vascular disorder. Weaimed to examine the expression of ADAMTS13 and PCNA in the placentas of gestational diabe- tes mellitus (GDM) patients to investigate the effects of hypoxia, induced by GDM, on proliferation and extracellular matrix formation in the maternal and fetal placenta cells. A total of 60 placentas were collected from pregnant women admitted to the obstetrics clinic. Thirty of them were diagnosed with GDM, and 30 of them were diagnosed with non-GDM patients. Samples were fixed in 10 % formaldehyde, after routine follow-up, embedded in paraffin wax. Sections of 5 µm were cut stained with Mayer Hematoxylin-Eosin, examined under a light microscope. Sections for immunohistochemical analysis were cut and processed for antigen retrievalin citrate solution. Sections were incubated with ADAMTS13 and PCNA primary antibodies, counterstained with hematoxylin, and evaluate under a light microscope. In histopathological examination, the non-diabetic placentas showed that decidua cells in the maternal region were polygonal with oval nuclei and organized in groups. In the GDM group, there were pyknosis and apoptotic changes in decidua cell nuclei. Vacuolar areas were observed in large cavities in maternal connective tissue. -

MMP-9 Affects Gene Expression in Chronic Lymphocytic Leukemia
1 MMP-9 affects gene expression in chronic lymphocytic leukemia 2 revealing CD99 as an MMP-9 target and a novel partner in malignant cell 3 migration/arrest 4 Running title: Role of the MMP-9 target CD99 in CLL migration * * 5 Noemí Aguilera-Montilla1 , Elvira Bailón1 , Rebeca Uceda-Castro1, Estefanía 6 Ugarte-Berzal2, Andrea Santos1, Alejandra Gutiérrez-González1, Cristina 7 Pérez-Sánchez1, Philippe E. Van den Steen2, Ghislain Opdenakker2, José A. 8 García-Marco3 and Angeles García-Pardo1# 9 1Dept. of Molecular Biomedicine, Centro de Investigaciones Biológicas, 10 Consejo Superior de Investigaciones Científicas (CSIC), Madrid, Spain; 11 2Dept. of Microbiology and Immunology, Rega Institute for Medical Research, 12 University of Leuven, KU Leuven, Belgium; 3Dept. of Hematology, Hospital * 13 Universitario Puerta de Hierro, Madrid, Spain. These authors contributed 14 equally to this work. The authors declare no competing financial interests. 15 16 #Correspondence: Dr. Angeles García-Pardo, Dept. of Molecular Biomedicine, 17 Centro de Investigaciones Biológicas, CSIC. Ramiro de Maeztu 9, 28040 18 Madrid, Spain. Tel: 34-91-8373112ext 4430; e-mail: 19 [email protected] 20 Funding: Grants SAF2015-69180R and RD12/0036/0061 from the Ministry of 21 Economy and Competitivity (Spain), and S2010/BMD-2314 from the 22 Comunidad de Madrid/European Union (to AGP); Concerted Research 23 Actions C1 from KU Leuven (C16/17/010), and the Research Foundation of 24 Flander (FWO- Vlaanderen) (to EUB, PVdS and GO). 1 25 Abstract 26 We previously showed that MMP-9 contributes to CLL pathology by regulating 27 cell survival and migration and that, when present at high levels, MMP-9 28 induces cell arrest. -

ADAM23 Is Downregulated in Side Population and Suppresses Lung Metastasis of Lung Carcinoma Cells
ADAM23 is downregulated in side population and suppresses lung metastasis of lung carcinoma cells Masahide Ota,1,2 Satsuki Mochizuki,1 Masayuki Shimoda,1 Hitoshi Abe,1 Yuka Miyamae,1 Ken Ishii,3 Hiroshi Kimura2 and Yasunori Okada1,4 1Department of Pathology, Keio University School of Medicine, Tokyo; 2Second Department of Internal Medicine and Respiratory Medicine, Nara Medical University, Kashihara; 3Department of Orthopedic Surgery, Keio University School of Medicine, Tokyo; 4Department of Pathophysiology for Locomotive and Neoplastic Diseases, Juntendo University Graduate School of Medicine, Tokyo, Japan Key words Cancer cells contain a small population of cancer stem cells or cancer initiating ADAM23, colony formation, metastasis, non-small cell cells, which can be enriched in the side population (SP) after fluorescence acti- lung carcinoma cells, side population vated cell sorting. To examine the members of the ADAM, ADAMTS and MMP Correspondence gene families related to phenotypes of the SP and the main population (MP), we Yasunori Okada, Department of Pathophysiology for screened the expression of all the members in the propagated SP and MP of Locomotive and Neoplastic Diseases, Juntendo University A549 lung adenocarcinoma cells, and found that the relative expression ratio of Graduate School of Medicine, 2-1-1 Hongo, Bunkyo-ku, ADAM23 in the MP to the SP is most highly increased, but none of them are Tokyo 113-8421, Japan. increased in the SP. A similar result on the ADAM23 expression was obtained Tel: +81-3-5800-7531; Fax: +81-3-5800-7532; E-mail: [email protected] with another cell line, Calu-3 cells. Overexpression of ADAM23 inhibited colony formation, cell adhesion and migration, and knockdown of ADAM23 by shRNA Funding Information showed the reverse effects.