Deciphering Antibody Fingerprints with High Density Peptide Arrays: from Methodological Development Toward Disease Etiology
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Na+/K+-Atpase) and Its Isoforms Kaur R1,2, Sodhi M2, Sharma VL1, Sharma A2, Mann S2 and Mukesh M 2
TRJ VOL. 2 ISSUE 2 MAR-APR 2016 ISSN: 2454-7301 (PRINT) | ISSN: 2454-4930 (ONLINE) A Review: Sodium-Potassium Adenosine Triphosphatase (Na+/K+-ATPase) and its Isoforms Kaur R1,2, Sodhi M2, Sharma VL1, Sharma A2, Mann S2 and Mukesh M 2. 1Department of Zoology, Panjab University, Chandigarh 2National Bureau of Animal Genetic Resources, Karnal *Email: [email protected] these enzymes use the hydrolysis of ATP to drive the transport Abstract - Na+/K+-ATPase, found in all mammalian cell of cations against an electrochemical potential. membranes, is necessary for proper cellular function since it The characteristic feature of P type ATPase is the helps to preserve the ionic gradients across the cell membrane formation of the phosphorylated intermediate state during the and thus the membrane potential and osmotic equilibrium of reaction cycle [5]. Hence this features differentiates it from the cell. The sodium-potassium ATPase (Na+/K+-ATPase) is other types of ATPases i.e. F-ATPases (present in an important ion transporter which pumps sodium out of cells mitochondria, chloroplasts and bacterial plasma membranes) in exchange for potassium, thereby generating large gradients and V-ATPases (present in eukaryotic vacuoles). The presence of these ions across the plasma membrane. The isoforms α (αl, of conserved sequence motifs in the cytoplasmic domains α2, α3 and α4) and β (β1, β2 and β3) combine to form a defines the primary structure of P-type ATPases. Nucleotide- number of Na+/K+-ATPase isozymes. So in present study binding (N), phosphorylation (P) and an actuator (A) domain paper information regarding molecular regulation of the are the three domains present in the cytoplasmic domain [6]. -

1/05661 1 Al
(12) INTERNATIONAL APPLICATION PUBLISHED UNDER THE PATENT COOPERATION TREATY (PCT) (19) World Intellectual Property Organization International Bureau (10) International Publication Number (43) International Publication Date _ . ... - 12 May 2011 (12.05.2011) W 2 11/05661 1 Al (51) International Patent Classification: (81) Designated States (unless otherwise indicated, for every C12Q 1/00 (2006.0 1) C12Q 1/48 (2006.0 1) kind of national protection available): AE, AG, AL, AM, C12Q 1/42 (2006.01) AO, AT, AU, AZ, BA, BB, BG, BH, BR, BW, BY, BZ, CA, CH, CL, CN, CO, CR, CU, CZ, DE, DK, DM, DO, (21) Number: International Application DZ, EC, EE, EG, ES, FI, GB, GD, GE, GH, GM, GT, PCT/US20 10/054171 HN, HR, HU, ID, IL, IN, IS, JP, KE, KG, KM, KN, KP, (22) International Filing Date: KR, KZ, LA, LC, LK, LR, LS, LT, LU, LY, MA, MD, 26 October 2010 (26.10.2010) ME, MG, MK, MN, MW, MX, MY, MZ, NA, NG, NI, NO, NZ, OM, PE, PG, PH, PL, PT, RO, RS, RU, SC, SD, (25) Filing Language: English SE, SG, SK, SL, SM, ST, SV, SY, TH, TJ, TM, TN, TR, (26) Publication Language: English TT, TZ, UA, UG, US, UZ, VC, VN, ZA, ZM, ZW. (30) Priority Data: (84) Designated States (unless otherwise indicated, for every 61/255,068 26 October 2009 (26.10.2009) US kind of regional protection available): ARIPO (BW, GH, GM, KE, LR, LS, MW, MZ, NA, SD, SL, SZ, TZ, UG, (71) Applicant (for all designated States except US): ZM, ZW), Eurasian (AM, AZ, BY, KG, KZ, MD, RU, TJ, MYREXIS, INC. -

Fhit Proteins Can Also Recognize Substrates Other Than Dinucleoside Polyphosphates
FEBS Letters 582 (2008) 3152–3158 Fhit proteins can also recognize substrates other than dinucleoside polyphosphates Andrzej Guranowskia,*, Anna M. Wojdyłaa, Małgorzata Pietrowska-Borekb, Paweł Bieganowskic, Elena N. Khursd, Matthew J. Cliffe, G. Michael Blackburnf, Damian Błaziakg, Wojciech J. Stecg a Department of Biochemistry and Biotechnology, The University of Life Sciences, 35 Wołyn´ska Street, 60-637 Poznan´, Poland b Department of Plant Physiology, The University of Life Sciences, 60-637 Poznan´, Poland c International Institute of Molecular and Cell Biology, Warsaw, Poland d Engelhardt Institute of Molecular Biology, Russian Academy of Sciences, Moscow, Russia e Department of Molecular Biology and Biotechnology, Krebs Institute, University of Sheffield, Sheffield, UK f Department of Chemistry, Krebs Institute, University of Sheffield, Sheffield, UK g Center of Molecular and Macromolecular Studies, Polish Academy of Sciences, Ło´dz´, Poland Received 1 July 2008; revised 17 July 2008; accepted 31 July 2008 Available online 9 August 2008 Edited by Hans Eklund This work is dedicated to Professor Antonio Sillero on the occasion of his 70th birthday and to Professor Maria Antonia Gu¨nther Sillero. 1. Introduction Abstract We show here that Fhit proteins, in addition to their function as dinucleoside triphosphate hydrolases, act similarly to adenylylsulfatases and nucleoside phosphoramidases, liberat- Cells contain various minor nucleotides. Among these are 0 000 1 n 0 ing nucleoside 50-monophosphates from such natural metabolites the dinucleoside 5 ,5 -P ,P -polyphosphates (NpnN s, where as adenosine 50-phosphosulfate and adenosine 50-phosphorami- N and N0 are 50-O-nucleosides and n represents the number date. Moreover, Fhits recognize synthetic nucleotides, such as of phosphate residues in the polyphosphate chain that esterifies 0 0 0 0 0 adenosine 5 -O-phosphorofluoridate and adenosine 5 -O-(c-flu- N and N at their 5 position) [1].NpnN s accumulate as a re- orotriphosphate), and release AMP from them. -
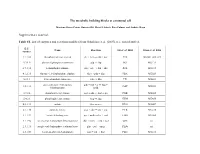
The Metabolic Building Blocks of a Minimal Cell Supplementary
The metabolic building blocks of a minimal cell Mariana Reyes-Prieto, Rosario Gil, Mercè Llabrés, Pere Palmer and Andrés Moya Supplementary material. Table S1. List of enzymes and reactions modified from Gabaldon et. al. (2007). n.i.: non identified. E.C. Name Reaction Gil et. al. 2004 Glass et. al. 2006 number 2.7.1.69 phosphotransferase system glc + pep → g6p + pyr PTS MG041, 069, 429 5.3.1.9 glucose-6-phosphate isomerase g6p ↔ f6p PGI MG111 2.7.1.11 6-phosphofructokinase f6p + atp → fbp + adp PFK MG215 4.1.2.13 fructose-1,6-bisphosphate aldolase fbp ↔ gdp + dhp FBA MG023 5.3.1.1 triose-phosphate isomerase gdp ↔ dhp TPI MG431 glyceraldehyde-3-phosphate gdp + nad + p ↔ bpg + 1.2.1.12 GAP MG301 dehydrogenase nadh 2.7.2.3 phosphoglycerate kinase bpg + adp ↔ 3pg + atp PGK MG300 5.4.2.1 phosphoglycerate mutase 3pg ↔ 2pg GPM MG430 4.2.1.11 enolase 2pg ↔ pep ENO MG407 2.7.1.40 pyruvate kinase pep + adp → pyr + atp PYK MG216 1.1.1.27 lactate dehydrogenase pyr + nadh ↔ lac + nad LDH MG460 1.1.1.94 sn-glycerol-3-phosphate dehydrogenase dhp + nadh → g3p + nad GPS n.i. 2.3.1.15 sn-glycerol-3-phosphate acyltransferase g3p + pal → mag PLSb n.i. 2.3.1.51 1-acyl-sn-glycerol-3-phosphate mag + pal → dag PLSc MG212 acyltransferase 2.7.7.41 phosphatidate cytidyltransferase dag + ctp → cdp-dag + pp CDS MG437 cdp-dag + ser → pser + 2.7.8.8 phosphatidylserine synthase PSS n.i. cmp 4.1.1.65 phosphatidylserine decarboxylase pser → peta PSD n.i. -

Leigh's Disease: Significance of the Biochemical Changes in Brain'
Journal ofNeurology, Neurosurgery, and Psychiatry, 1975, 38, 1100-1103 J Neurol Neurosurg Psychiatry: first published as 10.1136/jnnp.38.11.1100 on 1 November 1975. Downloaded from Leigh's disease: significance of the biochemical changes in brain' JEROME V. MURPHY2 AND LINDA CRAIG From the Departments ofPediatrics and Neurology of the University ofPittsburgh School of Medicine and the Children's Hospital ofPittsburgh, Pittsburgh, Pennsylvania 15213, U.S.A. SYNOPSIS Analysis of five brains from patients with Leigh's disease demonstrates an accumulation of thiamine pyrophosphate and a deficiency of thiamine triphosphate. The enzyme which converts thiamine pyrophosphate to thiamine triphosphate was normally active in two of these brains, suggesting that the inhibitor found in Leigh's disease is probably producing the observed neuro- chemical changes. Reasons for the histological similarity between Leigh's and Wernicke's diseases are suggested. Leigh's disease, subacute necrotizing encephalo- detected in body fluids of untreated patients with guest. Protected by copyright. myelopathy, is a recessively inherited disorder Leigh's disease as well as in the urine of obligate afflicting children of all ages (Pincus, 1972). The carriers of the Leigh's disease gene (Murphy clinical findings are nonspecific and the diagnosis et al., 1974). Thiamine appears to inactivate the is made byfindingthe typical pathological changes inhibitor, as treatment of either patients or in the brain of the patient or the brain of a carriers with large amounts of thiamine effec- similarly afflicted sibling (Montpetit et al., 1971). tively reduces the activity of inhibitor in urine Two patients with Leigh's disease have had a (Murphy, 1973; Murphy et al., 1974). -

An Alternative Role of Fof1-ATP Synthase in Escherichia Coli
An alternative role of FoF1-ATP synthase in Escherichia coli: synthesis of thiamine SUBJECT AREAS: BIOCHEMISTRY triphosphate ENZYMES Tiziana Gigliobianco1, Marjorie Gangolf1, Bernard Lakaye1, Bastien Pirson1, Christoph von Ballmoos2, CELL BIOLOGY Pierre Wins1 & Lucien Bettendorff1 CELLULAR MICROBIOLOGY 1Unit of Bioenergetics and cerebral Excitability, GIGA-Neurosciences, University of Lie`ge, B-4000 Lie`ge, Belgium, 2Department of Received Biochemistry and Biophysics, Arrhenius Laboratories for Natural Sciences, Stockholm University, SE-106 91 Stockholm, Sweden. 23 November 2012 Accepted In E. coli, thiamine triphosphate (ThTP), a putative signaling molecule, transiently accumulates in response 21 December 2012 to amino acid starvation. This accumulation requires the presence of an energy substrate yielding pyruvate. Published Here we show that in intact bacteria ThTP is synthesized from free thiamine diphosphate (ThDP) and Pi, the reaction being energized by the proton-motive force (Dp) generated by the respiratory chain. ThTP 15 January 2013 production is suppressed in strains carrying mutations in F1 or a deletion of the atp operon. Transformation with a plasmid encoding the whole atp operon fully restored ThTP production, highlighting the requirement for FoF1-ATP synthase in ThTP synthesis. Our results show that, under specific conditions of Correspondence and nutritional downshift, FoF1-ATP synthase catalyzes the synthesis of ThTP, rather than ATP, through a requests for materials highly regulated process requiring pyruvate oxidation. Moreover, this chemiosmotic mechanism for ThTP production is conserved from E. coli to mammalian brain mitochondria. should be addressed to L.B. (L.Bettendorff@ulg. ac.be) hiamine (vitamin B1) is an essential compound for all known life forms. In most organisms, the well-known cofactor thiamine diphosphate (ThDP) is the major thiamine compound. -

Ca2+-Atpase Molecules As a Calcium-Sensitive Membrane-Endoskeleton of Sarcoplasmic Reticulum
International Journal of Molecular Sciences Article Ca2+-ATPase Molecules as a Calcium-Sensitive Membrane-Endoskeleton of Sarcoplasmic Reticulum Jun Nakamura 1,*, Yuusuke Maruyama 1, Genichi Tajima 2, Yuto Komeiji 1, Makiko Suwa 3 and Chikara Sato 1,* 1 Health and Medical Research Institute, National Institute of Advanced Industrial Science and Technology (AIST), Central 6, 1-1-1 Higashi, Tsukuba, Ibaraki 305-8566, Japan; [email protected] (Y.M.); [email protected] (Y.K.) 2 Institute for Excellence in Higher Education, Tohoku University, 41 Kawauchi, Aoba-ku, Sendai, Miyagi 980-8576, Japan; [email protected] 3 Biological Science Course, Graduate School of Science and Engineering, Aoyama Gakuin University, 5-10-1 Fuchinobe, Chuou-ku, Sagamihara, Kanagawa 252-5258, Japan; [email protected] * Correspondence: [email protected] (J.N.); [email protected] (C.S.) Abstract: The Ca2+-transport ATPase of sarcoplasmic reticulum (SR) is an integral, transmembrane protein. It sequesters cytoplasmic calcium ions released from SR during muscle contraction, and causes muscle relaxation. Based on negative staining and transmission electron microscopy of SR vesicles isolated from rabbit skeletal muscle, we propose that the ATPase molecules might also be a calcium-sensitive membrane-endoskeleton. Under conditions when the ATPase molecules scarcely transport Ca2+, i.e., in the presence of ATP and ≤ 0.9 nM Ca2+, some of the ATPase particles on the SR vesicle surface gathered to form tetramers. The tetramers crystallized into a cylindrical helical array in some vesicles and probably resulted in the elongated protrusion that extended from some round SRs. -

Small Gtpases of the Ras and Rho Families Switch On/Off Signaling
International Journal of Molecular Sciences Review Small GTPases of the Ras and Rho Families Switch on/off Signaling Pathways in Neurodegenerative Diseases Alazne Arrazola Sastre 1,2, Miriam Luque Montoro 1, Patricia Gálvez-Martín 3,4 , Hadriano M Lacerda 5, Alejandro Lucia 6,7, Francisco Llavero 1,6,* and José Luis Zugaza 1,2,8,* 1 Achucarro Basque Center for Neuroscience, Science Park of the Universidad del País Vasco/Euskal Herriko Unibertsitatea (UPV/EHU), 48940 Leioa, Spain; [email protected] (A.A.S.); [email protected] (M.L.M.) 2 Department of Genetics, Physical Anthropology, and Animal Physiology, Faculty of Science and Technology, UPV/EHU, 48940 Leioa, Spain 3 Department of Pharmacy and Pharmaceutical Technology, Faculty of Pharmacy, University of Granada, 180041 Granada, Spain; [email protected] 4 R&D Human Health, Bioibérica S.A.U., 08950 Barcelona, Spain 5 Three R Labs, Science Park of the UPV/EHU, 48940 Leioa, Spain; [email protected] 6 Faculty of Sport Science, European University of Madrid, 28670 Madrid, Spain; [email protected] 7 Research Institute of the Hospital 12 de Octubre (i+12), 28041 Madrid, Spain 8 IKERBASQUE, Basque Foundation for Science, 48013 Bilbao, Spain * Correspondence: [email protected] (F.L.); [email protected] (J.L.Z.) Received: 25 July 2020; Accepted: 29 August 2020; Published: 31 August 2020 Abstract: Small guanosine triphosphatases (GTPases) of the Ras superfamily are key regulators of many key cellular events such as proliferation, differentiation, cell cycle regulation, migration, or apoptosis. To control these biological responses, GTPases activity is regulated by guanine nucleotide exchange factors (GEFs), GTPase activating proteins (GAPs), and in some small GTPases also guanine nucleotide dissociation inhibitors (GDIs). -

G Protein Coupled Receptor Signaling Through the Src and Stat3 Pathway: Role in Proliferation and Transformation
Oncogene (2001) 20, 1601 ± 1606 ã 2001 Nature Publishing Group All rights reserved 0950 ± 9232/01 $15.00 www.nature.com/onc G protein coupled receptor signaling through the Src and Stat3 pathway: role in proliferation and transformation Prahlad T Ram*,1 and Ravi Iyengar1 1Department of Pharmacology, Mount Sinai School of Medicine, New York, NY 10029, USA Extracellular signals when routed through signaling transducin; the Gai family which includes Gai, Gao pathways that use heterotrimeric G proteins can and Gaz; the Gaq/Ga11 family; and the Ga12/13 engage multiple signaling pathways leading to diverse family. As these new Ga subunits were identi®ed the biological consequences. One locus at which signal eectors pathways for these subunits were sought out, sorting occurs is at the level of G proteins. G protein and over the years a number of eector pathways such a-subunits appear to be capable of interacting with as the PLC, Rho/Cdc42 pathways, and the Ca2+ dierent eectors leading to engagement of distinct channels have been identi®ed (Buhl et al., 1995; signaling pathways. Regulation of dierent pathways Diverse-Pierluissi, 1995; rench-Mullen et al., 1994; in turn leads to dierent biological outcomes. The Taylor et al., 1990). In addition to activation of process of neoplastic transformation is controlled to a second messenger molecules, Ga subunits can also large extent through the activation and inhibition of modulate the activity of transcription factors, thereby signaling pathways. Signaling pathways such as the regulating gene expression (Corre et al., 1999; Fan et Ras-MAPK, v-Src-Stat3 pathways are activated in the al., 2000; Montminy, 1997; Ram et al., 2000). -

12) United States Patent (10
US007635572B2 (12) UnitedO States Patent (10) Patent No.: US 7,635,572 B2 Zhou et al. (45) Date of Patent: Dec. 22, 2009 (54) METHODS FOR CONDUCTING ASSAYS FOR 5,506,121 A 4/1996 Skerra et al. ENZYME ACTIVITY ON PROTEIN 5,510,270 A 4/1996 Fodor et al. MICROARRAYS 5,512,492 A 4/1996 Herron et al. 5,516,635 A 5/1996 Ekins et al. (75) Inventors: Fang X. Zhou, New Haven, CT (US); 5,532,128 A 7/1996 Eggers Barry Schweitzer, Cheshire, CT (US) 5,538,897 A 7/1996 Yates, III et al. s s 5,541,070 A 7/1996 Kauvar (73) Assignee: Life Technologies Corporation, .. S.E. al Carlsbad, CA (US) 5,585,069 A 12/1996 Zanzucchi et al. 5,585,639 A 12/1996 Dorsel et al. (*) Notice: Subject to any disclaimer, the term of this 5,593,838 A 1/1997 Zanzucchi et al. patent is extended or adjusted under 35 5,605,662 A 2f1997 Heller et al. U.S.C. 154(b) by 0 days. 5,620,850 A 4/1997 Bamdad et al. 5,624,711 A 4/1997 Sundberg et al. (21) Appl. No.: 10/865,431 5,627,369 A 5/1997 Vestal et al. 5,629,213 A 5/1997 Kornguth et al. (22) Filed: Jun. 9, 2004 (Continued) (65) Prior Publication Data FOREIGN PATENT DOCUMENTS US 2005/O118665 A1 Jun. 2, 2005 EP 596421 10, 1993 EP 0619321 12/1994 (51) Int. Cl. EP O664452 7, 1995 CI2O 1/50 (2006.01) EP O818467 1, 1998 (52) U.S. -

The RHO Family Gtpases: Mechanisms of Regulation and Signaling
cells Review The RHO Family GTPases: Mechanisms of Regulation and Signaling Niloufar Mosaddeghzadeh and Mohammad Reza Ahmadian * Institute of Biochemistry and Molecular Biology II, Medical Faculty of the Heinrich Heine University, Universitätsstrasse 1, Building 22.03.05, 40225 Düsseldorf, Germany; [email protected] * Correspondence: [email protected] Abstract: Much progress has been made toward deciphering RHO GTPase functions, and many studies have convincingly demonstrated that altered signal transduction through RHO GTPases is a recurring theme in the progression of human malignancies. It seems that 20 canonical RHO GTPases are likely regulated by three GDIs, 85 GEFs, and 66 GAPs, and eventually interact with >70 downstream effectors. A recurring theme is the challenge in understanding the molecular determinants of the specificity of these four classes of interacting proteins that, irrespective of their functions, bind to common sites on the surface of RHO GTPases. Identified and structurally verified hotspots as functional determinants specific to RHO GTPase regulation by GDIs, GEFs, and GAPs as well as signaling through effectors are presented, and challenges and future perspectives are discussed. Keywords: CDC42; effectors; RAC1; RHOA; RHOGAP; RHOGDI; RHOGEF; RHO signaling 1. Introduction Citation: Mosaddeghzadeh, N.; The RHO (RAS homolog) family is an integral part of the RAS superfamily of guanine Ahmadian, M.R. The RHO Family nucleotide-binding proteins. RHO family proteins are crucial for several reasons: (i) ap- GTPases: Mechanisms of Regulation proximately 1% of the human genome encodes proteins that either regulate or are regulated and Signaling. Cells 2021, 10, 1831. by direct interaction with RHO proteins; (ii) they control almost all fundamental cellular https://doi.org/10.3390/cells10071831 processes in eukaryotes including morphogenesis, polarity, movement, cell division, gene expression, and cytoskeleton reorganization [1]; and (iii) they are associated with a series Academic Editor: Bor Luen Tang of human diseases (Figure1)[2]. -

Activation and Inhibition of Gtpase Translation Factors on the Prokaryotic Ribosome
Western Washington University Western CEDAR WWU Graduate School Collection WWU Graduate and Undergraduate Scholarship 2010 Activation and inhibition of GTPase translation factors on the prokaryotic ribosome Justin D. Walter Western Washington University Follow this and additional works at: https://cedar.wwu.edu/wwuet Part of the Chemistry Commons Recommended Citation Walter, Justin D., "Activation and inhibition of GTPase translation factors on the prokaryotic ribosome" (2010). WWU Graduate School Collection. 96. https://cedar.wwu.edu/wwuet/96 This Masters Thesis is brought to you for free and open access by the WWU Graduate and Undergraduate Scholarship at Western CEDAR. It has been accepted for inclusion in WWU Graduate School Collection by an authorized administrator of Western CEDAR. For more information, please contact [email protected]. Activation and Inhibition of GTPase Translation Factors on the Prokaryotic Ribosome By Justin D. Walter Accepted in Partial Completion Of the Requirements for the Degree Master of Science Moheb A. Ghali, Dean of the Graduate School ADVISORY COMMITTEE Chair, Dr. P. Clint Spiegel Dr. Spencer Anthony-Cahill Dr. Gerry Prody MASTER’S THESIS In presenting this thesis in partial fulfillment of the requirements for a master’s degree at Western Washington University, I grant to Western Washington University the non‐exclusive royalty‐free right to archive, reproduce, distribute, and display the thesis in any and all forms, including electronic format, via any digital library mechanisms maintained by WWU. I represent and warrant this is my original work, and does not infringe or violate any rights of others. I warrant that I have obtained written permissions from the owner of any third party copyrighted material included in these files.