Analysis of Molecular Events Involved in Chondrogenesis and Somitogenesis by Global Gene Expression Profiling
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Gene Symbol Gene Description ACVR1B Activin a Receptor, Type IB
Table S1. Kinase clones included in human kinase cDNA library for yeast two-hybrid screening Gene Symbol Gene Description ACVR1B activin A receptor, type IB ADCK2 aarF domain containing kinase 2 ADCK4 aarF domain containing kinase 4 AGK multiple substrate lipid kinase;MULK AK1 adenylate kinase 1 AK3 adenylate kinase 3 like 1 AK3L1 adenylate kinase 3 ALDH18A1 aldehyde dehydrogenase 18 family, member A1;ALDH18A1 ALK anaplastic lymphoma kinase (Ki-1) ALPK1 alpha-kinase 1 ALPK2 alpha-kinase 2 AMHR2 anti-Mullerian hormone receptor, type II ARAF v-raf murine sarcoma 3611 viral oncogene homolog 1 ARSG arylsulfatase G;ARSG AURKB aurora kinase B AURKC aurora kinase C BCKDK branched chain alpha-ketoacid dehydrogenase kinase BMPR1A bone morphogenetic protein receptor, type IA BMPR2 bone morphogenetic protein receptor, type II (serine/threonine kinase) BRAF v-raf murine sarcoma viral oncogene homolog B1 BRD3 bromodomain containing 3 BRD4 bromodomain containing 4 BTK Bruton agammaglobulinemia tyrosine kinase BUB1 BUB1 budding uninhibited by benzimidazoles 1 homolog (yeast) BUB1B BUB1 budding uninhibited by benzimidazoles 1 homolog beta (yeast) C9orf98 chromosome 9 open reading frame 98;C9orf98 CABC1 chaperone, ABC1 activity of bc1 complex like (S. pombe) CALM1 calmodulin 1 (phosphorylase kinase, delta) CALM2 calmodulin 2 (phosphorylase kinase, delta) CALM3 calmodulin 3 (phosphorylase kinase, delta) CAMK1 calcium/calmodulin-dependent protein kinase I CAMK2A calcium/calmodulin-dependent protein kinase (CaM kinase) II alpha CAMK2B calcium/calmodulin-dependent -

Bioinformatic Analyses of Integral Membrane Transport Proteins Encoded Within the Genome of the Planctomycetes Species, Rhodopirellula Baltica
UC San Diego UC San Diego Previously Published Works Title Bioinformatic analyses of integral membrane transport proteins encoded within the genome of the planctomycetes species, Rhodopirellula baltica. Permalink https://escholarship.org/uc/item/0f85q1z7 Journal Biochimica et biophysica acta, 1838(1 Pt B) ISSN 0006-3002 Authors Paparoditis, Philipp Västermark, Ake Le, Andrew J et al. Publication Date 2014 DOI 10.1016/j.bbamem.2013.08.007 Peer reviewed eScholarship.org Powered by the California Digital Library University of California Biochimica et Biophysica Acta 1838 (2014) 193–215 Contents lists available at ScienceDirect Biochimica et Biophysica Acta journal homepage: www.elsevier.com/locate/bbamem Bioinformatic analyses of integral membrane transport proteins encoded within the genome of the planctomycetes species, Rhodopirellula baltica Philipp Paparoditis a, Åke Västermark a,AndrewJ.Lea, John A. Fuerst b, Milton H. Saier Jr. a,⁎ a Department of Molecular Biology, Division of Biological Sciences, University of California at San Diego, La Jolla, CA, 92093–0116, USA b School of Chemistry and Molecular Biosciences, University of Queensland, Brisbane, Queensland, 9072, Australia article info abstract Article history: Rhodopirellula baltica (R. baltica) is a Planctomycete, known to have intracellular membranes. Because of its un- Received 12 April 2013 usual cell structure and ecological significance, we have conducted comprehensive analyses of its transmembrane Received in revised form 8 August 2013 transport proteins. The complete proteome of R. baltica was screened against the Transporter Classification Data- Accepted 9 August 2013 base (TCDB) to identify recognizable integral membrane transport proteins. 342 proteins were identified with a Available online 19 August 2013 high degree of confidence, and these fell into several different classes. -

Transcriptional Regulation of RKIP in Prostate Cancer Progression
Health Science Campus FINAL APPROVAL OF DISSERTATION Doctor of Philosophy in Biomedical Sciences Transcriptional Regulation of RKIP in Prostate Cancer Progression Submitted by: Sandra Marie Beach In partial fulfillment of the requirements for the degree of Doctor of Philosophy in Biomedical Sciences Examination Committee Major Advisor: Kam Yeung, Ph.D. Academic William Maltese, Ph.D. Advisory Committee: Sonia Najjar, Ph.D. Han-Fei Ding, M.D., Ph.D. Manohar Ratnam, Ph.D. Senior Associate Dean College of Graduate Studies Michael S. Bisesi, Ph.D. Date of Defense: May 16, 2007 Transcriptional Regulation of RKIP in Prostate Cancer Progression Sandra Beach University of Toledo ACKNOWLDEGMENTS I thank my major advisor, Dr. Kam Yeung, for the opportunity to pursue my degree in his laboratory. I am also indebted to my advisory committee members past and present, Drs. Sonia Najjar, Han-Fei Ding, Manohar Ratnam, James Trempe, and Douglas Pittman for generously and judiciously guiding my studies and sharing reagents and equipment. I owe extended thanks to Dr. William Maltese as a committee member and chairman of my department for supporting my degree progress. The entire Department of Biochemistry and Cancer Biology has been most kind and helpful to me. Drs. Roy Collaco and Hong-Juan Cui have shared their excellent technical and practical advice with me throughout my studies. I thank members of the Yeung laboratory, Dr. Sungdae Park, Hui Hui Tang, Miranda Yeung for their support and collegiality. The data mining studies herein would not have been possible without the helpful advice of Dr. Robert Trumbly. I am also grateful for the exceptional assistance and shared microarray data of Dr. -

A Computational Approach for Defining a Signature of Β-Cell Golgi Stress in Diabetes Mellitus
Page 1 of 781 Diabetes A Computational Approach for Defining a Signature of β-Cell Golgi Stress in Diabetes Mellitus Robert N. Bone1,6,7, Olufunmilola Oyebamiji2, Sayali Talware2, Sharmila Selvaraj2, Preethi Krishnan3,6, Farooq Syed1,6,7, Huanmei Wu2, Carmella Evans-Molina 1,3,4,5,6,7,8* Departments of 1Pediatrics, 3Medicine, 4Anatomy, Cell Biology & Physiology, 5Biochemistry & Molecular Biology, the 6Center for Diabetes & Metabolic Diseases, and the 7Herman B. Wells Center for Pediatric Research, Indiana University School of Medicine, Indianapolis, IN 46202; 2Department of BioHealth Informatics, Indiana University-Purdue University Indianapolis, Indianapolis, IN, 46202; 8Roudebush VA Medical Center, Indianapolis, IN 46202. *Corresponding Author(s): Carmella Evans-Molina, MD, PhD ([email protected]) Indiana University School of Medicine, 635 Barnhill Drive, MS 2031A, Indianapolis, IN 46202, Telephone: (317) 274-4145, Fax (317) 274-4107 Running Title: Golgi Stress Response in Diabetes Word Count: 4358 Number of Figures: 6 Keywords: Golgi apparatus stress, Islets, β cell, Type 1 diabetes, Type 2 diabetes 1 Diabetes Publish Ahead of Print, published online August 20, 2020 Diabetes Page 2 of 781 ABSTRACT The Golgi apparatus (GA) is an important site of insulin processing and granule maturation, but whether GA organelle dysfunction and GA stress are present in the diabetic β-cell has not been tested. We utilized an informatics-based approach to develop a transcriptional signature of β-cell GA stress using existing RNA sequencing and microarray datasets generated using human islets from donors with diabetes and islets where type 1(T1D) and type 2 diabetes (T2D) had been modeled ex vivo. To narrow our results to GA-specific genes, we applied a filter set of 1,030 genes accepted as GA associated. -

Tepzz¥ 6Z54za T
(19) TZZ¥ ZZ_T (11) EP 3 260 540 A1 (12) EUROPEAN PATENT APPLICATION (43) Date of publication: (51) Int Cl.: 27.12.2017 Bulletin 2017/52 C12N 15/113 (2010.01) A61K 9/127 (2006.01) A61K 31/713 (2006.01) C12Q 1/68 (2006.01) (21) Application number: 17000579.7 (22) Date of filing: 12.11.2011 (84) Designated Contracting States: • Sarma, Kavitha AL AT BE BG CH CY CZ DE DK EE ES FI FR GB Philadelphia, PA 19146 (US) GR HR HU IE IS IT LI LT LU LV MC MK MT NL NO • Borowsky, Mark PL PT RO RS SE SI SK SM TR Needham, MA 02494 (US) • Ohsumi, Toshiro Kendrick (30) Priority: 12.11.2010 US 412862 P Cambridge, MA 02141 (US) 20.12.2010 US 201061425174 P 28.07.2011 US 201161512754 P (74) Representative: Clegg, Richard Ian et al Mewburn Ellis LLP (62) Document number(s) of the earlier application(s) in City Tower accordance with Art. 76 EPC: 40 Basinghall Street 11840099.3 / 2 638 163 London EC2V 5DE (GB) (71) Applicant: The General Hospital Corporation Remarks: Boston, MA 02114 (US) •Thecomplete document including Reference Tables and the Sequence Listing can be downloaded from (72) Inventors: the EPO website • Lee, Jeannie T •This application was filed on 05-04-2017 as a Boston, MA 02114 (US) divisional application to the application mentioned • Zhao, Jing under INID code 62. San Diego, CA 92122 (US) •Claims filed after the date of receipt of the divisional application (Rule 68(4) EPC). (54) POLYCOMB-ASSOCIATED NON-CODING RNAS (57) This invention relates to long non-coding RNAs (IncRNAs), libraries of those ncRNAs that bind chromatin modifiers, such as Polycomb Repressive Complex 2, inhibitory nucleic acids and methods and compositions for targeting IncRNAs. -

HNRPH1 (HNRNPH1) (NM 005520) Human Tagged ORF Clone Product Data
OriGene Technologies, Inc. 9620 Medical Center Drive, Ste 200 Rockville, MD 20850, US Phone: +1-888-267-4436 [email protected] EU: [email protected] CN: [email protected] Product datasheet for RC201834 HNRPH1 (HNRNPH1) (NM_005520) Human Tagged ORF Clone Product data: Product Type: Expression Plasmids Product Name: HNRPH1 (HNRNPH1) (NM_005520) Human Tagged ORF Clone Tag: Myc-DDK Symbol: HNRNPH1 Synonyms: hnRNPH; HNRPH; HNRPH1 Vector: pCMV6-Entry (PS100001) E. coli Selection: Kanamycin (25 ug/mL) Cell Selection: Neomycin This product is to be used for laboratory only. Not for diagnostic or therapeutic use. View online » ©2021 OriGene Technologies, Inc., 9620 Medical Center Drive, Ste 200, Rockville, MD 20850, US 1 / 6 HNRPH1 (HNRNPH1) (NM_005520) Human Tagged ORF Clone – RC201834 ORF Nucleotide >RC201834 ORF sequence Sequence: Red=Cloning site Blue=ORF Green=Tags(s) TTTTGTAATACGACTCACTATAGGGCGGCCGGGAATTCGTCGACTGGATCCGGTACCGAGGAGATCTGCC GCCGCGATCGCC ATGATGTTGGGCACGGAAGGTGGAGAGGGATTCGTGGTGAAGGTCCGGGGCTTGCCCTGGTCTTGCTCGG CCGATGAAGTGCAGAGGTTTTTTTCTGACTGCAAAATTCAAAATGGGGCTCAAGGTATTCGTTTCATCTA CACCAGAGAAGGCAGACCAAGTGGCGAGGCTTTTGTTGAACTTGAATCAGAAGATGAAGTCAAATTGGCC CTGAAAAAAGACAGAGAAACTATGGGACACAGATATGTTGAAGTATTCAAGTCAAACAACGTTGAAATGG ATTGGGTGTTGAAGCATACTGGTCCAAATAGTCCTGACACGGCCAATGATGGCTTTGTACGGCTTAGAGG ACTTCCCTTTGGATGTAGCAAGGAAGAAATTGTTCAGTTCTTCTCAGGGTTGGAAATCGTGCCAAATGGG ATAACATTGCCGGTGGACTTCCAGGGGAGGAGTACGGGGGAGGCCTTCGTGCAGTTTGCTTCACAGGAAA TAGCTGAAAAGGCTCTAAAGAAACACAAGGAAAGAATAGGGCACAGGTATATTGAAATCTTTAAGAGCAG TAGAGCTGAAGTTAGAACTCATTATGATCCACCACGAAAGCTTATGGCCATGCAGCGGCCAGGTCCTTAT -

Supplementary Table 1: Adhesion Genes Data Set
Supplementary Table 1: Adhesion genes data set PROBE Entrez Gene ID Celera Gene ID Gene_Symbol Gene_Name 160832 1 hCG201364.3 A1BG alpha-1-B glycoprotein 223658 1 hCG201364.3 A1BG alpha-1-B glycoprotein 212988 102 hCG40040.3 ADAM10 ADAM metallopeptidase domain 10 133411 4185 hCG28232.2 ADAM11 ADAM metallopeptidase domain 11 110695 8038 hCG40937.4 ADAM12 ADAM metallopeptidase domain 12 (meltrin alpha) 195222 8038 hCG40937.4 ADAM12 ADAM metallopeptidase domain 12 (meltrin alpha) 165344 8751 hCG20021.3 ADAM15 ADAM metallopeptidase domain 15 (metargidin) 189065 6868 null ADAM17 ADAM metallopeptidase domain 17 (tumor necrosis factor, alpha, converting enzyme) 108119 8728 hCG15398.4 ADAM19 ADAM metallopeptidase domain 19 (meltrin beta) 117763 8748 hCG20675.3 ADAM20 ADAM metallopeptidase domain 20 126448 8747 hCG1785634.2 ADAM21 ADAM metallopeptidase domain 21 208981 8747 hCG1785634.2|hCG2042897 ADAM21 ADAM metallopeptidase domain 21 180903 53616 hCG17212.4 ADAM22 ADAM metallopeptidase domain 22 177272 8745 hCG1811623.1 ADAM23 ADAM metallopeptidase domain 23 102384 10863 hCG1818505.1 ADAM28 ADAM metallopeptidase domain 28 119968 11086 hCG1786734.2 ADAM29 ADAM metallopeptidase domain 29 205542 11085 hCG1997196.1 ADAM30 ADAM metallopeptidase domain 30 148417 80332 hCG39255.4 ADAM33 ADAM metallopeptidase domain 33 140492 8756 hCG1789002.2 ADAM7 ADAM metallopeptidase domain 7 122603 101 hCG1816947.1 ADAM8 ADAM metallopeptidase domain 8 183965 8754 hCG1996391 ADAM9 ADAM metallopeptidase domain 9 (meltrin gamma) 129974 27299 hCG15447.3 ADAMDEC1 ADAM-like, -

Nematostella Genome
Sea anemone genome reveals the gene repertoire and genomic organization of the eumetazoan ancestor Nicholas H. Putnam[1], Mansi Srivastava[2], Uffe Hellsten[1], Bill Dirks[2], Jarrod Chapman[1], Asaf Salamov[1], Astrid Terry[1], Harris Shapiro[1], Erika Lindquist[1], Vladimir V. Kapitonov[3], Jerzy Jurka[3], Grigory Genikhovich[4], Igor Grigoriev[1], JGI Sequencing Team[1], Robert E. Steele[5], John Finnerty[6], Ulrich Technau[4], Mark Q. Martindale[7], Daniel S. Rokhsar[1,2] [1] Department of Energy Joint Genome Institute, Walnut Creek, CA 94598 [2] Center for Integrative Genomics and Department of Molecular and Cell Biology, University of California, Berkeley CA 94720 [3] Genetic Information Research Institute, 1925 Landings Drive, Mountain View, CA 94043 [4] Sars International Centre for Marine Molecular Biology, University of Bergen, Thormoeøhlensgt 55; 5008, Bergen, Norway [5] Department of Biological Chemistry and the Developmental Biology Center, University of California, Irvine, CA 92697 [6] Department of Biology, Boston University, Boston, MA 02215 [7] Kewalo Marine Laboratory, University of Hawaii, Honolulu, HI 96813 Abstract Sea anemones are seemingly primitive animals that, along with corals, jellyfish, and hydras, constitute the Cnidaria, the oldest eumetazoan phylum. Here we report a comparative analysis of the draft genome of an emerging cnidarian model, the starlet anemone Nematostella vectensis. The anemone genome is surprisingly complex, with a gene repertoire, exon-intron structure, and large-scale gene linkage more similar to vertebrates than to flies or nematodes. These results imply that the genome of the eumetazoan ancestor was similarly complex, and that fly and nematode genomes have been modified via sequence divergence, gene and intron loss, and genomic rearrangement. -

9, 2015 Glasgow, Scotland, United Kingdom Abstracts
Volume 23 Supplement 1 June 2015 www.nature.com/ejhg European Human Genetics Conference 2015 June 6 - 9, 2015 Glasgow, Scotland, United Kingdom Abstracts EJHG_OFC.indd 1 4/1/2006 10:58:05 AM ABSTRACTS European Human Genetics Conference joint with the British Society of Genetics Medicine June 6 - 9, 2015 Glasgow, Scotland, United Kingdom Abstracts ESHG 2015 | GLASGOW, SCOTLAND, UK | WWW.ESHG.ORG 1 ABSTRACTS Committees – Board - Organisation European Society of Human Genetics ESHG Office Executive Board 2014-2015 Scientific Programme Committee European Society President Chair of Human Genetics Helena Kääriäinen, FI Brunhilde Wirth, DE Andrea Robinson Vice-President Members Karin Knob Han Brunner, NL Tara Clancy, UK c/o Vienna Medical Academy Martina Cornel, NL Alser Strasse 4 President-Elect Yanick Crow, FR 1090 Vienna Feliciano Ramos, ES Paul de Bakker, NL Austria Secretary-General Helene Dollfus, FR T: 0043 1 405 13 83 20 or 35 Gunnar Houge, NO David FitzPatrick, UK F: 0043 1 407 82 74 Maurizio Genuardi, IT E: [email protected] Deputy-Secretary-General Daniel Grinberg, ES www.eshg.org Karin Writzl, SI Gunnar Houge, NO Treasurer Erik Iwarsson, SE Andrew Read, UK Xavier Jeunemaitre, FR Mark Longmuir, UK Executive Officer Jose C. Machado, PT Jerome del Picchia, AT Dominic McMullan, UK Giovanni Neri, IT William Newman, UK Minna Nyström, FI Pia Ostergaard, UK Francesc Palau, ES Anita Rauch, CH Samuli Ripatti, FI Peter N. Robinson, DE Kristel van Steen, BE Joris Veltman, NL Joris Vermeesch, BE Emma Woodward, UK Karin Writzl, SI Board Members Liaison Members Yasemin Alanay, TR Stan Lyonnet, FR Martina Cornel, NL Martijn Breuning, NL Julie McGaughran, AU Ulf Kristoffersson, SE Pascal Borry, BE Bela Melegh, HU Thomas Liehr, DE Nina Canki-Klain, HR Will Newman, UK Milan Macek Jr., CZ Ana Carrió, ES Markus Nöthen, DE Tayfun Ozcelik, TR Isabella Ceccherini, IT Markus Perola, FI Milena Paneque, PT Angus John Clarke, UK Dijana Plaseska-Karanfilska, MK Hans Scheffer, NL Koen Devriendt, BE Trine E. -

Genetic and Pharmacological Approaches to Preventing Neurodegeneration
University of Pennsylvania ScholarlyCommons Publicly Accessible Penn Dissertations 2012 Genetic and Pharmacological Approaches to Preventing Neurodegeneration Marco Boccitto University of Pennsylvania, [email protected] Follow this and additional works at: https://repository.upenn.edu/edissertations Part of the Neuroscience and Neurobiology Commons Recommended Citation Boccitto, Marco, "Genetic and Pharmacological Approaches to Preventing Neurodegeneration" (2012). Publicly Accessible Penn Dissertations. 494. https://repository.upenn.edu/edissertations/494 This paper is posted at ScholarlyCommons. https://repository.upenn.edu/edissertations/494 For more information, please contact [email protected]. Genetic and Pharmacological Approaches to Preventing Neurodegeneration Abstract The Insulin/Insulin-like Growth Factor 1 Signaling (IIS) pathway was first identified as a major modifier of aging in C.elegans. It has since become clear that the ability of this pathway to modify aging is phylogenetically conserved. Aging is a major risk factor for a variety of neurodegenerative diseases including the motor neuron disease, Amyotrophic Lateral Sclerosis (ALS). This raises the possibility that the IIS pathway might have therapeutic potential to modify the disease progression of ALS. In a C. elegans model of ALS we found that decreased IIS had a beneficial effect on ALS pathology in this model. This beneficial effect was dependent on activation of the transcription factor daf-16. To further validate IIS as a potential therapeutic target for treatment of ALS, manipulations of IIS in mammalian cells were investigated for neuroprotective activity. Genetic manipulations that increase the activity of the mammalian ortholog of daf-16, FOXO3, were found to be neuroprotective in a series of in vitro models of ALS toxicity. -

Advances in Prognostic Methylation Biomarkers for Prostate Cancer
cancers Review Advances in Prognostic Methylation Biomarkers for Prostate Cancer 1 1,2 1,2, 1,2, , Dilys Lam , Susan Clark , Clare Stirzaker y and Ruth Pidsley * y 1 Epigenetics Research Laboratory, Genomics and Epigenetics Division, Garvan Institute of Medical Research, Sydney, New South Wales 2010, Australia; [email protected] (D.L.); [email protected] (S.C.); [email protected] (C.S.) 2 St. Vincent’s Clinical School, University of New South Wales, Sydney, New South Wales 2010, Australia * Correspondence: [email protected]; Tel.: +61-2-92958315 These authors have contributed equally. y Received: 22 September 2020; Accepted: 13 October 2020; Published: 15 October 2020 Simple Summary: Prostate cancer is a major cause of cancer-related death in men worldwide. There is an urgent clinical need for improved prognostic biomarkers to better predict the likely outcome and course of the disease and thus inform the clinical management of these patients. Currently, clinically recognised prognostic markers lack sensitivity and specificity in distinguishing aggressive from indolent disease, particularly in patients with localised, intermediate grade prostate cancer. Thus, there is major interest in identifying new molecular biomarkers to complement existing standard clinicopathological markers. DNA methylation is a frequent alteration in the cancer genome and offers potential as a reliable and robust biomarker. In this review, we provide a comprehensive overview of the current state of DNA methylation biomarker studies in prostate cancer prognosis. We highlight advances in this field that have enabled the discovery of novel prognostic genes and discuss the potential of methylation biomarkers for noninvasive liquid-biopsy testing. -
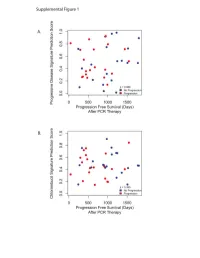
Supplementary Data
Progressive Disease Signature Upregulated probes with progressive disease U133Plus2 ID Gene Symbol Gene Name 239673_at NR3C2 nuclear receptor subfamily 3, group C, member 2 228994_at CCDC24 coiled-coil domain containing 24 1562245_a_at ZNF578 zinc finger protein 578 234224_at PTPRG protein tyrosine phosphatase, receptor type, G 219173_at NA NA 218613_at PSD3 pleckstrin and Sec7 domain containing 3 236167_at TNS3 tensin 3 1562244_at ZNF578 zinc finger protein 578 221909_at RNFT2 ring finger protein, transmembrane 2 1552732_at ABRA actin-binding Rho activating protein 59375_at MYO15B myosin XVB pseudogene 203633_at CPT1A carnitine palmitoyltransferase 1A (liver) 1563120_at NA NA 1560098_at AKR1C2 aldo-keto reductase family 1, member C2 (dihydrodiol dehydrogenase 2; bile acid binding pro 238576_at NA NA 202283_at SERPINF1 serpin peptidase inhibitor, clade F (alpha-2 antiplasmin, pigment epithelium derived factor), m 214248_s_at TRIM2 tripartite motif-containing 2 204766_s_at NUDT1 nudix (nucleoside diphosphate linked moiety X)-type motif 1 242308_at MCOLN3 mucolipin 3 1569154_a_at NA NA 228171_s_at PLEKHG4 pleckstrin homology domain containing, family G (with RhoGef domain) member 4 1552587_at CNBD1 cyclic nucleotide binding domain containing 1 220705_s_at ADAMTS7 ADAM metallopeptidase with thrombospondin type 1 motif, 7 232332_at RP13-347D8.3 KIAA1210 protein 1553618_at TRIM43 tripartite motif-containing 43 209369_at ANXA3 annexin A3 243143_at FAM24A family with sequence similarity 24, member A 234742_at SIRPG signal-regulatory protein gamma