Identification of Genetic Variation Using Next-Generation Sequencing
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

DNA Sequencing
1 DNA Sequencing Description of Module Subject Name Paper Name Module Name/Title DNA Sequencing Dr. Vijaya Khader Dr. MC Varadaraj 2 DNA Sequencing 1. Objectives 1. DNA sequencing Introduction 2. Types of methods available 3. Next generation sequencing 2. Lay Out 3 DNA Sequencing Sequencing Maxam Gilbert Sanger's Next generation 3 Introduction 4 DNA Sequencing In the mid 1970s when molecular cloning techniques in general were rapidly improving, simple methods were also developed to determine the nucleotide sequence of DNA. These advances laid the foundation for the detailed analysis of the structure and function of the large number of genes. Other fields which utilize DNA sequencing include diagnostic and forensic biology. With the invention of DNA sequencing, research and discovery of new genes is increased many folds. Using this technique whole genomes of many animal (human), plant, and microbial genomes have been sequenced. The first attempts to sequence DNA mirrored techniques developed in 1960s to sequence RNA. These involved : a) Specific cleavage to the DNA in to smaller fragments by enzymatic digestion or chemical digestion b) Nearest neighbour analysis c) Wandering spot method. Indeed in some studies the DNA was transcribed in to RNA with E.coli RNA polymerase and then sequenced as RNA So there were several questions unanswered; How many base pairs (bp) are there in a human genome?( ~3 billion (haploid)) How much did it cost to sequence the first human genome? ~$2.7 billion How long did it take to sequence the first human genome?( ~13 years) When was the first human genome sequence complete? (2000-2003) Goal figuring the order of nucleotides across a genome Problem Current DNA sequencing methods can handle only short stretches of DNA at once (<1-2Kbp) Solution 5 DNA Sequencing Sequence and then use computers to assemble the small pieces 3.1 Maxam-Gilbert sequencing Also known as chemical sequencing, here there is use radioactive labeling at one 5' end of the DNA fragment which is to be sequenced. -

Targeted Genes and Methodology Details for Neuromuscular Genetic Panels
Targeted Genes and Methodology Details for Neuromuscular Genetic Panels Reference transcripts based on build GRCh37 (hg19) interrogated by Neuromuscular Genetic Panels Next-generation sequencing (NGS) and/or Sanger sequencing is performed Motor Neuron Disease Panel to test for the presence of a mutation in these genes. Gene GenBank Accession Number Regions of homology, high GC-rich content, and repetitive sequences may ALS2 NM_020919 not provide accurate sequence. Therefore, all reported alterations detected ANG NM_001145 by NGS are confirmed by an independent reference method based on laboratory developed criteria. However, this does not rule out the possibility CHMP2B NM_014043 of a false-negative result in these regions. ERBB4 NM_005235 Sanger sequencing is used to confirm alterations detected by NGS when FIG4 NM_014845 appropriate.(Unpublished Mayo method) FUS NM_004960 HNRNPA1 NM_031157 OPTN NM_021980 PFN1 NM_005022 SETX NM_015046 SIGMAR1 NM_005866 SOD1 NM_000454 SQSTM1 NM_003900 TARDBP NM_007375 UBQLN2 NM_013444 VAPB NM_004738 VCP NM_007126 ©2018 Mayo Foundation for Medical Education and Research Page 1 of 14 MC4091-83rev1018 Muscular Dystrophy Panel Muscular Dystrophy Panel Gene GenBank Accession Number Gene GenBank Accession Number ACTA1 NM_001100 LMNA NM_170707 ANO5 NM_213599 LPIN1 NM_145693 B3GALNT2 NM_152490 MATR3 NM_199189 B4GAT1 NM_006876 MYH2 NM_017534 BAG3 NM_004281 MYH7 NM_000257 BIN1 NM_139343 MYOT NM_006790 BVES NM_007073 NEB NM_004543 CAPN3 NM_000070 PLEC NM_000445 CAV3 NM_033337 POMGNT1 NM_017739 CAVIN1 NM_012232 POMGNT2 -

Preclinical Research 2020 Sections
Sackler Faculty of Medicine Preclinical Research 2020 Sections Cancer and Molecular Therapies 8 Dental Health and Medicine 52 Diabetes, Metabolic and Endocrine Diseases 62 Genomics & Personalized Medicine 78 Hearing, Language & Speech Sciences and Disorders 98 Infectious Diseases 117 Inflammatory and Autoimmune Diseases 137 Medical Education and Ethics 147 Nervous System and Brain Disorders 154 Nursing, Occupational and Physical Therapy 200 Public Health 235 Reproduction, Development and Evolution 263 Stem Cells and Regenerative Medicine 280 Cover images (from bottom left, clockwise): Image 1: Human embryonic stem cell derived cardiomyocytes stained with fluorescent antibodies. The cardiac marker alpha-actinin (green), calcium channel modulator, Ahnak1 (red) – Shimrit Oz, Nathan Dascal. Image 2: Islet of Langerhans containing insulin-producing beta-cells (green) and glucagon- producing alpha-cells (red) – Daria Baer, Limor Landsman. Image 3: β-catenin in C. elegans vulva – Michal Caspi, Limor Broday, Rina Rosin-Arbesfeld. Image 4: Stereocilia of a sensory outer hair cell from a mouse inner ear – Shaked Shivatzki, Karen Avraham. Image 5: Electron scanning micrograph of middle ear ossicles from a mouse ear stained with pseudo colors – Shaked Shivatski, Karen Avraham. Image 6: Resistin-like molecule alpha (red), eosinophil major basic protein (green) and DAPI (blue) staining of asthmatic mice – Danielle Karo-Atar, Ariel Munitz. © All rights reserved Editor: Prof. Karen Avraham Graphic design: Michal Semo Kovetz, TAU Graphic Design Studio February 2020 Sackler Faculty of Medicine Research 2020 2 The Sackler Faculty of Medicine The Sackler Faculty of Medicine is Israel’s largest diabetes, neurodegenerative diseases, infectious medical research and training complex. The Sackler diseases and genetic diseases, including but not Faculty of Medicine of Tel Aviv University (TAU) was imited to Alzheimer’s disease, Parkinson’s disease founded in 1964 following the generous contributions and HIV/AIDS. -

A Computational Approach for Defining a Signature of Β-Cell Golgi Stress in Diabetes Mellitus
Page 1 of 781 Diabetes A Computational Approach for Defining a Signature of β-Cell Golgi Stress in Diabetes Mellitus Robert N. Bone1,6,7, Olufunmilola Oyebamiji2, Sayali Talware2, Sharmila Selvaraj2, Preethi Krishnan3,6, Farooq Syed1,6,7, Huanmei Wu2, Carmella Evans-Molina 1,3,4,5,6,7,8* Departments of 1Pediatrics, 3Medicine, 4Anatomy, Cell Biology & Physiology, 5Biochemistry & Molecular Biology, the 6Center for Diabetes & Metabolic Diseases, and the 7Herman B. Wells Center for Pediatric Research, Indiana University School of Medicine, Indianapolis, IN 46202; 2Department of BioHealth Informatics, Indiana University-Purdue University Indianapolis, Indianapolis, IN, 46202; 8Roudebush VA Medical Center, Indianapolis, IN 46202. *Corresponding Author(s): Carmella Evans-Molina, MD, PhD ([email protected]) Indiana University School of Medicine, 635 Barnhill Drive, MS 2031A, Indianapolis, IN 46202, Telephone: (317) 274-4145, Fax (317) 274-4107 Running Title: Golgi Stress Response in Diabetes Word Count: 4358 Number of Figures: 6 Keywords: Golgi apparatus stress, Islets, β cell, Type 1 diabetes, Type 2 diabetes 1 Diabetes Publish Ahead of Print, published online August 20, 2020 Diabetes Page 2 of 781 ABSTRACT The Golgi apparatus (GA) is an important site of insulin processing and granule maturation, but whether GA organelle dysfunction and GA stress are present in the diabetic β-cell has not been tested. We utilized an informatics-based approach to develop a transcriptional signature of β-cell GA stress using existing RNA sequencing and microarray datasets generated using human islets from donors with diabetes and islets where type 1(T1D) and type 2 diabetes (T2D) had been modeled ex vivo. To narrow our results to GA-specific genes, we applied a filter set of 1,030 genes accepted as GA associated. -

Conserved and Novel Properties of Clathrin-Mediated Endocytosis in Dictyostelium Discoideum" (2012)
Rockefeller University Digital Commons @ RU Student Theses and Dissertations 2012 Conserved and Novel Properties of Clathrin- Mediated Endocytosis in Dictyostelium Discoideum Laura Macro Follow this and additional works at: http://digitalcommons.rockefeller.edu/ student_theses_and_dissertations Part of the Life Sciences Commons Recommended Citation Macro, Laura, "Conserved and Novel Properties of Clathrin-Mediated Endocytosis in Dictyostelium Discoideum" (2012). Student Theses and Dissertations. Paper 163. This Thesis is brought to you for free and open access by Digital Commons @ RU. It has been accepted for inclusion in Student Theses and Dissertations by an authorized administrator of Digital Commons @ RU. For more information, please contact [email protected]. CONSERVED AND NOVEL PROPERTIES OF CLATHRIN- MEDIATED ENDOCYTOSIS IN DICTYOSTELIUM DISCOIDEUM A Thesis Presented to the Faculty of The Rockefeller University in Partial Fulfillment of the Requirements for the degree of Doctor of Philosophy by Laura Macro June 2012 © Copyright by Laura Macro 2012 CONSERVED AND NOVEL PROPERTIES OF CLATHRIN- MEDIATED ENDOCYTOSIS IN DICTYOSTELIUM DISCOIDEUM Laura Macro, Ph.D. The Rockefeller University 2012 The protein clathrin mediates one of the major pathways of endocytosis from the extracellular milieu and plasma membrane. Clathrin functions with a network of interacting accessory proteins, one of which is the adaptor complex AP-2, to co-ordinate vesicle formation. Disruption of genes involved in clathrin-mediated endocytosis causes embryonic lethality in multicellular animals suggesting that clathrin-mediated endocytosis is a fundamental cellular process. However, loss of clathrin-mediated endocytosis genes in single cell eukaryotes, such as S.cerevisiae (yeast), does not cause lethality, suggesting that clathrin may convey specific advantages for multicellularity. -

DNA Sequencing - I
SUBJECT FORENSIC SCIENCE Paper No. and Title PAPER No.13: DNA Forensics Module No. and Title MODULE No.21: DNA Sequencing - I Module Tag FSC_P13_M21 FORENSIC SCIENCE PAPER No.13: DNA Forensics MODULE No.21: DNA Sequencing - I TABLE OF CONTENTS 1. Learning Outcomes 2. Introduction 3. First Generation Sequencing Methods 4. Second Generation Sequencing Techniques 5. Third Generation Sequencing – emerging technologies 6. DNA Sequence analysis 7. Summary FORENSIC SCIENCE PAPER No.13: DNA Forensics MODULE No.21: DNA Sequencing - I 1. Learning Outcomes After studying this module, reader shall be able to understand - DNA Sequencing Methods of DNA Sequencing DNA sequence analysis 2. Introduction In 1953, James Watson and Francis Crick discovered double-helix model of DNA, based on crystallized X-ray structures studied by Rosalind Franklin. As per this model, DNA comprises of two strands of nucleotides coiled around each other, allied by hydrogen bonds and moving in opposite directions. Each strand is composed of four complementary nucleotides – adenine (A), cytosine (C), guanine (G) and thymine (T) – with A always paired with T and C always paired with G with 2 & 3 hydrogen bonds respectively. The sequence of the bases (A,T,G,C) along DNA contains the complete set of instructions that make up the genetic inheritance. Defining the arrangement of these nucleotide bases in DNA strand is a primary step in assessing regulatory sequences, coding and non-coding regions. The term DNA sequencing denotes to methods for identifying the sequence of these nucleotides bases in a molecule of DNA. The basis for sequencing proteins was initially placed by the effort of Fred Sanger who by 1955 had accomplished the arrangement of all the amino acids in insulin, a small protein produced by the pancreas. -
NICU Gene List Generator.Xlsx
Neonatal Crisis Sequencing Panel Gene List Genes: A2ML1 - B3GLCT A2ML1 ADAMTS9 ALG1 ARHGEF15 AAAS ADAMTSL2 ALG11 ARHGEF9 AARS1 ADAR ALG12 ARID1A AARS2 ADARB1 ALG13 ARID1B ABAT ADCY6 ALG14 ARID2 ABCA12 ADD3 ALG2 ARL13B ABCA3 ADGRG1 ALG3 ARL6 ABCA4 ADGRV1 ALG6 ARMC9 ABCB11 ADK ALG8 ARPC1B ABCB4 ADNP ALG9 ARSA ABCC6 ADPRS ALK ARSL ABCC8 ADSL ALMS1 ARX ABCC9 AEBP1 ALOX12B ASAH1 ABCD1 AFF3 ALOXE3 ASCC1 ABCD3 AFF4 ALPK3 ASH1L ABCD4 AFG3L2 ALPL ASL ABHD5 AGA ALS2 ASNS ACAD8 AGK ALX3 ASPA ACAD9 AGL ALX4 ASPM ACADM AGPS AMELX ASS1 ACADS AGRN AMER1 ASXL1 ACADSB AGT AMH ASXL3 ACADVL AGTPBP1 AMHR2 ATAD1 ACAN AGTR1 AMN ATL1 ACAT1 AGXT AMPD2 ATM ACE AHCY AMT ATP1A1 ACO2 AHDC1 ANK1 ATP1A2 ACOX1 AHI1 ANK2 ATP1A3 ACP5 AIFM1 ANKH ATP2A1 ACSF3 AIMP1 ANKLE2 ATP5F1A ACTA1 AIMP2 ANKRD11 ATP5F1D ACTA2 AIRE ANKRD26 ATP5F1E ACTB AKAP9 ANTXR2 ATP6V0A2 ACTC1 AKR1D1 AP1S2 ATP6V1B1 ACTG1 AKT2 AP2S1 ATP7A ACTG2 AKT3 AP3B1 ATP8A2 ACTL6B ALAS2 AP3B2 ATP8B1 ACTN1 ALB AP4B1 ATPAF2 ACTN2 ALDH18A1 AP4M1 ATR ACTN4 ALDH1A3 AP4S1 ATRX ACVR1 ALDH3A2 APC AUH ACVRL1 ALDH4A1 APTX AVPR2 ACY1 ALDH5A1 AR B3GALNT2 ADA ALDH6A1 ARFGEF2 B3GALT6 ADAMTS13 ALDH7A1 ARG1 B3GAT3 ADAMTS2 ALDOB ARHGAP31 B3GLCT Updated: 03/15/2021; v.3.6 1 Neonatal Crisis Sequencing Panel Gene List Genes: B4GALT1 - COL11A2 B4GALT1 C1QBP CD3G CHKB B4GALT7 C3 CD40LG CHMP1A B4GAT1 CA2 CD59 CHRNA1 B9D1 CA5A CD70 CHRNB1 B9D2 CACNA1A CD96 CHRND BAAT CACNA1C CDAN1 CHRNE BBIP1 CACNA1D CDC42 CHRNG BBS1 CACNA1E CDH1 CHST14 BBS10 CACNA1F CDH2 CHST3 BBS12 CACNA1G CDK10 CHUK BBS2 CACNA2D2 CDK13 CILK1 BBS4 CACNB2 CDK5RAP2 -

Supplementary Information
Electronic Supplementary Material (ESI) for Integrative Biology This journal is © The Royal Society of Chemistry 2012 SUPPLEMENTARY INFORMATION: Table S1 : A comparative account of degree in 228 coexpressing genes in normal (CONT), unipolar depression (DEP), bipolar disorder (BPD) and schizophrenia (SCZ). GENE CONT DEP BPD SCZ NRG1 88 89 105 96 DISC1 69 108 69 72 COMT 82 78 87 73 BDNF 90 76 85 83 ISLR 67 76 97 94 POMZP3 68 88 78 87 KRT17 80 68 78 86 POLR2A 85 75 98 73 PIK3R3 74 77 83 104 EML2 70 71 75 76 DSTYK 74 71 87 72 NFASC 89 84 78 84 IKBKAP 80 85 83 102 GCN1L1 78 71 75 70 DOT1L 70 67 101 83 TLX1 67 85 74 74 PPRC1 69 74 103 79 OR2F1 76 72 80 70 CPSF4 81 70 107 86 PXDN 69 82 75 76 PKD1 67 76 101 76 GOLGA3 67 67 75 79 REM1 76 72 90 83 TUBA3CDE 86 82 79 91 TUBA3CD 108 78 102 90 GNE 73 77 70 79 ARIH2 69 71 97 70 WDR45 77 76 106 76 SNRNP70 82 68 73 78 FBXO21 73 75 75 71 CYP2B6 74 74 81 79 KPNA6 74 78 86 78 COL4A4 71 94 70 75 Electronic Supplementary Material (ESI) for Integrative Biology This journal is © The Royal Society of Chemistry 2012 PDPN 75 80 83 79 MRPL33 75 71 91 75 EFNA5 71 70 71 94 PSG9 72 78 67 78 SFRS16 74 73 75 67 CCT7 76 81 90 73 COL11A2 78 68 80 74 CNKSR1 74 81 64 103 CD9 71 85 74 93 EP400 70 79 76 81 AKAP8 75 76 98 77 SIVA1 71 72 100 71 IDO1 85 76 69 84 COX7A2L 67 86 97 77 SLC29A1 75 83 73 94 STX12 73 87 95 92 CSNK1E 75 76 71 78 CRKRS 82 99 76 72 TUBG1 100 78 86 92 IFNG 77 70 82 85 SUMO2-3 73 95 89 103 MTF1 62 69 91 81 ST3GAL6 75 82 74 67 COBRA1 69 75 82 73 CDV3 82 105 77 101 FOXH1 64 74 86 79 DPH1 79 66 93 77 HEG1 89 -

AP4S1 Splice-Site Mutation in a Case of Spastic Paraplegia Type 52 with Polymicrogyria
CLINICAL/SCIENTIFIC NOTES OPEN ACCESS AP4S1 splice-site mutation in a case of spastic paraplegia type 52 with polymicrogyria Susana Carmona, PhD, Clara Marecos, MD, Marta Amorim, MD, Ana C. Ferreira, MD, Carla Conceição, MD, Correspondence Jos´eBr´as, PhD, Sofia T. Duarte, MD, PhD,* and Rita Guerreiro, PhD* Dr. Guerreiro [email protected] Neurol Genet 2018;4:e273. doi:10.1212/NXG.0000000000000273 Hereditary spastic paraplegias (HSPs) are a group of rare inherited neurodegenerative dis- orders that result from primary retrograde dysfunction of the long descending fibers of the corticospinal tract, causing lower limb spasticity and muscular weakness. This group of diseases has a heterogeneous clinical presentation. An extensive list of associated genes, different in- heritance patterns, and ages at onset have been reported in HSPs.1 Spastic paraplegia type 52 (SPG52) is an autosomal recessive disease caused by AP4S1 mutations. The disease is char- acterized by neonatal hypotonia that progresses to hypertonia and spasticity in early childhood, developmental delay, mental retardation, and poor or absent speech. Febrile or afebrile seizures – may also occur.2 4 Clinical case presentation We report the case of a Portuguese 2-year-old boy born to healthy nonconsanguineous parents after a full-term gestation with intrauterine growth restriction after week 37. During the first months of life, the patient presented poor weight gain, hyperammonemia with elevation of glutamine and ornithine, low citrulline, and negative orotic acid. Weight recovery and nor- malization of amino acid profile were observed after protein restriction and remained normal after reintroduction of normal diet. Genetic study of urea cycle disorders (NAGS, CPS, and OTC) was negative. -
Cldn19 Clic2 Clmp Cln3
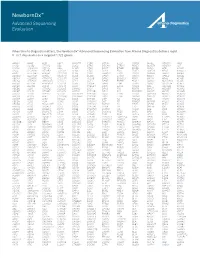
NewbornDx™ Advanced Sequencing Evaluation When time to diagnosis matters, the NewbornDx™ Advanced Sequencing Evaluation from Athena Diagnostics delivers rapid, 5- to 7-day results on a targeted 1,722-genes. A2ML1 ALAD ATM CAV1 CLDN19 CTNS DOCK7 ETFB FOXC2 GLUL HOXC13 JAK3 AAAS ALAS2 ATP1A2 CBL CLIC2 CTRC DOCK8 ETFDH FOXE1 GLYCTK HOXD13 JUP AARS2 ALDH18A1 ATP1A3 CBS CLMP CTSA DOK7 ETHE1 FOXE3 GM2A HPD KANK1 AASS ALDH1A2 ATP2B3 CC2D2A CLN3 CTSD DOLK EVC FOXF1 GMPPA HPGD K ANSL1 ABAT ALDH3A2 ATP5A1 CCDC103 CLN5 CTSK DPAGT1 EVC2 FOXG1 GMPPB HPRT1 KAT6B ABCA12 ALDH4A1 ATP5E CCDC114 CLN6 CUBN DPM1 EXOC4 FOXH1 GNA11 HPSE2 KCNA2 ABCA3 ALDH5A1 ATP6AP2 CCDC151 CLN8 CUL4B DPM2 EXOSC3 FOXI1 GNAI3 HRAS KCNB1 ABCA4 ALDH7A1 ATP6V0A2 CCDC22 CLP1 CUL7 DPM3 EXPH5 FOXL2 GNAO1 HSD17B10 KCND2 ABCB11 ALDOA ATP6V1B1 CCDC39 CLPB CXCR4 DPP6 EYA1 FOXP1 GNAS HSD17B4 KCNE1 ABCB4 ALDOB ATP7A CCDC40 CLPP CYB5R3 DPYD EZH2 FOXP2 GNE HSD3B2 KCNE2 ABCB6 ALG1 ATP8A2 CCDC65 CNNM2 CYC1 DPYS F10 FOXP3 GNMT HSD3B7 KCNH2 ABCB7 ALG11 ATP8B1 CCDC78 CNTN1 CYP11B1 DRC1 F11 FOXRED1 GNPAT HSPD1 KCNH5 ABCC2 ALG12 ATPAF2 CCDC8 CNTNAP1 CYP11B2 DSC2 F13A1 FRAS1 GNPTAB HSPG2 KCNJ10 ABCC8 ALG13 ATR CCDC88C CNTNAP2 CYP17A1 DSG1 F13B FREM1 GNPTG HUWE1 KCNJ11 ABCC9 ALG14 ATRX CCND2 COA5 CYP1B1 DSP F2 FREM2 GNS HYDIN KCNJ13 ABCD3 ALG2 AUH CCNO COG1 CYP24A1 DST F5 FRMD7 GORAB HYLS1 KCNJ2 ABCD4 ALG3 B3GALNT2 CCS COG4 CYP26C1 DSTYK F7 FTCD GP1BA IBA57 KCNJ5 ABHD5 ALG6 B3GAT3 CCT5 COG5 CYP27A1 DTNA F8 FTO GP1BB ICK KCNJ8 ACAD8 ALG8 B3GLCT CD151 COG6 CYP27B1 DUOX2 F9 FUCA1 GP6 ICOS KCNK3 ACAD9 ALG9 -

Deep Multiomics Profiling of Brain Tumors Identifies Signaling Networks
ARTICLE https://doi.org/10.1038/s41467-019-11661-4 OPEN Deep multiomics profiling of brain tumors identifies signaling networks downstream of cancer driver genes Hong Wang 1,2,3, Alexander K. Diaz3,4, Timothy I. Shaw2,5, Yuxin Li1,2,4, Mingming Niu1,4, Ji-Hoon Cho2, Barbara S. Paugh4, Yang Zhang6, Jeffrey Sifford1,4, Bing Bai1,4,10, Zhiping Wu1,4, Haiyan Tan2, Suiping Zhou2, Laura D. Hover4, Heather S. Tillman 7, Abbas Shirinifard8, Suresh Thiagarajan9, Andras Sablauer 8, Vishwajeeth Pagala2, Anthony A. High2, Xusheng Wang 2, Chunliang Li 6, Suzanne J. Baker4 & Junmin Peng 1,2,4 1234567890():,; High throughput omics approaches provide an unprecedented opportunity for dissecting molecular mechanisms in cancer biology. Here we present deep profiling of whole proteome, phosphoproteome and transcriptome in two high-grade glioma (HGG) mouse models driven by mutated RTK oncogenes, PDGFRA and NTRK1, analyzing 13,860 proteins and 30,431 phosphosites by mass spectrometry. Systems biology approaches identify numerous master regulators, including 41 kinases and 23 transcription factors. Pathway activity computation and mouse survival indicate the NTRK1 mutation induces a higher activation of AKT down- stream targets including MYC and JUN, drives a positive feedback loop to up-regulate multiple other RTKs, and confers higher oncogenic potency than the PDGFRA mutation. A mini-gRNA library CRISPR-Cas9 validation screening shows 56% of tested master regulators are important for the viability of NTRK-driven HGG cells, including TFs (Myc and Jun) and metabolic kinases (AMPKa1 and AMPKa2), confirming the validity of the multiomics inte- grative approaches, and providing novel tumor vulnerabilities. -

DNA Sequencing Genetically Inherited Diseases
DNA, MUTATION DETECTION TYPES OF DNA There are two major types of DNA: Genomic DNA and Mitochondrial DNA Genomic DNA / Nuclear DNA: Comprises the genome of an organism and lead to an expression of genetic traits. Controls expression of the various traits in an organism. Sequenced as part of the Human Genome Project to study the various functions of the different regions of the genome Usually, during DNA replication there is a recombination of genes bringing about a change in sequence leading to individual specific characteristics. This way the difference in sequence could be studied from individual to individual. MITOCHONDRIAL DNA(MT DNA) mtDNA is a double stranded circular molecule. mtDNA is always Maternally inherited. Each Mitochodrion contains about 2-10 mtDNA molecules. mtDNA does not change from parent to offspring (Without recombination) Containing little repetitive DNA, and codes for 37 genes, which include two types of ribosomal RNA, 22 transfer RNAs and 13 protein subunits for some enzymes HUMAN STRUCTURAL GENE 1. Helix–turn–helix 2. Zinc finger 3. Leucine zipper 4. Helix–loop–helix GENE TO PROTEIN Facilitate transport of the mRNA to the cytoplasm and attachment to the ribosome Protect the mRNA from from 5' exonuclease Acts as a buffer to the 3' exonuclease in order to increase the half life of mRNA. TRANSLATION TYPES OF DNA SEQUENCE VARIATION VNTR: Variable Number of Tandem Repeats or minisatellite (Telomeric DNA, Hypervariable minisatellite DNA) ~6-100 bp core unit SSR : Simple Sequence Repeat or STR (short