Identificação De Genes E O Problema Do Alinhamento Spliced Múltiplo
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Atlas Journal
Atlas of Genetics and Cytogenetics in Oncology and Haematology Home Genes Leukemias Tumors Cancer prone Deep Insight Case Reports Portal Journals Teaching X Y 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 NA Atlas Journal Atlas Journal versus Atlas Database: the accumulation of the issues of the Journal constitutes the body of the Database/Text-Book. TABLE OF CONTENTS Volume 13, Number 7, July 2009 Previous Issue / Next Issue Genes ABL1 (v-abl Abelson murine leukemia viral oncogene homolog 1) (9q34.1) - updated. Ali G Turhan. Atlas Genet Cytogenet Oncol Haematol 2009; 13 (7): 757-766. [Full Text] [PDF] URL : http://atlasgeneticsoncology.org/Genes/ABL.html BCL2L12 (BCL2-like 12 (proline-rich)) (19q13.3). Christos Kontos, Hellinida Thomadaki, Andreas Scorilas. Atlas Genet Cytogenet Oncol Haematol 2009; 13 (7): 767-771. [Full Text] [PDF] URL : http://atlasgeneticsoncology.org/Genes/BCL2L12ID773ch19q13.html BCR (Breakpoint cluster region) (22q11.2) - updated. Ali G Turhan. Atlas Genet Cytogenet Oncol Haematol 2009; 13 (7): 772-779. [Full Text] [PDF] URL : http://atlasgeneticsoncology.org/Genes/BCR.html ENAH (enabled homolog (Drosophila)) (1q42.12). Paola Nisticò, Francesca Di Modugno. Atlas Genet Cytogenet Oncol Haematol 2009; 13 (7): 780-785. [Full Text] [PDF] URL : http://atlasgeneticsoncology.org/Genes/ENAHID44148ch1q42.html FGFR2 (fibroblast growth factor receptor 2) (10q26.13). Masaru Katoh. Atlas Genet Cytogenet Oncol Haematol 2009; 13 (7): 786-799. [Full Text] [PDF] URL : http://atlasgeneticsoncology.org/Genes/FGFR2ID40570ch10q26.html MAPK6 (mitogen-activated protein kinase 6) (15q21.2). Sylvain Meloche. Atlas Genet Cytogenet Oncol Haematol 2009; 13 (7): 800-804. -

Human Social Genomics in the Multi-Ethnic Study of Atherosclerosis
Getting “Under the Skin”: Human Social Genomics in the Multi-Ethnic Study of Atherosclerosis by Kristen Monét Brown A dissertation submitted in partial fulfillment of the requirements for the degree of Doctor of Philosophy (Epidemiological Science) in the University of Michigan 2017 Doctoral Committee: Professor Ana V. Diez-Roux, Co-Chair, Drexel University Professor Sharon R. Kardia, Co-Chair Professor Bhramar Mukherjee Assistant Professor Belinda Needham Assistant Professor Jennifer A. Smith © Kristen Monét Brown, 2017 [email protected] ORCID iD: 0000-0002-9955-0568 Dedication I dedicate this dissertation to my grandmother, Gertrude Delores Hampton. Nanny, no one wanted to see me become “Dr. Brown” more than you. I know that you are standing over the bannister of heaven smiling and beaming with pride. I love you more than my words could ever fully express. ii Acknowledgements First, I give honor to God, who is the head of my life. Truly, without Him, none of this would be possible. Countless times throughout this doctoral journey I have relied my favorite scripture, “And we know that all things work together for good, to them that love God, to them who are called according to His purpose (Romans 8:28).” Secondly, I acknowledge my parents, James and Marilyn Brown. From an early age, you two instilled in me the value of education and have been my biggest cheerleaders throughout my entire life. I thank you for your unconditional love, encouragement, sacrifices, and support. I would not be here today without you. I truly thank God that out of the all of the people in the world that He could have chosen to be my parents, that He chose the two of you. -

Downloaded, Each with Over 20 Samples for AD-Specific Pathways, Biological Processes, and Driver Each Specific Brain Region in Each Condition
Xiang et al. BMC Medical Genomics 2018, 11(Suppl 6):115 https://doi.org/10.1186/s12920-018-0431-1 RESEARCH Open Access Condition-specific gene co-expression network mining identifies key pathways and regulators in the brain tissue of Alzheimer’s disease patients Shunian Xiang1,2, Zhi Huang4, Wang Tianfu1, Zhi Han3, Christina Y. Yu3,5, Dong Ni1*, Kun Huang3* and Jie Zhang2* From 29th International Conference on Genome Informatics Yunnan, China. 3-5 December 2018 Abstract Background: Gene co-expression network (GCN) mining is a systematic approach to efficiently identify novel disease pathways, predict novel gene functions and search for potential disease biomarkers. However, few studies have systematically identified GCNs in multiple brain transcriptomic data of Alzheimer’s disease (AD) patients and looked for their specific functions. Methods: In this study, we first mined GCN modules from AD and normal brain samples in multiple datasets respectively; then identified gene modules that are specific to AD or normal samples; lastly, condition-specific modules with similar functional enrichments were merged and enriched differentially expressed upstream transcription factors were further examined for the AD/normal-specific modules. Results: We obtained 30 AD-specific modules which showed gain of correlation in AD samples and 31 normal- specific modules with loss of correlation in AD samples compared to normal ones, using the network mining tool lmQCM. Functional and pathway enrichment analysis not only confirmed known gene functional categories related to AD, but also identified novel regulatory factors and pathways. Remarkably, pathway analysis suggested that a variety of viral, bacteria, and parasitic infection pathways are activated in AD samples. -

Genomic Rearrangements Near Genes Leading to Upregulation Across a Diverse Subset of Human Cancers
bioRxiv preprint doi: https://doi.org/10.1101/099861; this version posted January 12, 2017. The copyright holder for this preprint (which was not certified by peer review) is the author/funder. All rights reserved. No reuse allowed without permission. Genomic rearrangements near genes leading to upregulation across a diverse subset of human cancers Yiqun Zhang1, Fengju Chen1, Chad J. Creighton1,2,3,4 1. Dan L. Duncan Comprehensive Cancer Center, Baylor College of Medicine, Houston, TX 77030, USA. 2. Department of Bioinformatics and Computational Biology, The University of Texas MD Anderson Cancer Center, Houston, TX 77030, USA. 3. Department of Medicine, Baylor College of Medicine, Houston, TX 77030, USA. 4. Human Genome Sequencing Center, Baylor College of Medicine, Houston, TX 77030, USA. Correspondence to: Chad J. Creighton ([email protected]) Abbreviations: PCAWG, the Pan-Cancer Analysis of Whole Genomes project; SV, Structural Variant; Running header: Genomic rearrangements altering transcription bioRxiv preprint doi: https://doi.org/10.1101/099861; this version posted January 12, 2017. The copyright holder for this preprint (which was not certified by peer review) is the author/funder. All rights reserved. No reuse allowed without permission. Abstract Using a dataset of somatic Structural Variants (SVs) in cancers from 2658 patients—1220 with corresponding gene expression data—we identified hundreds of genes for which the nearby presence (within 100kb) of an SV breakpoint was associated with altered expression. For the vast majority of these genes, expression was increased rather than decreased with corresponding SV event. Well-known up-regulated cancer-associated genes impacted by this phenomenon included TERT, MDM2, CDK4, ERBB2, and IGF2. -

Agricultural University of Athens
ΓΕΩΠΟΝΙΚΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΑΘΗΝΩΝ ΣΧΟΛΗ ΕΠΙΣΤΗΜΩΝ ΤΩΝ ΖΩΩΝ ΤΜΗΜΑ ΕΠΙΣΤΗΜΗΣ ΖΩΙΚΗΣ ΠΑΡΑΓΩΓΗΣ ΕΡΓΑΣΤΗΡΙΟ ΓΕΝΙΚΗΣ ΚΑΙ ΕΙΔΙΚΗΣ ΖΩΟΤΕΧΝΙΑΣ ΔΙΔΑΚΤΟΡΙΚΗ ΔΙΑΤΡΙΒΗ Εντοπισμός γονιδιωματικών περιοχών και δικτύων γονιδίων που επηρεάζουν παραγωγικές και αναπαραγωγικές ιδιότητες σε πληθυσμούς κρεοπαραγωγικών ορνιθίων ΕΙΡΗΝΗ Κ. ΤΑΡΣΑΝΗ ΕΠΙΒΛΕΠΩΝ ΚΑΘΗΓΗΤΗΣ: ΑΝΤΩΝΙΟΣ ΚΟΜΙΝΑΚΗΣ ΑΘΗΝΑ 2020 ΔΙΔΑΚΤΟΡΙΚΗ ΔΙΑΤΡΙΒΗ Εντοπισμός γονιδιωματικών περιοχών και δικτύων γονιδίων που επηρεάζουν παραγωγικές και αναπαραγωγικές ιδιότητες σε πληθυσμούς κρεοπαραγωγικών ορνιθίων Genome-wide association analysis and gene network analysis for (re)production traits in commercial broilers ΕΙΡΗΝΗ Κ. ΤΑΡΣΑΝΗ ΕΠΙΒΛΕΠΩΝ ΚΑΘΗΓΗΤΗΣ: ΑΝΤΩΝΙΟΣ ΚΟΜΙΝΑΚΗΣ Τριμελής Επιτροπή: Aντώνιος Κομινάκης (Αν. Καθ. ΓΠΑ) Ανδρέας Κράνης (Eρευν. B, Παν. Εδιμβούργου) Αριάδνη Χάγερ (Επ. Καθ. ΓΠΑ) Επταμελής εξεταστική επιτροπή: Aντώνιος Κομινάκης (Αν. Καθ. ΓΠΑ) Ανδρέας Κράνης (Eρευν. B, Παν. Εδιμβούργου) Αριάδνη Χάγερ (Επ. Καθ. ΓΠΑ) Πηνελόπη Μπεμπέλη (Καθ. ΓΠΑ) Δημήτριος Βλαχάκης (Επ. Καθ. ΓΠΑ) Ευάγγελος Ζωίδης (Επ.Καθ. ΓΠΑ) Γεώργιος Θεοδώρου (Επ.Καθ. ΓΠΑ) 2 Εντοπισμός γονιδιωματικών περιοχών και δικτύων γονιδίων που επηρεάζουν παραγωγικές και αναπαραγωγικές ιδιότητες σε πληθυσμούς κρεοπαραγωγικών ορνιθίων Περίληψη Σκοπός της παρούσας διδακτορικής διατριβής ήταν ο εντοπισμός γενετικών δεικτών και υποψηφίων γονιδίων που εμπλέκονται στο γενετικό έλεγχο δύο τυπικών πολυγονιδιακών ιδιοτήτων σε κρεοπαραγωγικά ορνίθια. Μία ιδιότητα σχετίζεται με την ανάπτυξη (σωματικό βάρος στις 35 ημέρες, ΣΒ) και η άλλη με την αναπαραγωγική -

Genetic Technologies for Building a Better Mouse
Downloaded from genesdev.cshlp.org on September 29, 2021 - Published by Cold Spring Harbor Laboratory Press REVIEW A most formidable arsenal: genetic technologies for building a better mouse James F. Clark,1 Colin J. Dinsmore,1 and Philippe Soriano Department of Cell, Developmental, and Regenerative Biology, Icahn School of Medicine at Mt. Sinai, New York, New York 10029, USA The mouse is one of the most widely used model organ- scribing Mendelian inheritance for mouse coat color char- isms for genetic study. The tools available to alter the acteristics (Cuénot 1902). Mendel himself had started mouse genome have developed over the preceding decades down this same road fifty years earlier, only for his bishop from forward screens to gene targeting in stem cells to the to snuff out the nascent breeding program because a monk recent influx of CRISPR approaches. In this review, we had no business, frankly, breeding (Paigen 2003). first consider the history of mice in genetic study, the Following Cuénot’s papers (Cuénot 1902), genetic re- development of classic approaches to genome modifica- search using the mouse began in earnest, often through tion, and how such approaches have been used and im- the study of spontaneous mouse mutants. One line of re- proved in recent years. We then turn to the recent surge search further explored the complicated genetics of pig- of nuclease-mediated techniques and how they are chang- mentation and the sometimes-surprising coincident ing the field of mouse genetics. Finally, we survey com- phenotypes. The Dominant white spotting allele W (an al- mon classes of alleles used in mice and discuss how lele of the receptor tyrosine kinase Kit) was found to affect they might be engineered using different methods. -

Parent-Of-Origin-Specific Allelic Associations Among 106 Genomic Loci for Age at Menarche
This is a repository copy of Parent-of-origin-specific allelic associations among 106 genomic loci for age at menarche. White Rose Research Online URL for this paper: http://eprints.whiterose.ac.uk/107834/ Version: Accepted Version Article: Perry, JRB, Day, F, Elks, CE et al. (200 more authors) (2014) Parent-of-origin-specific allelic associations among 106 genomic loci for age at menarche. Nature, 514 (7520). 92-+. ISSN 0028-0836 https://doi.org/10.1038/nature13545 Reuse Unless indicated otherwise, fulltext items are protected by copyright with all rights reserved. The copyright exception in section 29 of the Copyright, Designs and Patents Act 1988 allows the making of a single copy solely for the purpose of non-commercial research or private study within the limits of fair dealing. The publisher or other rights-holder may allow further reproduction and re-use of this version - refer to the White Rose Research Online record for this item. Where records identify the publisher as the copyright holder, users can verify any specific terms of use on the publisher’s website. Takedown If you consider content in White Rose Research Online to be in breach of UK law, please notify us by emailing [email protected] including the URL of the record and the reason for the withdrawal request. [email protected] https://eprints.whiterose.ac.uk/ Europe PMC Funders Group Author Manuscript Nature. Author manuscript; available in PMC 2015 April 02. Published in final edited form as: Nature. 2014 October 2; 514(7520): 92– 97. doi:10.1038/nature13545. Europe PMC Funders Author Manuscripts Parent-of-origin specific allelic associations among 106 genomic loci for age at menarche A full list of authors and affiliations appears at the end of the article. -
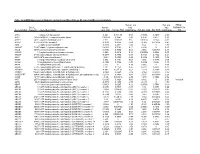
Mrna Expression in Human Leiomyoma and Eker Rats As Measured by Microarray Analysis
Table 3S: mRNA Expression in Human Leiomyoma and Eker Rats as Measured by Microarray Analysis Human_avg Rat_avg_ PENG_ Entrez. Human_ log2_ log2_ RAPAMYCIN Gene.Symbol Gene.ID Gene Description avg_tstat Human_FDR foldChange Rat_avg_tstat Rat_FDR foldChange _DN A1BG 1 alpha-1-B glycoprotein 4.982 9.52E-05 0.68 -0.8346 0.4639 -0.38 A1CF 29974 APOBEC1 complementation factor -0.08024 0.9541 -0.02 0.9141 0.421 0.10 A2BP1 54715 ataxin 2-binding protein 1 2.811 0.01093 0.65 0.07114 0.954 -0.01 A2LD1 87769 AIG2-like domain 1 -0.3033 0.8056 -0.09 -3.365 0.005704 -0.42 A2M 2 alpha-2-macroglobulin -0.8113 0.4691 -0.03 6.02 0 1.75 A4GALT 53947 alpha 1,4-galactosyltransferase 0.4383 0.7128 0.11 6.304 0 2.30 AACS 65985 acetoacetyl-CoA synthetase 0.3595 0.7664 0.03 3.534 0.00388 0.38 AADAC 13 arylacetamide deacetylase (esterase) 0.569 0.6216 0.16 0.005588 0.9968 0.00 AADAT 51166 aminoadipate aminotransferase -0.9577 0.3876 -0.11 0.8123 0.4752 0.24 AAK1 22848 AP2 associated kinase 1 -1.261 0.2505 -0.25 0.8232 0.4689 0.12 AAMP 14 angio-associated, migratory cell protein 0.873 0.4351 0.07 1.656 0.1476 0.06 AANAT 15 arylalkylamine N-acetyltransferase -0.3998 0.7394 -0.08 0.8486 0.456 0.18 AARS 16 alanyl-tRNA synthetase 5.517 0 0.34 8.616 0 0.69 AARS2 57505 alanyl-tRNA synthetase 2, mitochondrial (putative) 1.701 0.1158 0.35 0.5011 0.6622 0.07 AARSD1 80755 alanyl-tRNA synthetase domain containing 1 4.403 9.52E-05 0.52 1.279 0.2609 0.13 AASDH 132949 aminoadipate-semialdehyde dehydrogenase -0.8921 0.4247 -0.12 -2.564 0.02993 -0.32 AASDHPPT 60496 aminoadipate-semialdehyde -
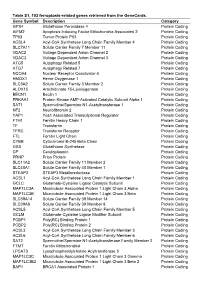
Table S1. 103 Ferroptosis-Related Genes Retrieved from the Genecards
Table S1. 103 ferroptosis-related genes retrieved from the GeneCards. Gene Symbol Description Category GPX4 Glutathione Peroxidase 4 Protein Coding AIFM2 Apoptosis Inducing Factor Mitochondria Associated 2 Protein Coding TP53 Tumor Protein P53 Protein Coding ACSL4 Acyl-CoA Synthetase Long Chain Family Member 4 Protein Coding SLC7A11 Solute Carrier Family 7 Member 11 Protein Coding VDAC2 Voltage Dependent Anion Channel 2 Protein Coding VDAC3 Voltage Dependent Anion Channel 3 Protein Coding ATG5 Autophagy Related 5 Protein Coding ATG7 Autophagy Related 7 Protein Coding NCOA4 Nuclear Receptor Coactivator 4 Protein Coding HMOX1 Heme Oxygenase 1 Protein Coding SLC3A2 Solute Carrier Family 3 Member 2 Protein Coding ALOX15 Arachidonate 15-Lipoxygenase Protein Coding BECN1 Beclin 1 Protein Coding PRKAA1 Protein Kinase AMP-Activated Catalytic Subunit Alpha 1 Protein Coding SAT1 Spermidine/Spermine N1-Acetyltransferase 1 Protein Coding NF2 Neurofibromin 2 Protein Coding YAP1 Yes1 Associated Transcriptional Regulator Protein Coding FTH1 Ferritin Heavy Chain 1 Protein Coding TF Transferrin Protein Coding TFRC Transferrin Receptor Protein Coding FTL Ferritin Light Chain Protein Coding CYBB Cytochrome B-245 Beta Chain Protein Coding GSS Glutathione Synthetase Protein Coding CP Ceruloplasmin Protein Coding PRNP Prion Protein Protein Coding SLC11A2 Solute Carrier Family 11 Member 2 Protein Coding SLC40A1 Solute Carrier Family 40 Member 1 Protein Coding STEAP3 STEAP3 Metalloreductase Protein Coding ACSL1 Acyl-CoA Synthetase Long Chain Family Member 1 Protein -

Programmed DNA Elimination of Germline Development Genes in Songbirds
bioRxiv preprint doi: https://doi.org/10.1101/444364; this version posted December 22, 2018. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY 4.0 International license. 1 Programmed DNA elimination of germline development genes in songbirds 2 3 Cormac M. Kinsella1,2*, Francisco J. Ruiz-Ruano3*, Anne-Marie Dion-Côté1,4, Alexander J. 4 Charles5, Toni I. Gossmann5, Josefa Cabrero3, Dennis Kappei6, Nicola Hemmings5, Mirre J. P. 5 Simons5, Juan P. M. Camacho3, Wolfgang Forstmeier7, Alexander Suh1 6 7 1Department of Evolutionary Biology, Evolutionary Biology Centre (EBC), Science for Life 8 Laboratory, Uppsala University, SE-752 36, Uppsala, Sweden. 9 2Laboratory of Experimental Virology, Department of Medical Microbiology, Amsterdam UMC, 10 University of Amsterdam, 1105 AZ, Amsterdam, The Netherlands. 11 3Department of Genetics, University of Granada, E-18071, Granada, Spain. 12 4Department of Molecular Biology & Genetics, Cornell University, NY 14853, Ithaca, United 13 States. 14 5Department of Animal and Plant Sciences, University of Sheffield, S10 2TN, Sheffield, United 15 Kingdom. 16 6Cancer Science Institute of Singapore, National University of Singapore, 117599, Singapore. 17 7Max Planck Institute for Ornithology, D-82319, Seewiesen, Germany. 18 19 *These authors contributed equally to this work (alphabetical order). 20 21 Correspondence and requests for materials should be addressed to F.J.R.R. (email: 22 [email protected]) and A.S. (email: [email protected]). 23 1 bioRxiv preprint doi: https://doi.org/10.1101/444364; this version posted December 22, 2018. -
Linkage Analysis and a QTL Study of Sexual Compatibility Factors and Floral Traits
Copyright 0 1997 by the Genetics Society of America An Interspecific Backcross of Lycopersicon esculentum X L. hirsutum: Linkage Analysis and a QTL Study of Sexual Compatibility Factors and Floral Traits Dario Bernacchi and Steven D. Tanksley Department of Plant Breeding and Biometry, Cornell University, Ithaca, New York, 14850 Manuscript received February 7, 1997 Accepted for publication July 10, 1997 ABSTRACT A BC, population of the self-compatible tomato Lycopersicon escuhtum and its wild self-incompatible relative L. hirsutum f. typicum was used for restriction fragment length polymorphism linkage analysis and quantitative trait loci (QTL) mappingof reproductive behavior and floral traits. The self-incompati- bility locus, S, on chromosome 1 harbored the only QTL for self-incompatibility indicating that the transition to self-compatibility in the lineage leading to the cultivated tomato was primarily the result of mutations at the S locus. Moreover, the major QTL controlling unilateral incongruityalso mapped to the S locus, supporting the hypothesis that self-incompatibility and unilateral incongruity are not independent mechanisms. The mating behaviorof near-isogenic lines carrying the L. hirsutum allele for the S locus on chromosome 1 in an otherwise L. esculentum background support these conclusions. The S locus region of chromosome 1 also harbors most major QTL for several floral traits important to pollination biology (e.g., number and sizeof flowers), suggesting a gene complex controlling both genetic and morphological mechanisms of reproduction control. Similar associationsin other flowering plants suggest~- that such complex may have been conserved since early periods of plant evolution or else reflect a convergent evolutionary process. N angiosperms numerous mechanisms are known to and FRANKLIN1992). -

Network-Based Stratification of Tumor Mutations
ARTICLES OPEN Network-based stratification of tumor mutations Matan Hofree1, John P Shen2, Hannah Carter2, Andrew Gross3 & Trey Ideker1–3 Many forms of cancer have multiple subtypes with different small-cell lung cancer, subtypes derived from expression profiles do causes and clinical outcomes. Somatic tumor genome sequences not correlate with any clinical phenotype including patient survival provide a rich new source of data for uncovering these and response to chemotherapy2,10. These results might be due to subtypes but have proven difficult to compare, as two tumors limitations of expression-based analysis11 such as issues with RNA rarely share the same mutations. Here we introduce network- sample quality, lack of reproducibility between biological replicates based stratification (NBS), a method to integrate somatic and ample opportunities for overfitting of data. tumor genomes with gene networks. This approach allows for A promising new source of data for tumor stratification is the stratification of cancer into informative subtypes by clustering somatic mutation profile, in which high-throughput sequencing together patients with mutations in similar network regions. is used to compare the genome or exome of a patient’s tumor We demonstrate NBS in ovarian, uterine and lung cancer cohorts to that of the germ line to identify mutations that have become from The Cancer Genome Atlas. For each tissue, NBS identifies enriched in the tumor cell population12. As this set of mutations subtypes that are predictive of clinical outcomes such as is presumed to contain the causal drivers of tumor progression13, patient survival, response to therapy or tumor histology. We similarities and differences in mutations across patients could identify network regions characteristic of each subtype and provide invaluable information for stratification.