Dissertacao Simone Cristina Girardi.Pdf
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

§4-71-6.5 LIST of CONDITIONALLY APPROVED ANIMALS November
§4-71-6.5 LIST OF CONDITIONALLY APPROVED ANIMALS November 28, 2006 SCIENTIFIC NAME COMMON NAME INVERTEBRATES PHYLUM Annelida CLASS Oligochaeta ORDER Plesiopora FAMILY Tubificidae Tubifex (all species in genus) worm, tubifex PHYLUM Arthropoda CLASS Crustacea ORDER Anostraca FAMILY Artemiidae Artemia (all species in genus) shrimp, brine ORDER Cladocera FAMILY Daphnidae Daphnia (all species in genus) flea, water ORDER Decapoda FAMILY Atelecyclidae Erimacrus isenbeckii crab, horsehair FAMILY Cancridae Cancer antennarius crab, California rock Cancer anthonyi crab, yellowstone Cancer borealis crab, Jonah Cancer magister crab, dungeness Cancer productus crab, rock (red) FAMILY Geryonidae Geryon affinis crab, golden FAMILY Lithodidae Paralithodes camtschatica crab, Alaskan king FAMILY Majidae Chionocetes bairdi crab, snow Chionocetes opilio crab, snow 1 CONDITIONAL ANIMAL LIST §4-71-6.5 SCIENTIFIC NAME COMMON NAME Chionocetes tanneri crab, snow FAMILY Nephropidae Homarus (all species in genus) lobster, true FAMILY Palaemonidae Macrobrachium lar shrimp, freshwater Macrobrachium rosenbergi prawn, giant long-legged FAMILY Palinuridae Jasus (all species in genus) crayfish, saltwater; lobster Panulirus argus lobster, Atlantic spiny Panulirus longipes femoristriga crayfish, saltwater Panulirus pencillatus lobster, spiny FAMILY Portunidae Callinectes sapidus crab, blue Scylla serrata crab, Samoan; serrate, swimming FAMILY Raninidae Ranina ranina crab, spanner; red frog, Hawaiian CLASS Insecta ORDER Coleoptera FAMILY Tenebrionidae Tenebrio molitor mealworm, -

Interindividual Size Heteromorphism of NOR and Chromosomal Location of 5S Rrna Genes in Iheringichthys Labrosus
141 Vol.50, n. 1 : pp.141-146, January 2007 ISSN 1516-8913 Printed in Brazil BRAZILIAN ARCHIVES OF BIOLOGY AND TECHNOLOGY AN INTERNATIONAL JOURNAL Interindividual Size Heteromorphism of NOR and Chromosomal Location of 5S rRNA Genes in Iheringichthys labrosus Rafael Augusto de Carvalho and Ana Lúcia Dias * Universidade Estadual de Londrina; Departamento de Biologia Geral; CCB; [email protected]; C. P. 6001; 86051-970; Londrina - PR - Brasil ABSTRACT Twenty-five specimens of Iheringichthys labrosus from the Capivara Reservoir were analysed cytogenetically. AgNORs were detected in a pair of ST chromosomes, in the telomeric region of the long arm. Some individuals showed size heteromorphism of this region between homologous chromosomes. Treatment with CMA 3 displayed GC-rich regions corresponding to the AgNOR pair, plus other fluorescent staining. In situ hybridization by fluorescence (FISH) with the 18S rDNA probe showed only one pair of stained chromosomes, confirming the heteromorphism observed with AgNO 3 and CMA 3 in some individuals. The 5S rDNA probe revealed telomeric staining on the long arm of a pair of chromosomes of the ST-A group, probably different from the NOR pair. Key words : F ish chromosome, AgNOR, in situ hibridization, chromomycin A 3, heteromorphism INTRODUCTION used in chromosome preparations of fish to demonstrate the location of NORs, because Ribosomal cistrons are organized in multiple according to Salvadori et al . (1995), these regions copies that are repeated in tandem throughout the are associated with DNA families rich in GC cellular genome. Two different arrangements are bases. More recently, FISH with rDNA probes has known: one that contains 45S ribosomal DNA been utilized with precision to determine the exact (rDNA) which codes for 28S, 5.8S and 18S location and number of ribosomal cístrons. -

Iheringichthys Labrosus, Pimelodidae)
Neotropical Ichthyology, 9(4): 889-894, 2011 Copyright © 2011 Sociedade Brasileira de Ictiologia Sound production and pectoral spine locking in a Neotropical catfish (Iheringichthys labrosus, Pimelodidae) Javier S. Tellechea1, Franco Teixeira-de Mello2,3, Iván Gonzalez-Bergonzoni2,3 and Nicolás Vidal2 Catfishes may have two sonic organs: pectoral spines for stridulation and swimbladder drumming muscles. The aim of this study was to characterize the sound production of the catfish Iheringichthys labrosus. The I. labrosus male and female emits two different types of sounds: stridulatory sounds (655.8 + 230 Hz) consisting of a train of pulses, and drumming sounds (220 + 46 Hz), which are composed of single-pulse harmonic signals. Stridulatory sounds are emitted during abduction of the pectoral spine. At the base of the spine there is a dorsal process that bears a series of ridges on its latero-ventral surface, and by pressing the ridges against the groove (with an unspecialized rough surface) during a fin sweep, the animal produce a series of short pulses. Drumming sound is produced by an extrinsic sonic muscle, originated on a flat tendon of the transverse process of the fourth vertebra and inserted on the rostral and ventral surface of the swimbladder. The sounds emitted by both mechanisms are emitted in distress situation. Distress was induced by manipulating fish in a laboratory tank while sounds were recorded. Our results indicate that the catfish initially emits a stridulatory sound, which is followed by a drumming sound. Simultaneous production of stridulatory and drumming sounds was also observed. The catfish drumming sounds were lower in dominant frequency than stridulatory sounds, and also exhibited a small degree of dominant frequency modulation. -

Inland Waters
477 Fish, crustaceans, molluscs, etc Capture production by species items America, South - Inland waters C-03 Poissons, crustacés, mollusques, etc Captures par catégories d'espèces Amérique du Sud - Eaux continentales (a) Peces, crustáceos, moluscos, etc Capturas por categorías de especies América del Sur - Aguas continentales English name Scientific name Species group Nom anglais Nom scientifique Groupe d'espèces 2012 2013 2014 2015 2016 2017 2018 Nombre inglés Nombre científico Grupo de especies t t t t t t t Common carp Cyprinus carpio 11 321 114 134 179 169 46 240 Cyprinids nei Cyprinidae 11 425 429 423 400 400 400 400 ...A Caquetaia kraussii 12 ... ... 11 182 111 559 64 Nile tilapia Oreochromis niloticus 12 5 7 3 6 255 257 159 Tilapias nei Oreochromis (=Tilapia) spp 12 9 133 9 210 9 093 8 690 8 600 8 600 8 600 Oscar Astronotus ocellatus 12 1 847 1 862 1 951 1 941 1 825 1 813 1 815 Velvety cichlids Astronotus spp 12 391 385 318 571 330 345 334 Green terror Aequidens rivulatus 12 26 38 20 24 36 30 34 Cichlids nei Cichlidae 12 13 013 13 123 12 956 12 400 12 403 12 735 12 428 Arapaima Arapaima gigas 13 1 478 1 504 1 484 2 232 1 840 2 441 1 647 Arawana Osteoglossum bicirrhosum 13 1 642 1 656 1 635 1 570 1 571 2 200 2 056 Banded astyanax Astyanax fasciatus 13 1 043 1 052 1 039 1 000 1 000 1 000 1 000 ...A Brycon orbignyanus 13 8 8 8 8 8 9 14 ...A Brycon dentex 13 35 20 5 6 11 10 6 ...A Brycon spp 13 .. -

(Monogenea: Dactylogyridae) and a Redescription of D
Journal of Helminthology (2018) 92, 228–243 doi:10.1017/S0022149X17000256 © Cambridge University Press 2017 Morphology and molecular characterization of Demidospermus spirophallus n. sp., D. prolixus n. sp. (Monogenea: Dactylogyridae) and a redescription of D. anus in siluriform catfish from Brazil L. Franceschini1*, A.C. Zago1, M.I. Müller1, C.J. Francisco1, R.M. Takemoto2 and R.J. da Silva1 1São Paulo State University (Unesp), Institute of Biosciences, Botucatu, Brazil, CEP 18618-689: 2State University of Maringá (UEM), Limnology, Ichthyology and Aquaculture Research Center (Nupélia), Maringá, Brazil, CEP 87020-900 (Received 29 September 2016; Accepted 26 February 2017; First published online 6 April 2017) Abstract The present study describes Demidospermus spirophallus n. sp. and Demidosper- mus prolixus n. sp. (Monogenea, Dactylogyridae) from the siluriform catfish Loricaria prolixa Isbrücker & Nijssen, 1978 (Siluriformes, Loricariidae) from the state of São Paulo, Brazil, supported by morphological and molecular data. In add- ition, notes on the circumscription of the genus with a redescription of Demisdospermus anus are presented. Demidospermus spirophallus n. sp. differed from other congeners mainly because of the morphology of the male copulatory organ (MCO), which exhibited 2½ counterclockwise rings, a tubular accessory piece with one bifurcated end and a weakly sclerotized vagina with sinistral open- ing. Demidospermus prolixus n. sp. presents a counterclockwise-coiled MCO with 1½ rings, an ovate base, a non-articulated groove-like accessory piece serving as an MCO guide, two different hook shapes, inconspicuous tegumental annulations, a non-sclerotized vagina with sinistral opening and the absence of eyes or acces- sory eyespots. The present study provides, for the first time, molecular character- ization data using the partial ribosomal gene (28S) of two new species of Demidospermus from Brazil (D. -

Food Ecology of Hassar Affinis (Actinopterygii: Doradidae)
Research, Society and Development, v. 10, n. 8, e10110816973, 2021 (CC BY 4.0) | ISSN 2525-3409 | DOI: http://dx.doi.org/10.33448/rsd-v10i8.16973 Food ecology of Hassar affinis (Actinopterygii: Doradidae) in two lakes of a wet zone of international importance in Northeast Brazil Ecologia alimentar de Hassar affinis (Actinopterygii: Doradidae) em dois lagos de uma zona úmida de importância internacional no Nordeste do Brasil Ecología alimentaria de Hassar affinis (Actinopterygii: Doradidae) en dos lagos de una zona húmeda de importancia internacional en el Noreste de Brasil Received: 06/08/2021 | Reviewed: 06/16/2021 | Accept: 06/21/2021 | Published: 07/07/2021 Maria Fabiene de Sousa Barros ORCID https://orcid.org/0000-0003-4280-443X Universidade Estadual do Maranhão, Brazil E-mail: [email protected] Zafira da Silva de Almeida ORCID https://orcid.org/0000-0002-8295-5040 Universidade Estadual do Maranhão, Brazil E-mail: [email protected] Marina Bezerra Figueiredo ORCID http://orcid.org/0000-0001-7485-8593 Universidade Estadual do Maranhão, Brazil E-mail: [email protected] Jorge Luiz Silva Nunes ORCID https://orcid.org/0000-0001-6223-1785 Universidade Estadual do Maranhão, Brazil E-mail: [email protected] Raimunda Nonata Fortes Carvalho Neta ORCID http://orcid.org/0000-0002-3519-5237 Universidade Estadual do Maranhão, Brazil E-mail: [email protected] Abstract The study aimed to describe the aspects of trophic ecology and feeding strategy of the Hassar affinis species in two lakes in the Baixada Maranhense region a wetland of international ecological interest (Site Ramsar). Individuals were collected monthly for one year. -

Contributions to the Taxonomy of Trachelyopterus (Siluriformes): Comparative Cytogenetic Analysis in Three Species of Auchenipteridae
Neotropical Ichthyology Original article https://doi.org/10.1590/1982-0224-2020-0115 Contributions to the taxonomy of Trachelyopterus (Siluriformes): comparative cytogenetic analysis in three species of Auchenipteridae 1 1 1 Correspondence: Dayane Petik dos Santos , Denise Felicetti , Lucas Baumgärtner , 1 2 Roberto Laridondo Lui Vladimir Pavan Margarido , Daniel Rodrigues Blanco , [email protected] Orlando Moreira-Filho3 and Roberto Laridondo Lui1 Auchenipteridae is divided into subfamilies Centromochlinae and Auchenipterinae. Parauchenipterus is included in the latter and is subject of taxonomic discussions concerning its validation or synonymization with Trachelyopterus. Herein, three species from two hydrographic basins were cytogenetically analyzed: Parauchenipterus striatulus from Doce River and two sympatric species, P. galeatus and Trachelyopterus coriaceus, from the Araguaia River. Diploid number of 58 chromosomes was verified for all species, but P. striatulus has different karyotype formula from the others. The three species have heterochromatin located in terminal regions of almost all chromosomes and in pericentromeric region on acrocentric chromosomes. Simple NORs was verified on a subtelocentric chromosome for all species. 5S rDNA sites were detected in three submetacentric chromosome pairs in P. striatulus; in a metacentric chromosome pair and submetacentric pair in T. coriaceus; and in one metacentric chromosome pair in P. galeatus. The similarities found in the karyotypes of the three species suggest the existence of only one genus, Trachelyopterus; therefore, Submitted October 15, 2020 our data refutes the validation of Parauchenipterus. Moreover, the differences in 5S Accepted February 10, 2021 rDNA distribution in P. galeatus in comparison with other populations already by Carlos DoNascimiento studied, indicate the existence of a new taxonomic unit, which suggests a species Epub March 31, 2021 complex in P. -

Illegal Hunting and Fishing in Brazil
n a t u r e z a & c o n s e r v a ç ã o 1 3 (2 0 1 5) 183–189 Natureza & Conservação Brazilian Journal of Nature Conservation Supported by Boticário Group Foundation for Nature Protection ht tp://www.naturezaeconservacao.com.br Research Letters Illegal hunting and fishing in Brazil: a study based on data provided by environmental military police Aline Torres de Azevedo Chagas, Maisa Aparecida da Costa, ∗ Ana Paula Vimieiro Martins, Leonardo Cardoso Resende, Evanguedes Kalapothakis Laboratório de Biotecnologia e Marcadores Moleculares, Department of General Biology, Instituto de Ciências Biológicas, Universidade Federal de Minas Gerais, Belo Horizonte, MG, Brazil a r a t i c l e i n f o b s t r a c t Article history: Illegal hunting and fishing activities are of great relevance to conservation policies. Few Received 6 November 2015 studies with regional focus of the impacts of these activities in Brazil are available. The aim Accepted 9 November 2015 of this study was to characterize illegal hunting and fishing on a national level by collecting Available online 2 December 2015 data from the environmental police. We analyzed reports prepared by 16 states, all of them which contained a variety of information about seized species, and showed a lack of stan- Keywords: dardization of data collection and presentation. Illegal fish seizures were predominantly of Amazonian species. Illegal hunting seizures showed the most uniform territorial distribu- Illegal hunting Illegal fishing tion. Armadillos (Dasypodidae family), pacas (Cuniculus paca), and capybaras (Hydrochoerus Environmental police hydrochaeris) were the most frequently seized species, and numerous seizures of Brazilian Species of forensic interest guinea pig (Cavia aperea) were reported in northeastern Brazil. -

A 1 Case Study with Amazonian Fishes
bioRxiv preprint doi: https://doi.org/10.1101/2021.04.18.440157; this version posted April 21, 2021. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY-NC 4.0 International license. 1 The critical role of natural history museums in advancing eDNA for biodiversity studies: a 2 case study with Amazonian fishes 3 4 C. David de Santana1*, Lynne R. Parenti1, Casey B. Dillman2, Jonathan A. Coddington3, D. A. 5 Bastos 4, Carole C. Baldwin1, Jansen Zuanon5, Gislene Torrente-Vilara6, Raphaël Covain7, 6 Naércio A. Menezes8, Aléssio Datovo8, T. Sado9, M. Miya9 7 8 1 Division of Fishes, Department of Vertebrate Zoology, MRC 159, National Museum of 9 Natural History, PO Box 37012, Smithsonian Institution, Washington, DC 20013-7012, USA 10 2 Cornell University Museum of Vertebrates, Department of Ecology and Evolutionary Biology, 11 Cornell University, Ithaca, NY, 14850, USA 12 3 Global Genome Initiative, National Museum of Natural History, PO Box 37012, Smithsonian 13 Institution, Washington, DC 20013-7012, USA 14 4 Programa de PósGraduação em Ciências Biológicas (BADPI), Instituto Nacional de 15 Pesquisas da Amazônia, Manaus, Brazil 16 5 Coordenacão de Biodiversidade, Instituto Nacional de Pesquisas da Amazonia, Manaus, 17 Amazonas, Brazil 18 6 Instituto do Mar, Universidade Federal de São Paulo, Campus Baixada Santista, Santos, São 19 Paulo, Brazil 20 7 Muséum d’histoire naturelle, Département d’herpétologie et d’ichtyologie, route de Malagnou 21 1, case postale 6434, CH-1211, Genève 6, Switzerland 22 8 Museu de Zoologia da Universidade de São Paulo (MZUSP), Av. -

Redalyc.Checklist of the Freshwater Fishes of Colombia
Biota Colombiana ISSN: 0124-5376 [email protected] Instituto de Investigación de Recursos Biológicos "Alexander von Humboldt" Colombia Maldonado-Ocampo, Javier A.; Vari, Richard P.; Saulo Usma, José Checklist of the Freshwater Fishes of Colombia Biota Colombiana, vol. 9, núm. 2, 2008, pp. 143-237 Instituto de Investigación de Recursos Biológicos "Alexander von Humboldt" Bogotá, Colombia Available in: http://www.redalyc.org/articulo.oa?id=49120960001 How to cite Complete issue Scientific Information System More information about this article Network of Scientific Journals from Latin America, the Caribbean, Spain and Portugal Journal's homepage in redalyc.org Non-profit academic project, developed under the open access initiative Biota Colombiana 9 (2) 143 - 237, 2008 Checklist of the Freshwater Fishes of Colombia Javier A. Maldonado-Ocampo1; Richard P. Vari2; José Saulo Usma3 1 Investigador Asociado, curador encargado colección de peces de agua dulce, Instituto de Investigación de Recursos Biológicos Alexander von Humboldt. Claustro de San Agustín, Villa de Leyva, Boyacá, Colombia. Dirección actual: Universidade Federal do Rio de Janeiro, Museu Nacional, Departamento de Vertebrados, Quinta da Boa Vista, 20940- 040 Rio de Janeiro, RJ, Brasil. [email protected] 2 Division of Fishes, Department of Vertebrate Zoology, MRC--159, National Museum of Natural History, PO Box 37012, Smithsonian Institution, Washington, D.C. 20013—7012. [email protected] 3 Coordinador Programa Ecosistemas de Agua Dulce WWF Colombia. Calle 61 No 3 A 26, Bogotá D.C., Colombia. [email protected] Abstract Data derived from the literature supplemented by examination of specimens in collections show that 1435 species of native fishes live in the freshwaters of Colombia. -
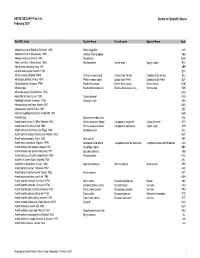
ASFIS ISSCAAP Fish List February 2007 Sorted on Scientific Name
ASFIS ISSCAAP Fish List Sorted on Scientific Name February 2007 Scientific name English Name French name Spanish Name Code Abalistes stellaris (Bloch & Schneider 1801) Starry triggerfish AJS Abbottina rivularis (Basilewsky 1855) Chinese false gudgeon ABB Ablabys binotatus (Peters 1855) Redskinfish ABW Ablennes hians (Valenciennes 1846) Flat needlefish Orphie plate Agujón sable BAF Aborichthys elongatus Hora 1921 ABE Abralia andamanika Goodrich 1898 BLK Abralia veranyi (Rüppell 1844) Verany's enope squid Encornet de Verany Enoploluria de Verany BLJ Abraliopsis pfefferi (Verany 1837) Pfeffer's enope squid Encornet de Pfeffer Enoploluria de Pfeffer BJF Abramis brama (Linnaeus 1758) Freshwater bream Brème d'eau douce Brema común FBM Abramis spp Freshwater breams nei Brèmes d'eau douce nca Bremas nep FBR Abramites eques (Steindachner 1878) ABQ Abudefduf luridus (Cuvier 1830) Canary damsel AUU Abudefduf saxatilis (Linnaeus 1758) Sergeant-major ABU Abyssobrotula galatheae Nielsen 1977 OAG Abyssocottus elochini Taliev 1955 AEZ Abythites lepidogenys (Smith & Radcliffe 1913) AHD Acanella spp Branched bamboo coral KQL Acanthacaris caeca (A. Milne Edwards 1881) Atlantic deep-sea lobster Langoustine arganelle Cigala de fondo NTK Acanthacaris tenuimana Bate 1888 Prickly deep-sea lobster Langoustine spinuleuse Cigala raspa NHI Acanthalburnus microlepis (De Filippi 1861) Blackbrow bleak AHL Acanthaphritis barbata (Okamura & Kishida 1963) NHT Acantharchus pomotis (Baird 1855) Mud sunfish AKP Acanthaxius caespitosa (Squires 1979) Deepwater mud lobster Langouste -

The Living Planet Index (Lpi) for Migratory Freshwater Fish Technical Report
THE LIVING PLANET INDEX (LPI) FOR MIGRATORY FRESHWATER FISH LIVING PLANET INDEX TECHNICAL1 REPORT LIVING PLANET INDEXTECHNICAL REPORT ACKNOWLEDGEMENTS We are very grateful to a number of individuals and organisations who have worked with the LPD and/or shared their data. A full list of all partners and collaborators can be found on the LPI website. 2 INDEX TABLE OF CONTENTS Stefanie Deinet1, Kate Scott-Gatty1, Hannah Rotton1, PREFERRED CITATION 2 1 1 Deinet, S., Scott-Gatty, K., Rotton, H., Twardek, W. M., William M. Twardek , Valentina Marconi , Louise McRae , 5 GLOSSARY Lee J. Baumgartner3, Kerry Brink4, Julie E. Claussen5, Marconi, V., McRae, L., Baumgartner, L. J., Brink, K., Steven J. Cooke2, William Darwall6, Britas Klemens Claussen, J. E., Cooke, S. J., Darwall, W., Eriksson, B. K., Garcia Eriksson7, Carlos Garcia de Leaniz8, Zeb Hogan9, Joshua de Leaniz, C., Hogan, Z., Royte, J., Silva, L. G. M., Thieme, 6 SUMMARY 10 11, 12 13 M. L., Tickner, D., Waldman, J., Wanningen, H., Weyl, O. L. Royte , Luiz G. M. Silva , Michele L. Thieme , David Tickner14, John Waldman15, 16, Herman Wanningen4, Olaf F., Berkhuysen, A. (2020) The Living Planet Index (LPI) for 8 INTRODUCTION L. F. Weyl17, 18 , and Arjan Berkhuysen4 migratory freshwater fish - Technical Report. World Fish Migration Foundation, The Netherlands. 1 Indicators & Assessments Unit, Institute of Zoology, Zoological Society 11 RESULTS AND DISCUSSION of London, United Kingdom Edited by Mark van Heukelum 11 Data set 2 Fish Ecology and Conservation Physiology Laboratory, Department of Design Shapeshifter.nl Biology and Institute of Environmental Science, Carleton University, Drawings Jeroen Helmer 12 Global trend Ottawa, ON, Canada 15 Tropical and temperate zones 3 Institute for Land, Water and Society, Charles Sturt University, Albury, Photography We gratefully acknowledge all of the 17 Regions New South Wales, Australia photographers who gave us permission 20 Migration categories 4 World Fish Migration Foundation, The Netherlands to use their photographic material.