Mechanistic Insights Into the PARP10-Mediated ADP
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

CD56+ T-Cells in Relation to Cytomegalovirus in Healthy Subjects and Kidney Transplant Patients
CD56+ T-cells in Relation to Cytomegalovirus in Healthy Subjects and Kidney Transplant Patients Institute of Infection and Global Health Department of Clinical Infection, Microbiology and Immunology Thesis submitted in accordance with the requirements of the University of Liverpool for the degree of Doctor in Philosophy by Mazen Mohammed Almehmadi December 2014 - 1 - Abstract Human T cells expressing CD56 are capable of tumour cell lysis following activation with interleukin-2 but their role in viral immunity has been less well studied. The work described in this thesis aimed to investigate CD56+ T-cells in relation to cytomegalovirus infection in healthy subjects and kidney transplant patients (KTPs). Proportions of CD56+ T cells were found to be highly significantly increased in healthy cytomegalovirus-seropositive (CMV+) compared to cytomegalovirus-seronegative (CMV-) subjects (8.38% ± 0.33 versus 3.29%± 0.33; P < 0.0001). In donor CMV-/recipient CMV- (D-/R-)- KTPs levels of CD56+ T cells were 1.9% ±0.35 versus 5.42% ±1.01 in D+/R- patients and 5.11% ±0.69 in R+ patients (P 0.0247 and < 0.0001 respectively). CD56+ T cells in both healthy CMV+ subjects and KTPs expressed markers of effector memory- RA T-cells (TEMRA) while in healthy CMV- subjects and D-/R- KTPs the phenotype was predominantly that of naïve T-cells. Other surface markers, CD8, CD4, CD58, CD57, CD94 and NKG2C were expressed by a significantly higher proportion of CD56+ T-cells in healthy CMV+ than CMV- subjects. Functional studies showed levels of pro-inflammatory cytokines IFN-γ and TNF-α, as well as granzyme B and CD107a were significantly higher in CD56+ T-cells from CMV+ than CMV- subjects following stimulation with CMV antigens. -

DNA Damage Sensitization of Breast Cancer Cells with PARP10/ARTD10 Inhibitor Mikko Hukkanen
Pro gradu DNA damage sensitization of breast cancer cells with PARP10/ARTD10 inhibitor Mikko Hukkanen University of Oulu Faculty of Biochemistry and Molecular Medicine 2019 This thesis was completed at the Faculty of Biochemistry and Molecular Medicine, University of Oulu. Oulu, Finland Supervisors: Professor Lari Lehtiö, Dr. Jarkko Koivunen and MSc Sudarshan Murthy Acknowledgements The work of this thesis was made in Faculty of Biochemistry and Molecular Medicine (FBMM) of University of Oulu. Firstly, I would like to thank Professor Lari Lehtiö for the opportunity working in his group, and the advice I received for my thesis. My gratitude is expressed also to my other supervisors, Jarkko Koivunen, for all the guidance I received in the cell culture, and Sudarshan Murthy, for the guidance to express and purify protein, and to conclude IC50 analysis. I would also thank all the personnel in LL group for all the advice I received in laboratory. I would especially thank Sven Sowa who made the Mantis run for IC50 analysis possible. Abbreviations 5-FU – 5-fluorouracil aa – Aminoacid ADP – Adenosine diphosphate ADPr – ADP-ribosylation AEC – Anion-exchange chromatography AIF – Apoptosis inducing factor AIM – Auto-induction medium Arg – Arginine ART – ADP-ribosyltransferase ARTD – Diphtheria toxin –like ADP-ribosyltransferase Asp – Aspartate BRCA – Breast cancer gene BRCT – BRCA1 C terminus CPT – Camptothecin CRC – Colorectal cancer CRM1 – Chromosome region maintenance 1 Cys – Cysteine DDR – DNA damage response dH2O – Distilled water DMEM – Dulbecco’s -

Downloaded Per Proteome Cohort Via the Web- Site Links of Table 1, Also Providing Information on the Deposited Spectral Datasets
www.nature.com/scientificreports OPEN Assessment of a complete and classifed platelet proteome from genome‑wide transcripts of human platelets and megakaryocytes covering platelet functions Jingnan Huang1,2*, Frauke Swieringa1,2,9, Fiorella A. Solari2,9, Isabella Provenzale1, Luigi Grassi3, Ilaria De Simone1, Constance C. F. M. J. Baaten1,4, Rachel Cavill5, Albert Sickmann2,6,7,9, Mattia Frontini3,8,9 & Johan W. M. Heemskerk1,9* Novel platelet and megakaryocyte transcriptome analysis allows prediction of the full or theoretical proteome of a representative human platelet. Here, we integrated the established platelet proteomes from six cohorts of healthy subjects, encompassing 5.2 k proteins, with two novel genome‑wide transcriptomes (57.8 k mRNAs). For 14.8 k protein‑coding transcripts, we assigned the proteins to 21 UniProt‑based classes, based on their preferential intracellular localization and presumed function. This classifed transcriptome‑proteome profle of platelets revealed: (i) Absence of 37.2 k genome‑ wide transcripts. (ii) High quantitative similarity of platelet and megakaryocyte transcriptomes (R = 0.75) for 14.8 k protein‑coding genes, but not for 3.8 k RNA genes or 1.9 k pseudogenes (R = 0.43–0.54), suggesting redistribution of mRNAs upon platelet shedding from megakaryocytes. (iii) Copy numbers of 3.5 k proteins that were restricted in size by the corresponding transcript levels (iv) Near complete coverage of identifed proteins in the relevant transcriptome (log2fpkm > 0.20) except for plasma‑derived secretory proteins, pointing to adhesion and uptake of such proteins. (v) Underrepresentation in the identifed proteome of nuclear‑related, membrane and signaling proteins, as well proteins with low‑level transcripts. -

Mouse Parp10 Knockout Project (CRISPR/Cas9)
https://www.alphaknockout.com Mouse Parp10 Knockout Project (CRISPR/Cas9) Objective: To create a Parp10 knockout Mouse model (C57BL/6J) by CRISPR/Cas-mediated genome engineering. Strategy summary: The Parp10 gene (NCBI Reference Sequence: NM_001163576 ; Ensembl: ENSMUSG00000063268 ) is located on Mouse chromosome 15. 10 exons are identified, with the ATG start codon in exon 1 and the TGA stop codon in exon 10 (Transcript: ENSMUST00000165738). Exon 1~10 will be selected as target site. Cas9 and gRNA will be co-injected into fertilized eggs for KO Mouse production. The pups will be genotyped by PCR followed by sequencing analysis. Note: Exon 1 starts from about 0.03% of the coding region. Exon 1~10 covers 97.47% of the coding region. The size of effective KO region: ~9856 bp. The KO region does not have any other known gene. Page 1 of 9 https://www.alphaknockout.com Overview of the Targeting Strategy Wildtype allele 5' gRNA region gRNA region 3' 1 2 3 4 5 6 7 8 9 10 Legends Exon of mouse Parp10 Knockout region Page 2 of 9 https://www.alphaknockout.com Overview of the Dot Plot (up) Window size: 15 bp Forward Reverse Complement Sequence 12 Note: The 2000 bp section upstream of start codon is aligned with itself to determine if there are tandem repeats. Tandem repeats are found in the dot plot matrix. The gRNA site is selected outside of these tandem repeats. Overview of the Dot Plot (down) Window size: 15 bp Forward Reverse Complement Sequence 12 Note: The 2000 bp section downstream of Exon 10 is aligned with itself to determine if there are tandem repeats. -

Development of Novel Tools to Study PARP10-Mediated ADP-Ribosylation Reactions
Development of novel tools to study PARP10-mediated ADP-ribosylation reactions Von der Medizinischen Fakultät der Rheinisch-Westfälischen Technischen Hochschule Aachen zur Erlangung des akademischen Grades einer Doktorin der Medizin genehmigte Dissertation vorgelegt von Anne Kristin Braczynski aus Erkelenz Berichter: Herr Universitätsprofessor Dr.rer.nat. Bernhard Lüscher Frau Professorin Dr.rer.nat. Antje Ostareck-Lederer Tag der mündlichen Prüfung: 14. Juni 2012 Diese Dissertation ist auf den Internetseiten der Hochschulbibliothek online verfügbar. Abstract PARP10 belongs to the poly-ADP-ribose polymerase family of ADP-ribosylating en- zymes and was originally found to interact with MYC. ADP-ribosylation controls many cellular processes, including transcription, DNA repair, and bacterial toxic- ity. It has been shown recently that PARP10 differs from the bona fide PARP en- zymes because it lacks polymerase activity and instead functions as mono-ADP- ribosyltransferase. This difference can be explained by the lack of a catalytic gluta- mate in the active center that is also lacking in the majority of the other PARP family members. Therefore PARP10 is a prototype of a novel subclass of PARPs restricted to mono-ADP-ribosylation. So far only little is known about PARP10’s physiological function. New experimental methods would expand the attempts to gain insight into the biology of PARP10. As one instrument siRNA-resistant wildtype PARP10 expression vectors have been designed. Using siRNA-resistant vectors allows analyzing the effects of mutants with and without the presence of endogenous proteins. In the attempts of purifying a ADP-ribosylated peptide containing an automodifi- cation site of PARP10 for immunization purposes, we tried different approaches to define the suitability of the peptide as PARP10 substrate. -

Genomic Analyses Identify Hundreds of Variants Associated with Age at Menarche and Support a Role for Puberty Timing in Cancer Risk
Genomic analyses identify hundreds of variants associated with age at menarche and support a role for puberty timing in cancer risk The MIT Faculty has made this article openly available. Please share how this access benefits you. Your story matters. Citation Day, Felix R. et al. "Genomic analyses identify hundreds of variants associated with age at menarche and support a role for puberty timing in cancer risk." Nature Genetics 49 (April 2017): 834-841© 2017 Nature America, Inc., part of Springer Nature As Published http://dx.doi.org/10.1038/NG.3841 Publisher Nature Publishing Group Version Author's final manuscript Citable link https://hdl.handle.net/1721.1/121221 Terms of Use Creative Commons Attribution-Noncommercial-Share Alike Detailed Terms http://creativecommons.org/licenses/by-nc-sa/4.0/ HHS Public Access Author manuscript Author ManuscriptAuthor Manuscript Author Nat Genet Manuscript Author . Author manuscript; Manuscript Author available in PMC 2018 June 01. Published in final edited form as: Nat Genet. 2017 June ; 49(6): 834–841. doi:10.1038/ng.3841. Genomic analyses identify hundreds of variants associated with age at menarche and support a role for puberty timing in cancer risk A full list of authors and affiliations appears at the end of the article. Abstract The timing of puberty is a highly polygenic childhood trait that is epidemiologically associated with various adult diseases. Using 1000 Genomes Project–imputed genotype data in up to ~370,000 women, we identify 389 independent signals (P < 5 × 10−8) for age at menarche, a milestone in female pubertal development. In Icelandic data, these signals explain ~7.4% of the population variance in age at menarche, corresponding to ~25% of the estimated heritability. -

Integration of Lncrna and Mrna Transcriptome Analyses Reveals
G C A T T A C G G C A T genes Article Integration of lncRNA and mRNA Transcriptome Analyses Reveals Genes and Pathways Potentially Involved in Calf Intestinal Growth and Development during the Early Weeks of Life Eveline M. Ibeagha-Awemu 1,* ID , Duy N. Do 1,2, Pier-Luc Dudemaine 1, Bridget E. Fomenky 1,3 and Nathalie Bissonnette 1 ID 1 Agriculture and Agri-Food Canada, Sherbrooke Research and Development Centre, Sherbrooke, QC J1M 0C8, Canada; [email protected] (D.N.D.); [email protected] (P.-L.D.); [email protected] (B.E.F.); [email protected] (N.B.) 2 Department of Animal Science, McGill University, Ste-Anne-De Bellevue, QC H9X 3V9, Canada 3 Département des Sciences Animales, Université Laval, Québec, QC G1V 0A9, Canada * Correspondence: [email protected]; Tel.: +1-819-780-7249 Received: 12 November 2017; Accepted: 21 February 2018; Published: 5 March 2018 Abstract: A better understanding of the factors that regulate growth and immune response of the gastrointestinal tract (GIT) of calves will promote informed management practices in calf rearing. This study aimed to explore genomics (messenger RNA (mRNA)) and epigenomics (long non-coding RNA (lncRNA)) mechanisms regulating the development of the rumen and ileum in calves. Thirty-two calves (≈5-days-old) were reared for 96 days following standard procedures. Sixteen calves were humanely euthanized on experiment day 33 (D33) (pre-weaning) and another 16 on D96 (post-weaning) for collection of ileum and rumen tissues. RNA from tissues was subjected to next generation sequencing and 3310 and 4217 mRNAs were differentially expressed (DE) between D33 and D96 in ileum and rumen tissues, respectively. -

Regulation of Gsk3β by Mono-ADP-Ribosylation
Feijs et al. Cell Communication and Signaling 2013, 11:5 http://www.biosignaling.com/content/11/1/5 RESEARCH Open Access ARTD10 substrate identification on protein microarrays: regulation of GSK3β by mono-ADP-ribosylation Karla LH Feijs1, Henning Kleine1,3, Anne Braczynski1,4, Alexandra H Forst1, Nicolas Herzog1, Patricia Verheugd1, Ulrike Linzen1,5, Elisabeth Kremmer2 and Bernhard Lüscher1* Abstract Background: Although ADP-ribosylation has been described five decades ago, only recently a distinction has been made between eukaryotic intracellular poly- and mono-ADP-ribosylating enzymes. Poly-ADP-ribosylation by ARTD1 (formerly PARP1) is best known for its role in DNA damage repair. Other polymer forming enzymes are ARTD2 (formerly PARP2), ARTD3 (formerly PARP3) and ARTD5/6 (formerly Tankyrase 1/2), the latter being involved in Wnt signaling and regulation of 3BP2. Thus several different functions of poly-ADP-ribosylation have been well described whereas intracellular mono-ADP-ribosylation is currently largely undefined. It is for example not known which proteins function as substrate for the different mono-ARTDs. This is partially due to lack of suitable reagents to study mono-ADP-ribosylation, which limits the current understanding of this post-translational modification. Results: We have optimized a novel screening method employing protein microarrays, ProtoArrays®, applied here for the identification of substrates of ARTD10 (formerly PARP10) and ARTD8 (formerly PARP14). The results of this substrate screen were validated using in vitro ADP-ribosylation assays with recombinant proteins. Further analysis of the novel ARTD10 substrate GSK3β revealed mono-ADP-ribosylation as a regulatory mechanism of kinase activity by non-competitive inhibition in vitro. -
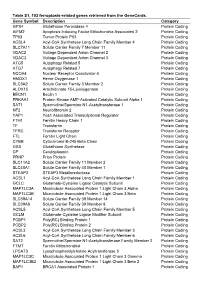
Table S1. 103 Ferroptosis-Related Genes Retrieved from the Genecards
Table S1. 103 ferroptosis-related genes retrieved from the GeneCards. Gene Symbol Description Category GPX4 Glutathione Peroxidase 4 Protein Coding AIFM2 Apoptosis Inducing Factor Mitochondria Associated 2 Protein Coding TP53 Tumor Protein P53 Protein Coding ACSL4 Acyl-CoA Synthetase Long Chain Family Member 4 Protein Coding SLC7A11 Solute Carrier Family 7 Member 11 Protein Coding VDAC2 Voltage Dependent Anion Channel 2 Protein Coding VDAC3 Voltage Dependent Anion Channel 3 Protein Coding ATG5 Autophagy Related 5 Protein Coding ATG7 Autophagy Related 7 Protein Coding NCOA4 Nuclear Receptor Coactivator 4 Protein Coding HMOX1 Heme Oxygenase 1 Protein Coding SLC3A2 Solute Carrier Family 3 Member 2 Protein Coding ALOX15 Arachidonate 15-Lipoxygenase Protein Coding BECN1 Beclin 1 Protein Coding PRKAA1 Protein Kinase AMP-Activated Catalytic Subunit Alpha 1 Protein Coding SAT1 Spermidine/Spermine N1-Acetyltransferase 1 Protein Coding NF2 Neurofibromin 2 Protein Coding YAP1 Yes1 Associated Transcriptional Regulator Protein Coding FTH1 Ferritin Heavy Chain 1 Protein Coding TF Transferrin Protein Coding TFRC Transferrin Receptor Protein Coding FTL Ferritin Light Chain Protein Coding CYBB Cytochrome B-245 Beta Chain Protein Coding GSS Glutathione Synthetase Protein Coding CP Ceruloplasmin Protein Coding PRNP Prion Protein Protein Coding SLC11A2 Solute Carrier Family 11 Member 2 Protein Coding SLC40A1 Solute Carrier Family 40 Member 1 Protein Coding STEAP3 STEAP3 Metalloreductase Protein Coding ACSL1 Acyl-CoA Synthetase Long Chain Family Member 1 Protein -

ADP-Ribose and Analogues Bound to the Demarylating Macrodomain from the Bat Coronavirus HKU4
ADP-ribose and analogues bound to the deMARylating macrodomain from the bat coronavirus HKU4 Robert G. Hammonda, Norbert Schormannb, Robert Lyle McPhersonc, Anthony K. L. Leungc,d,e, Champion C. S. Deivanayagamb, and Margaret A. Johnsona,1 aDepartment of Chemistry, University of Alabama at Birmingham, Birmingham, AL 35294; bDepartment of Biochemistry and Molecular Genetics, University of Alabama at Birmingham, Birmingham, AL 35294; cDepartment of Biochemistry and Molecular Biology, Bloomberg School of Public Health, Johns Hopkins University, Baltimore, MD 21205; dDepartment of Molecular Biology and Genetics, School of Medicine, Johns Hopkins University, Baltimore, MD 21205; and eDepartment of Oncology, School of Medicine, Johns Hopkins University, Baltimore, MD 21287 Edited by Timothy J. Mitchison, Harvard University, Boston, MA, and approved November 3, 2020 (received for review April 3, 2020) Macrodomains are proteins that recognize and hydrolyze ADP cleft most commonly used to bind mono-ADP ribose (ADPR) ribose (ADPR) modifications of intracellular proteins. Macrodo- (MAR) and poly-ADPR (PAR) (21–23). mains are implicated in viral genome replication and interference Viral macrodomains remove MAR and/or PAR modifications with host cell immune responses. They are important to the from acidic (D and E) residues by hydrolyzing the ester bond infectious cycle of Coronaviridae and Togaviridae viruses. We de- (i.e., deMARylation) (11, 24). The hepatitis E virus (HEV) mac- scribe crystal structures of the conserved macrodomain from the bat rodomain hydrolyzes PAR modifications from proteins efficiently coronavirus (CoV) HKU4 in complex with ligands. The structures re- in vitro through the association with another helicase, but similar veal a binding cavity that accommodates ADPR and analogs via local dePARylation activities were not observed in other proteins (24). -

The Antiviral Activities of Poly-ADP-Ribose Polymerases
viruses Review The Antiviral Activities of Poly-ADP-Ribose Polymerases Mathilde Malgras 1, Magali Garcia 1,2, Clément Jousselin 1,2, Charles Bodet 1 and Nicolas Lévêque 1,2,* 1 Laboratoire Inflammation Tissus Epithéliaux et Cytokines, Université de Poitiers, 86073 Poitiers, France; [email protected] (M.M.); [email protected] (M.G.); [email protected] (C.J.); [email protected] (C.B.) 2 Laboratoire de Virologie et Mycobactériologie, CHU de Poitiers, 86021 Poitiers, France * Correspondence: nicolas.lévê[email protected]; Tel.: +33-(0)5-49-44-38-17 Abstract: The poly-adenosine diphosphate (ADP)-ribose polymerases (PARPs) are responsible for ADP-ribosylation, a reversible post-translational modification involved in many cellular processes including DNA damage repair, chromatin remodeling, regulation of translation and cell death. In addition to these physiological functions, recent studies have highlighted the role of PARPs in host defenses against viruses, either by direct antiviral activity, targeting certain steps of virus replication cycle, or indirect antiviral activity, via modulation of the innate immune response. This review focuses on the antiviral activity of PARPs, as well as strategies developed by viruses to escape their action. Keywords: virus; PARP; antiviral; immunomodulation; viral escape mechanisms 1. Introduction Poly-adenosine diphosphate (ADP)-ribose polymerases (PARPs) are a family of en- Citation: Malgras, M.; Garcia, M.; zymes responsible for ADP-ribosylation, a reversible and transient post-translational modi- Jousselin, C.; Bodet, C.; Lévêque, N. The Antiviral Activities of fication of various target proteins including histones, enzymes, transcription factors and Poly-ADP-Ribose Polymerases. even PARPs themselves [1]. -

Genomics of Ovarian Cancer Progression Reveals Diverse Metastatic Trajectories Including Intraepithelial Metastasis to the Fallopian Tube
Published OnlineFirst October 7, 2016; DOI: 10.1158/2159-8290.CD-16-0607 RESEARCH BRIEF Genomics of Ovarian Cancer Progression Reveals Diverse Metastatic Trajectories Including Intraepithelial Metastasis to the Fallopian Tube Mark A. Eckert1, Shawn Pan1, Kyle M. Hernandez2, Rachel M. Loth1, Jorge Andrade2, Samuel L. Volchenboum2,3, Pieter Faber4, Anthony Montag5, Ricardo Lastra5, Marcus E. Peter6, S. Diane Yamada1, and Ernst Lengyel1 ABSTRACT Accumulating evidence has supported the fallopian tube rather than the ovary as the origin for high-grade serous ovarian cancer (HGSOC). To understand the relationship between putative precursor lesions and metastatic tumors, we performed whole-exome sequencing on specimens from eight HGSOC patient progression series consisting of serous tubal intraepithelial carcinomas (STIC), invasive fallopian tube lesions, invasive ovarian lesions, and omental metastases. Integration of copy number and somatic mutations revealed patient-specific patterns with similar mutational signatures and copy-number variation profiles across all anatomic sites, suggesting that genomic instability is an early event in HGSOC. Phylogenetic analyses supported STIC as precursor lesions in half of our patient cohort, but also identified STIC as metastases in 2 patients.Ex vivo assays revealed that HGSOC spheroids can implant in the fallopian tube epithelium and mimic STIC lesions. That STIC may represent metastases calls into question the assumption that STIC are always indicative of primary fallopian tube cancers. SIGNIFICANCE: We find that the putative precursor lesions for HGSOC, STIC, possess most of the genomic aberrations present in advanced cancers. In addition, a proportion of STIC represent intraepi- thelial metastases to the fallopian tube rather than the origin of HGSOC.