Diaezdbioinfo.Pdf
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Characterization of Genomic Copy Number Variation in Mus Musculus Associated with the Germline of Inbred and Wild Mouse Populations, Normal Development, and Cancer
Western University Scholarship@Western Electronic Thesis and Dissertation Repository 4-18-2019 2:00 PM Characterization of genomic copy number variation in Mus musculus associated with the germline of inbred and wild mouse populations, normal development, and cancer Maja Milojevic The University of Western Ontario Supervisor Hill, Kathleen A. The University of Western Ontario Graduate Program in Biology A thesis submitted in partial fulfillment of the equirr ements for the degree in Doctor of Philosophy © Maja Milojevic 2019 Follow this and additional works at: https://ir.lib.uwo.ca/etd Part of the Genetics and Genomics Commons Recommended Citation Milojevic, Maja, "Characterization of genomic copy number variation in Mus musculus associated with the germline of inbred and wild mouse populations, normal development, and cancer" (2019). Electronic Thesis and Dissertation Repository. 6146. https://ir.lib.uwo.ca/etd/6146 This Dissertation/Thesis is brought to you for free and open access by Scholarship@Western. It has been accepted for inclusion in Electronic Thesis and Dissertation Repository by an authorized administrator of Scholarship@Western. For more information, please contact [email protected]. Abstract Mus musculus is a human commensal species and an important model of human development and disease with a need for approaches to determine the contribution of copy number variants (CNVs) to genetic variation in laboratory and wild mice, and arising with normal mouse development and disease. Here, the Mouse Diversity Genotyping array (MDGA)-approach to CNV detection is developed to characterize CNV differences between laboratory and wild mice, between multiple normal tissues of the same mouse, and between primary mammary gland tumours and metastatic lung tissue. -

A Computational Approach for Defining a Signature of Β-Cell Golgi Stress in Diabetes Mellitus
Page 1 of 781 Diabetes A Computational Approach for Defining a Signature of β-Cell Golgi Stress in Diabetes Mellitus Robert N. Bone1,6,7, Olufunmilola Oyebamiji2, Sayali Talware2, Sharmila Selvaraj2, Preethi Krishnan3,6, Farooq Syed1,6,7, Huanmei Wu2, Carmella Evans-Molina 1,3,4,5,6,7,8* Departments of 1Pediatrics, 3Medicine, 4Anatomy, Cell Biology & Physiology, 5Biochemistry & Molecular Biology, the 6Center for Diabetes & Metabolic Diseases, and the 7Herman B. Wells Center for Pediatric Research, Indiana University School of Medicine, Indianapolis, IN 46202; 2Department of BioHealth Informatics, Indiana University-Purdue University Indianapolis, Indianapolis, IN, 46202; 8Roudebush VA Medical Center, Indianapolis, IN 46202. *Corresponding Author(s): Carmella Evans-Molina, MD, PhD ([email protected]) Indiana University School of Medicine, 635 Barnhill Drive, MS 2031A, Indianapolis, IN 46202, Telephone: (317) 274-4145, Fax (317) 274-4107 Running Title: Golgi Stress Response in Diabetes Word Count: 4358 Number of Figures: 6 Keywords: Golgi apparatus stress, Islets, β cell, Type 1 diabetes, Type 2 diabetes 1 Diabetes Publish Ahead of Print, published online August 20, 2020 Diabetes Page 2 of 781 ABSTRACT The Golgi apparatus (GA) is an important site of insulin processing and granule maturation, but whether GA organelle dysfunction and GA stress are present in the diabetic β-cell has not been tested. We utilized an informatics-based approach to develop a transcriptional signature of β-cell GA stress using existing RNA sequencing and microarray datasets generated using human islets from donors with diabetes and islets where type 1(T1D) and type 2 diabetes (T2D) had been modeled ex vivo. To narrow our results to GA-specific genes, we applied a filter set of 1,030 genes accepted as GA associated. -

Anti-MED30 (Aa 1-100) Polyclonal Antibody (CABT- BL5153) This Product Is for Research Use Only and Is Not Intended for Diagnostic Use
Anti-MED30 (aa 1-100) polyclonal antibody (CABT- BL5153) This product is for research use only and is not intended for diagnostic use. PRODUCT INFORMATION Product Overview Rabbit polyclonal antibody to Human MED30. Antigen Description The multiprotein TRAP/Mediator complex facilitates gene expression through a wide variety of transcriptional activators. MED30 is a component of this complex that appears to be metazoan specific (Baek et al., 2002 Immunogen Synthetic peptide conjugated to KLH derived from within residues 1 - 100 of Human MED30 / TRAP25. Isotype IgG Source/Host Rabbit Species Reactivity Human Purification Immunogen affinity purified Conjugate Unconjugated Cellular Localization Nuclear Format Liquid Size 100 μg Buffer 1% BSA, PBS, pH 7.4 Preservative 0.02% Sodium Azide Storage Store at 4°C short term (1-2 weeks). Aliquot and store at -20°C or -80°C. Avoid repeated freeze/thaw cycles. BACKGROUND Introduction The multiprotein TRAP/Mediator complex facilitates gene expression through a wide variety of transcriptional activators. MED30 is a component of this complex that appears to be metazoan specific (Baek et al., 2002 [PubMed 11909976]).[supplied by OMIM, Nov 2010] 45-1 Ramsey Road, Shirley, NY 11967, USA Email: [email protected] Tel: 1-631-624-4882 Fax: 1-631-938-8221 1 © Creative Diagnostics All Rights Reserved GENE INFORMATION Entrez Gene ID 90390 Protein Refseq NP_542382 UniProt ID Q96HR3 Chromosome Location 8q24.11 Pathway Developmental Biology, organism-specific biosystem; Gene Expression, organism-specific -

Identification of Protein Complexes Associated with Myocardial Infarction Using a Bioinformatics Approach
MOLECULAR MEDICINE REPORTS 18: 3569-3576, 2018 Identification of protein complexes associated with myocardial infarction using a bioinformatics approach NIANHUI JIAO1, YONGJIE QI1, CHANGLI LV2, HONGJUN LI3 and FENGYONG YANG1 1Intensive Care Unit; 2Emergency Department, Laiwu People's Hospital, Laiwu, Shandong 271199; 3Emergency Department, The Central Hospital of Tai'an, Tai'an, Shandong 271000, P.R. China Received November 3, 2016; Accepted January 3, 2018 DOI: 10.3892/mmr.2018.9414 Abstract. Myocardial infarction (MI) is a leading cause of clinical outcomes regarding high-risk MI. For example, muta- mortality and disability worldwide. Determination of the tions in the myocardial infarction-associated transcript have molecular mechanisms underlying the disease is crucial for been reported to cause susceptibility to MI (5). In addition, it identifying possible therapeutic targets and designing effective was demonstrated that mutations in the oxidized low-density treatments. On the basis that MI may be caused by dysfunc- lipoprotein receptor 1 gene may significantly increase the tional protein complexes rather than single genes, the present risk of MI (6). Although some MI-related genes have been study aimed to use a bioinformatics approach to identifying detected, many were identified independently and functional complexes that may serve important roles in the develop- associations among the genes have rarely been explored. ment of MI. By investigating the proteins involved in these Therefore, it is necessary to investigate MI from a systematic identified complexes, numerous proteins have been reported perspective, as the complex disease was reported to occur due that are related to MI, whereas other proteins interacted to the dysregulation of functional gene sets (7). -

Noelia Díaz Blanco
Effects of environmental factors on the gonadal transcriptome of European sea bass (Dicentrarchus labrax), juvenile growth and sex ratios Noelia Díaz Blanco Ph.D. thesis 2014 Submitted in partial fulfillment of the requirements for the Ph.D. degree from the Universitat Pompeu Fabra (UPF). This work has been carried out at the Group of Biology of Reproduction (GBR), at the Department of Renewable Marine Resources of the Institute of Marine Sciences (ICM-CSIC). Thesis supervisor: Dr. Francesc Piferrer Professor d’Investigació Institut de Ciències del Mar (ICM-CSIC) i ii A mis padres A Xavi iii iv Acknowledgements This thesis has been made possible by the support of many people who in one way or another, many times unknowingly, gave me the strength to overcome this "long and winding road". First of all, I would like to thank my supervisor, Dr. Francesc Piferrer, for his patience, guidance and wise advice throughout all this Ph.D. experience. But above all, for the trust he placed on me almost seven years ago when he offered me the opportunity to be part of his team. Thanks also for teaching me how to question always everything, for sharing with me your enthusiasm for science and for giving me the opportunity of learning from you by participating in many projects, collaborations and scientific meetings. I am also thankful to my colleagues (former and present Group of Biology of Reproduction members) for your support and encouragement throughout this journey. To the “exGBRs”, thanks for helping me with my first steps into this world. Working as an undergrad with you Dr. -

Functional Diversification of the Nleg Effector Family in Enterohemorrhagic Escherichia Coli
Functional diversification of the NleG effector family in enterohemorrhagic Escherichia coli Dylan Valleaua, Dustin J. Littleb, Dominika Borekc,d, Tatiana Skarinaa, Andrew T. Quailea, Rosa Di Leoa, Scott Houlistone,f, Alexander Lemake,f, Cheryl H. Arrowsmithe,f, Brian K. Coombesb, and Alexei Savchenkoa,g,1 aDepartment of Chemical Engineering and Applied Chemistry, University of Toronto, Toronto, ON M5S 3E5, Canada; bDepartment of Biochemistry and Biomedical Sciences, Michael G. DeGroote Institute for Infectious Disease Research, McMaster University, Hamilton, ON L8S 4K1, Canada; cDepartment of Biophysics, University of Texas Southwestern Medical Center, Dallas, TX 75390; dDepartment of Biochemistry, University of Texas Southwestern Medical Center, Dallas, TX 75390; ePrincess Margaret Cancer Centre, University of Toronto, Toronto, ON M5G 2M9, Canada; fDepartment of Medical Biophysics, University of Toronto, Toronto, ON M5G 1L7, Canada; and gDepartment of Microbiology, Immunology and Infectious Diseases, University of Calgary, Calgary, AB T2N 4N1, Canada Edited by Ralph R. Isberg, Howard Hughes Medical Institute and Tufts University School of Medicine, Boston, MA, and approved August 15, 2018 (receivedfor review November 6, 2017) The pathogenic strategy of Escherichia coli and many other gram- chains. Depending on the length and nature of the polyubiquitin negative pathogens relies on the translocation of a specific set of chain, it posttranslationally regulates the target protein’s locali- proteins, called effectors, into the eukaryotic host cell during in- zation, activation, or degradation. Ubiquitination is a multistep fection. These effectors act in concert to modulate host cell pro- process which begins with the ubiquitin-activating enzyme (E1) cesses in favor of the invading pathogen. Injected by the type III using ATP to “charge” ubiquitin, covalently binding the ubiquitin secretion system (T3SS), the effector arsenal of enterohemorrhagic C terminus by a thioester linkage. -

FBXL19 Recruits CDK-Mediator to Cpg Islands of Developmental Genes Priming Them for Activation During Lineage Commitment
RESEARCH ARTICLE FBXL19 recruits CDK-Mediator to CpG islands of developmental genes priming them for activation during lineage commitment Emilia Dimitrova1, Takashi Kondo2†, Angelika Feldmann1†, Manabu Nakayama3, Yoko Koseki2, Rebecca Konietzny4‡, Benedikt M Kessler4, Haruhiko Koseki2,5, Robert J Klose1* 1Department of Biochemistry, University of Oxford, Oxford, United Kingdom; 2Laboratory for Developmental Genetics, RIKEN Center for Integrative Medical Sciences, Yokohama, Japan; 3Department of Technology Development, Kazusa DNA Research Institute, Kisarazu, Japan; 4Nuffield Department of Medicine, TDI Mass Spectrometry Laboratory, Target Discovery Institute, University of Oxford, Oxford, United Kingdom; 5CREST, Japan Science and Technology Agency, Kawaguchi, Japan Abstract CpG islands are gene regulatory elements associated with the majority of mammalian promoters, yet how they regulate gene expression remains poorly understood. Here, we identify *For correspondence: FBXL19 as a CpG island-binding protein in mouse embryonic stem (ES) cells and show that it [email protected] associates with the CDK-Mediator complex. We discover that FBXL19 recruits CDK-Mediator to †These authors contributed CpG island-associated promoters of non-transcribed developmental genes to prime these genes equally to this work for activation during cell lineage commitment. We further show that recognition of CpG islands by Present address: ‡Agilent FBXL19 is essential for mouse development. Together this reveals a new CpG island-centric Technologies, Waldbronn, mechanism for CDK-Mediator recruitment to developmental gene promoters in ES cells and a Germany requirement for CDK-Mediator in priming these developmental genes for activation during cell lineage commitment. Competing interests: The DOI: https://doi.org/10.7554/eLife.37084.001 authors declare that no competing interests exist. -

Structure and Mechanism of the RNA Polymerase II Transcription Machinery
Downloaded from genesdev.cshlp.org on October 9, 2021 - Published by Cold Spring Harbor Laboratory Press REVIEW Structure and mechanism of the RNA polymerase II transcription machinery Allison C. Schier and Dylan J. Taatjes Department of Biochemistry, University of Colorado, Boulder, Colorado 80303, USA RNA polymerase II (Pol II) transcribes all protein-coding ingly high resolution, which has rapidly advanced under- genes and many noncoding RNAs in eukaryotic genomes. standing of the molecular basis of Pol II transcription. Although Pol II is a complex, 12-subunit enzyme, it lacks Structural biology continues to transform our under- the ability to initiate transcription and cannot consistent- standing of complex biological processes because it allows ly transcribe through long DNA sequences. To execute visualization of proteins and protein complexes at or near these essential functions, an array of proteins and protein atomic-level resolution. Combined with mutagenesis and complexes interact with Pol II to regulate its activity. In functional assays, structural data can at once establish this review, we detail the structure and mechanism of how enzymes function, justify genetic links to human dis- over a dozen factors that govern Pol II initiation (e.g., ease, and drive drug discovery. In the past few decades, TFIID, TFIIH, and Mediator), pausing, and elongation workhorse techniques such as NMR and X-ray crystallog- (e.g., DSIF, NELF, PAF, and P-TEFb). The structural basis raphy have been complemented by cryoEM, cross-linking for Pol II transcription regulation has advanced rapidly mass spectrometry (CXMS), and other methods. Recent in the past decade, largely due to technological innova- improvements in data collection and imaging technolo- tions in cryoelectron microscopy. -

Gene Ontology Functional Annotations and Pleiotropy
Network based analysis of genetic disease associations Sarah Gilman Submitted in partial fulfillment of the requirements for the degree of Doctor of Philosophy under the Executive Committee of the Graduate School of Arts and Sciences COLUMBIA UNIVERSITY 2014 © 2013 Sarah Gilman All Rights Reserved ABSTRACT Network based analysis of genetic disease associations Sarah Gilman Despite extensive efforts and many promising early findings, genome-wide association studies have explained only a small fraction of the genetic factors contributing to common human diseases. There are many theories about where this “missing heritability” might lie, but increasingly the prevailing view is that common variants, the target of GWAS, are not solely responsible for susceptibility to common diseases and a substantial portion of human disease risk will be found among rare variants. Relatively new, such variants have not been subject to purifying selection, and therefore may be particularly pertinent for neuropsychiatric disorders and other diseases with greatly reduced fecundity. Recently, several researchers have made great progress towards uncovering the genetics behind autism and schizophrenia. By sequencing families, they have found hundreds of de novo variants occurring only in affected individuals, both large structural copy number variants and single nucleotide variants. Despite studying large cohorts there has been little recurrence among the genes implicated suggesting that many hundreds of genes may underlie these complex phenotypes. The question -

Differential Roles of Transcriptional Mediator Complex Subunits
Genetics: Published Articles Ahead of Print, published on April 2, 2006 as 10.1534/genetics.105.055152 Differential roles of transcriptional mediator complex subunits Crsp34/Med27, Crsp150/Med14, and Trap100/Med24 during zebrafish retinal development Katrin Dürr, Jochen Holzschuh, Alida Filippi, Anne-Kathrin Ettl, Soojin Ryu, Iain T. Shepherd* and Wolfgang Driever Department of Developmental Biology, Institute for Biology 1, University of Freiburg, 79104 Freiburg, Germany *Department of Biology, Emory University, Rollins Research Center, Atlanta, GA 30322 Dürr et al. zebrafish crsp34 1 Running title: The mediator complex in development Keywords: transcriptional mediator complex, retina, zebrafish, cell differentiation Correspondence should be addressed to: W. Driever, Department of Developmental Biology, Institute for Biology 1, University of Freiburg, Hauptstrasse 1, D-79104 Freiburg, Germany. Tel. (xx49)-761-203-2587; fax (xx49)-761-203-2597 Email: [email protected] Dürr et al. zebrafish crsp34 2 ABSTRACT The transcriptional mediator complex has emerged as an important component of transcriptional regulation, yet it is largely unknown whether its subunits have differential functions in development. We demonstrate that the zebrafish mutation m885 disrupts a subunit of the mediator complex, Crsp34/Med27. In order to explore the role of the mediator in the control of retinal differentiation, we employed two additional mutations disrupting the mediator subunits Trap100/Med24 and Crsp150/Med14. Our analysis shows that loss of Crsp34/Med27 decreases amacrine cell number, but increases the number of rod photoreceptor cells. In contrast, loss of Trap100/Med24 decreases rod photoreceptor cells. Loss of Crsp150/Med14, on the other hand, only slightly reduces dopaminergic amacrine cells, which are absent from both crsp34m885 and trap100lessen mutant embryos. -

Genomic and Transcriptomic Investigations Into the Feed Efficiency Phenotype of Beef Cattle
Provided by the author(s) and NUI Galway in accordance with publisher policies. Please cite the published version when available. Title Genomic and transcriptomic investigations into the feed efficiency phenotype of beef cattle Author(s) Higgins, Marc Publication Date 2019-03-06 Publisher NUI Galway Item record http://hdl.handle.net/10379/15008 Downloaded 2021-09-25T18:07:39Z Some rights reserved. For more information, please see the item record link above. Genomic and Transcriptomic Investigations into the Feed Efficiency Phenotype of Beef Cattle Marc Higgins, B.Sc., M.Sc. A thesis submitted for the Degree of Doctor of Philosophy to the Discipline of Biochemistry, School of Natural Sciences, National University of Ireland, Galway. Supervisor: Dr. Derek Morris Discipline of Biochemistry, School of Natural Sciences, National University of Ireland, Galway. Supervisor: Dr. Sinéad Waters Teagasc, Animal and Bioscience Research Department, Animal & Grassland Research and Innovation Centre, Teagasc, Grange. Submitted November 2018 Table of Contents Declaration ................................................................................................................ vii Funding .................................................................................................................... viii Acknowledgements .................................................................................................... ix Abstract ...................................................................................................................... -
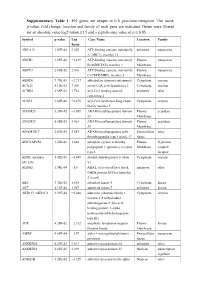
Supplementary Table 1
Supplementary Table 1. 492 genes are unique to 0 h post-heat timepoint. The name, p-value, fold change, location and family of each gene are indicated. Genes were filtered for an absolute value log2 ration 1.5 and a significance value of p ≤ 0.05. Symbol p-value Log Gene Name Location Family Ratio ABCA13 1.87E-02 3.292 ATP-binding cassette, sub-family unknown transporter A (ABC1), member 13 ABCB1 1.93E-02 −1.819 ATP-binding cassette, sub-family Plasma transporter B (MDR/TAP), member 1 Membrane ABCC3 2.83E-02 2.016 ATP-binding cassette, sub-family Plasma transporter C (CFTR/MRP), member 3 Membrane ABHD6 7.79E-03 −2.717 abhydrolase domain containing 6 Cytoplasm enzyme ACAT1 4.10E-02 3.009 acetyl-CoA acetyltransferase 1 Cytoplasm enzyme ACBD4 2.66E-03 1.722 acyl-CoA binding domain unknown other containing 4 ACSL5 1.86E-02 −2.876 acyl-CoA synthetase long-chain Cytoplasm enzyme family member 5 ADAM23 3.33E-02 −3.008 ADAM metallopeptidase domain Plasma peptidase 23 Membrane ADAM29 5.58E-03 3.463 ADAM metallopeptidase domain Plasma peptidase 29 Membrane ADAMTS17 2.67E-04 3.051 ADAM metallopeptidase with Extracellular other thrombospondin type 1 motif, 17 Space ADCYAP1R1 1.20E-02 1.848 adenylate cyclase activating Plasma G-protein polypeptide 1 (pituitary) receptor Membrane coupled type I receptor ADH6 (includes 4.02E-02 −1.845 alcohol dehydrogenase 6 (class Cytoplasm enzyme EG:130) V) AHSA2 1.54E-04 −1.6 AHA1, activator of heat shock unknown other 90kDa protein ATPase homolog 2 (yeast) AK5 3.32E-02 1.658 adenylate kinase 5 Cytoplasm kinase AK7