D:\Pindahan\Mydocuments D
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Lotus Wind Power Project
Initial Environmental Examination – Appendix H Project Number: 54211-001 March 2021 Document Stage: Draft Viet Nam: Lotus Wind Power Project Prepared by ERM Vietnam for Lien Lap Wind Power Joint Stock Company, Phong Huy Wind Power Joint Stock Company, and Phong Nguyen Wind Power Joint Stock Company as a requirement of the Asian Development Bank. The initial environmental examination is a document of the borrower. The views expressed herein do not necessarily represent those of ADB's Board of Directors, Management, or staff, and may be preliminary in nature. Your attention is directed to the “Terms of Use” section of this website. In preparing any country program or strategy, financing any project, or by making any designation of or reference to a particular territory or geographic area in this document, the Asian Development Bank does not intend to make any judgments as to the legal or other status of any territory or area. Biodiversity survey Wet season report Phong Huy Wind Power Project, Huong Hoa, Quang Tri, Viet Nam 7 July 2020 Prepared by ERM’s Subcontractor for ERM Vietnam Document details Document title Biodiversity survey Wet season report Document subtitle Phong Huy Wind Power Project, Huong Hoa, Quang Tri, Viet Nam Date 7 July 2020 Version 1.0 Author ERM’s Subcontractor Client Name ERM Vietnam Document history Version Revision Author Reviewed by ERM approval to issue Comments Name Date Draft 1.0 Name Name Name 00.00.0000 Text Version: 1.0 Client: ERM Vietnam 7 July 2020 BIODIVERSITY SURVEY WET SEASON REPORT CONTENTS Phong Huy Wind Power Project, Huong Hoa, Quang Tri, Viet Nam CONTENTS 1. -

Analysis of Colubroidea Snake Venoms by Liquid Chromatography with Mass Spectrometry: Evolutionary and Toxinological Implications
RAPID COMMUNICATIONS IN MASS SPECTROMETRY Rapid Commun. Mass Spectrom. 2003; 17: 2047–2062 Published online in Wiley InterScience (www.interscience.wiley.com). DOI: 10.1002/rcm.1148 Analysis of Colubroidea snake venoms by liquid chromatography with mass spectrometry: evolutionary and toxinological implications Bryan G. Fry1,2*, Wolfgang Wu¨ ster3, Sheik Fadil Ryan Ramjan2, Timothy Jackson1, Paolo Martelli4 and R. Manjunatha Kini2 1Australian Venom Research Unit, Department of Pharmacology, University of Melbourne, Parkville, Vic 3010, Australia 2Department of Biological Sciences, Faculty of Science, National University of Singapore 119260, Singapore 3School of Biological Sciences, University of Wales, Bangor LL57 2UW, Wales, UK 4Veterinary Department, Singapore Zoo, Mandai Rd., Singapore Received 12 June 2003; Revised 7 July 2003; Accepted 9 July 2003 The evolution of the venomous function of snakes and the diversification of the toxins has been of tremendous research interest and considerable debate. It has become recently evident that the evo- lution of the toxins in the advanced snakes (Colubroidea) predated the evolution of the advanced, front-fanged delivery mechanisms. Historically, the venoms of snakes lacking front-fanged venom- delivery systems (conventionally grouped into the paraphyletic family Colubridae) have been lar- gely neglected. In this study we used liquid chromatography with mass spectrometry (LC/MS) to analyze a large number of venoms from a wide array of species representing the major advanced snake clades Atractaspididae, -

MAHS Care Sheet Master List *By Eric Roscoe Care Sheets Are Often An
MAHS Care Sheet Master List *By Eric Roscoe Care sheets are often an excellent starting point for learning more about the biology and husbandry of a given species, including their housing/enclosure requirements, temperament and handling, diet , and other aspects of care. MAHS itself has created many such care sheets for a wide range of reptiles, amphibians, and invertebrates we believe to have straightforward care requirements, and thus make suitable family and beginner’s to intermediate level pets. Some species with much more complex, difficult to meet, or impracticable care requirements than what can be adequately explained in a one page care sheet may be multiple pages. We can also provide additional links, resources, and information on these species we feel are reliable and trustworthy if requested. If you would like to request a copy of a care sheet for any of the species listed below, or have a suggestion for an animal you don’t see on our list, contact us to let us know! Unfortunately, for liability reasons, MAHS is unable to create or publish care sheets for medically significant venomous species. This includes species in the families Crotilidae, Viperidae, and Elapidae, as well as the Helodermatidae (the Gila Monsters and Mexican Beaded Lizards) and some medically significant rear fanged Colubridae. Those that are serious about wishing to learn more about venomous reptile husbandry that cannot be adequately covered in one to three page care sheets should take the time to utilize all available resources by reading books and literature, consulting with, and working with an experienced and knowledgeable mentor in order to learn the ropes hands on. -

How Is the COVID-19 Outbreak Affecting Wildlife Around the World?
Open Journal of Ecology, 2020, 10, 497-517 https://www.scirp.org/journal/oje ISSN Online: 2162-1993 ISSN Print: 2162-1985 How Is the COVID-19 Outbreak Affecting Wildlife around the World? Abdel Fattah N. Abd Rabou Department of Biology, Faculty of Science, Islamic University of Gaza, Gaza Strip, Palestine How to cite this paper: Abd Rabou, A.N. Abstract (2020) How Is the COVID-19 Outbreak Affecting Wildlife around the World? Open The COVID-19 is the infectious disease caused by the most recently discov- Journal of Ecology, 10, 497-517. ered coronavirus at an animal market in Wuhan, China. Many wildlife spe- https://doi.org/10.4236/oje.2020.108032 cies have been suggested as possible intermediate sources for the transmission Received: June 2, 2020 of COVID-19 virus from bats to humans. The quick transmission of COVID-19 Accepted: August 1, 2020 outbreak has imposed quarantine measures across the world, and as a result, Published: August 4, 2020 most of the world’s towns and cities fell silent under lockdowns. The current Copyright © 2020 by author(s) and study comes to investigate the ways by which the COVID-19 outbreak affects Scientific Research Publishing Inc. wildlife globally. Hundreds of internet sites and scientific reports have been This work is licensed under the Creative reviewed to satisfy the needs of the study. Stories of seeing wild animals Commons Attribution International roaming the quiet, deserted streets and cities during the COVID-19 outbreak License (CC BY 4.0). http://creativecommons.org/licenses/by/4.0/ have been posted in the media and social media. -

New Record of Banded Krait Bungarus Fasciatus
Biological Forum – An International Journal 12(1): 29-32(2020) ISSN No. (Print): 0975-1130 ISSN No. (Online): 2249-3239 New Record of Banded Krait Bungarus fasciatus (Schneider, 1801) from Ranchi (Jharkhand) with its Preying on Checkered Keel-Back Snake Akhlaq Husain (Former Scientist E. Zoological Survey of India) 41, Hari Vihar, Vijay Park, Chakrata Road, Dehra Dun-248001, Uttarakhand, India. (Corresponding author: Akhlaq Husain) (Received 20 February 2020, Accepted 04 April, 2020) (Published by Research Trend, Website: www.researchtrend.net) ABSTRACT: In India Banded Krait Bungarus fasciatus (Schneider, 1801) commonly occurs in north-eastern India (Arunachal Pradesh, Assam, Manipur, Mizoram, Meghalaya, Nagaland and Tripura) but becomes lesser towards north-west, west, south and south-east (Uttarakhand in north-west; Chhattisgarh and Madhya Pradesh in central India; Maharashtra in west; Karnataka, Kerala and Tamil Nadu in south and Andhra Pradesh and Odisha in south-east). In Jharkhand it was recorded from Bokaro and Hazaribagh districts but presently it has been found in Ranchi district also, preying on Checkered Keel-back Snake which is a new record and adds to its distribution in the state. In present communication its synonymy, diagnostic features, altitudinal range, distribution, habitat, food & feeding, breeding, nature & behaviour, bite, venom & treatment, conservation status, threats and preying on Checkered Keel-back Snake are provided. Keywords: New record of Banded Krait from Ranchi with its preying on Checkered Keel-back Snake. INTRODCUCTION The records of distribution of Bungarus fasciatus (Schneider, 1801), the Banded Krait, in Jharkhand State have been from Bokaro and Hoshangabad districts only (Wikipedia; telegraphindia.com). During present study, a Banded Krait, preying on Checkered Keel-back snake (Fowlea piscator, Schneider, 1799), was sighted at Ormanjhi in Ranchi district which was found to be the new find from Ranchi and additional record for the state. -
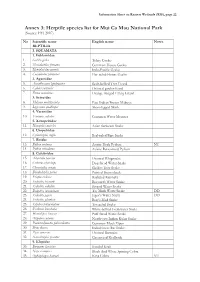
Information Sheet on Ramsar Wetlands (RIS), Page 22
Information Sheet on Ramsar Wetlands (RIS), page 22 Annex 3: Herptile species list for Mui Ca Mau National Park (Source: FFI 2007) No Scientific name English name Notes REPTILIA I. SQUAMATA 1. Gekkonidae 1. Gekko gecko Tokay Gecko 2. Hemidactylus frenatus Common House Gecko 3. Hemidactylus garnoti Indo-Pacific Gecko 4. Cosymbotus platyurus Flat-tailed House Gecko 2. Agamidae 5. Acanthosaura lepidogaster Scale-bellied Tree Lizard 6. Calotes versicolor Oriental garden lizard 7. Draco maculatus Orange-winged Flying Lizard 3. Scincidae 8. Mabuya multifasciata East Indian Brown Mabuya 9. Lygosoma quadrupes Short-legged Skink 4. Varanidae 10. Varanus salvator Common Water Monitor 5. Xenopeltidae 11. Xenopeltis unicolor Asian Sunbeam Snake 6. Uropeltidae 12. Cylindrophis ruffus Red-tailed Pipe Snake 7. Boidae 13. Python molurus Asiatic Rock Python NT 14. Python reticulatus Asiatic Reticulated Python 8. Colubridae 15. Ahaetulla prasina Oriental Whipsnake 16. Cerberus rhynchops Dog-faced Water Snake 17. Chrysopelea ornata Golden Tree Snake 18. Dendrelaphis pictus Painted Bronzeback 19. Elaphe radiata Radiated Ratsnake 20. Enhydris bocourti Bocourt's Water Snake 21. Enhydris enhydris Striped Water Snake 22. Enhydris innominata Tay Minh Water Snake DD 23. Enhydris jagori Jagor's Water Snake DD 24. Enhydris plumbea Boie's Mud Snake 25. Erpeton tentaculatum Tentacled Snake 26. Fordonia leucobalia White-bellied Freshwater Snake 27. Homalopsis buccata Puff-faced Water Snake 28. Oligodon cyclurus North-east Indian Kukri Snake 29. Psammodynastes pulverulentus Common Mock Viper 30. Ptyas korros Indochinese Rat Snake 31. Ptyas mucosus Oriental Ratsnake 32. Xenochrophis piscator Chequered Keelback 9. Elapidae 33. Bungarus fasciatus Banded Krait 34. Naja siamensis Black And White Spitting Cobra 35. -

Notice Warning Concerning Copyright Restrictions P.O
Publisher of Journal of Herpetology, Herpetological Review, Herpetological Circulars, Catalogue of American Amphibians and Reptiles, and three series of books, Facsimile Reprints in Herpetology, Contributions to Herpetology, and Herpetological Conservation Officers and Editors for 2015-2016 President AARON BAUER Department of Biology Villanova University Villanova, PA 19085, USA President-Elect RICK SHINE School of Biological Sciences University of Sydney Sydney, AUSTRALIA Secretary MARION PREEST Keck Science Department The Claremont Colleges Claremont, CA 91711, USA Treasurer ANN PATERSON Department of Natural Science Williams Baptist College Walnut Ridge, AR 72476, USA Publications Secretary BRECK BARTHOLOMEW Notice warning concerning copyright restrictions P.O. Box 58517 Salt Lake City, UT 84158, USA Immediate Past-President ROBERT ALDRIDGE Saint Louis University St Louis, MO 63013, USA Directors (Class and Category) ROBIN ANDREWS (2018 R) Virginia Polytechnic and State University, USA FRANK BURBRINK (2016 R) College of Staten Island, USA ALISON CREE (2016 Non-US) University of Otago, NEW ZEALAND TONY GAMBLE (2018 Mem. at-Large) University of Minnesota, USA LISA HAZARD (2016 R) Montclair State University, USA KIM LOVICH (2018 Cons) San Diego Zoo Global, USA EMILY TAYLOR (2018 R) California Polytechnic State University, USA GREGORY WATKINS-COLWELL (2016 R) Yale Peabody Mus. of Nat. Hist., USA Trustee GEORGE PISANI University of Kansas, USA Journal of Herpetology PAUL BARTELT, Co-Editor Waldorf College Forest City, IA 50436, USA TIFFANY -

Australasian Journal of Herpetology ISSN 1836-5698 (Print)1 Issue 12, 30 April 2012 ISSN 1836-5779 (Online) Australasian Journal of Herpetology
Australasian Journal of Herpetology ISSN 1836-5698 (Print)1 Issue 12, 30 April 2012 ISSN 1836-5779 (Online) Australasian Journal of Herpetology Hoser 2012 - Australasian Journal of Herpetology 9:1-64. Available online at www.herp.net Contents on pageCopyright- 2. Kotabi Publishing - All rights reserved 2 Australasian Journal of Herpetology Issue 12, 30 April 2012 Australasian Journal of Herpetology CONTENTS ISSN 1836-5698 (Print) ISSN 1836-5779 (Online) A New Genus of Coral Snake from Japan (Serpentes:Elapidae). Raymond T. Hoser, 3-5. A revision of the Asian Pitvipers, referred to the genus Cryptelytrops Cope, 1860, with the creation of a new genus Adelynhoserea to accommodate six divergent species (Serpentes:Viperidae:Crotalinae). Raymond T. Hoser, 6-8. A division of the South-east Asian Ratsnake genus Coelognathus (Serpentes: Colubridae). Raymond T. Hoser, 9-11. A new genus of Asian Snail-eating Snake (Serpentes:Pareatidae). Raymond T. Hoser, 10-12-15. The dissolution of the genus Rhadinophis Vogt, 1922 (Sepentes:Colubrinae). Raymond T. Hoser, 16-17. Three new species of Stegonotus from New Guinea (Serpentes: Colubridae). Raymond T. Hoser, 18-22. A new genus and new subgenus of snakes from the South African region (Serpentes: Colubridae). Raymond T. Hoser, 23-25. A division of the African Genus Psammophis Boie, 1825 into 4 genera and four further subgenera (Serpentes: Psammophiinae). Raymond T. Hoser, 26-31. A division of the African Tree Viper genus Atheris Cope, 1860 into four subgenera (Serpentes:Viperidae). Raymond T. Hoser, 32-35. A new Subgenus of Giant Snakes (Anaconda) from South America (Serpentes: Boidae). Raymond T. Hoser, 36-39. -

EU WILDLIFE TRADE 2017 Analysis of the European Union Annual Reports to CITES 2017 EU Wildlife Trade 2017
EU WILDLIFE TRADE 2017 Analysis of the European Union annual reports to CITES 2017 EU Wildlife Trade 2017 EU Wildlife Trade 2017: Analysis of the European Union’s annual reports to CITES 2017 Prepared for: The European Commission, Directorate General CONTENTS Environment, Directorate F – Global Sustainable Development, ENV.F3 – Multilateral Environmental Cooperation, Brussels, Belgium. Executive Summary 1 Published: January 2019. 1. Introduction 3 Copyright: European Commission 2019. 2. Overview of CITES trade 4 Citation: UNEP-WCMC. 2019. EU Wildlife Trade 2017: Analysis of Imports 4 the European Union’s annual reports to CITES 2017. Exports 8 3. Species showing noteworthy trends 13 Contributing Authors: Becky Price, Kelly Malsch, Aly Pavitt, Oliver Tallowin, Sophie Banks, Jessica Vitale, Aude Caromel, Emma Vovk Methods 13 and Pablo Sinovas. Taxa selected 13 4. Value of EU trade 20 Value of plant trade 20 Value of animal trade 23 The UN Environment World Conservation Monitoring Centre (UNEP-WCMC) is the specialist biodiversity centre of UN Annex A: Supplementary information 27 Environment, the world’s foremost intergovernmental environmental organisation. The Centre has been in operation for Annex B: Valuation methodology 30 40 years, combining scientific research with practical policy advice. This publication may be reproduced for educational or non-profit purposes without special permission, provided acknowledgement to the source is made. Reuse of any figures is subject to permission from the original rights holders. No use of this publication may be made for resale or any other commercial purpose without permission in writing from UN Environment. Applications for permission, with a statement of purpose and extent of reproduction, should be sent to the Director, UNEP- WCMC, 219 Huntingdon Road, Cambridge, CB3 0DL, UK. -

Boiga Cynodon • Characteristics
Photos of Common THAILAND SNAKES VERN LOVIC - THAILANDSNAKES.COM INTRODUCTION Welcome! Now that you’ve picked up this free ebook, share it with your friends. You can view it on almost any computer, smart phone, or tablet. It is available at the Apple iTunes store (free) and in PDF format at ThailandSnakes.com/ ebook/. This book covers what we believe to be the most common terrestrial (land-based) and freshwater snakes in Thailand, those you are likely to see - if you see any at all. We wrote this book to help educate the public and to hopefully save a few snakes from the shovel or machete. As you view the photos within, keep in mind that there are albino (no melanin) and melanistic (abundance of melanin) snakes that will not exhibit the same colors as most snakes of the species. Albino snakes can be pure white, or mostly white with a different colored pattern. Melanistic snakes are very dark, even solid black. So, you might see a white snake with a yellow pattern that looks exactly like the deadly Russell’s viper (Daboia russellii siamensis) - but the color is way off because it’s albino. It is still a deadly snake. We will release FREE UPDATES to this book in the future. If you haven’t signed up to be notified of updates - you won’t get them. Sign Up for Free Book Updates and Newsletter HERE (click red link) > If the link above does not work, visit: www.ThailandSnakes.com/ebook/ SNAKE BITE? Steps to take in the case of snake bite falls outside the scope of this ebook. -

On the Trail N°25
The defaunation bulletin Quarterly information and analysis report on animal poaching and smuggling n°25. Events from the 1st April 2019 to the 30 of June 2019 Published on January 9, 2020 Original version in French 1 On the Trail n°25. Robin des Bois Carried out by Robin des Bois (Robin Hood) with the support of the Brigitte Bardot Foundation, the Franz Weber Foundation and of the Ministry of Ecological and Solidarity Transition, France reconnue d’utilité publique 28, rue Vineuse - 75116 Paris Tél : 01 45 05 14 60 “On the Trail“, the defaunationwww.fondationbrigittebardot.fr magazine, aims to get out of the drip of daily news to draw up every three months an organized and analyzed survey of poaching, smuggling and worldwide market of animal species protected by national laws and international conventions. “ On the Trail “ highlights the new weapons of plunderers, the new modus operandi of smugglers, rumours intended to attract humans consumers of animals and their by-products.“ On the Trail “ gathers and disseminates feedback from institutions, individuals and NGOs that fight against poaching and smuggling. End to end, the “ On the Trail “ are the biological, social, ethnological, police, customs, legal and financial chronicle of poaching and other conflicts between humanity and animality. Previous issues in English http://www.robindesbois.org/en/a-la-trace-bulletin-dinformation-et-danalyses-sur-le-braconnage-et-la-contrebande/ Previous issues in French http://www.robindesbois.org/a-la-trace-bulletin-dinformation-et-danalyses-sur-le-braconnage-et-la-contrebande/ Non Governmental Organization for the Protection of Man and the Environment Since 1985 14 rue de l’Atlas 75019 Paris, France tel : 33 (1) 48.04.09.36 - fax : 33 (1) 48.04.56.41 www.robindesbois.org [email protected] Publication Director : Jacky Bonnemains Editor-in-Chief: Charlotte Nithart Art Directors : Charlotte Nithart and Jacky Bonnemains Coordination : Elodie Crépeau Writing: Jacky Bonnemains, Jean-Pierre Edin and Camille Habe. -

The Use of Midazolam, Isoflurane, and Nitrous Oxide for Sedation and Anesthesia of Ball Pythons (Python Regius)
The Use of Midazolam, Isoflurane, and Nitrous Oxide for Sedation and Anesthesia of Ball Pythons (Python regius) by Cédric B. Larouche A Thesis presented to The University of Guelph In partial fulfilment of requirements for the degree of Doctor of Veterinary Science in Pathobiology Guelph, Ontario, Canada © Cédric B. Larouche, August, 2019 ABSTRACT THE USE OF MIDAZOLAM, ISOFLURANE, AND NITROUS OXIDE FOR SEDATION AND ANESTHESIA OF BALL PYTHONS (PYTHON REGIUS) Cédric B. Larouche Co-advisors: University of Guelph, 2019 Nicole Nemeth Dorothee Bienzle The objectives of this project were to characterize the pharmacodynamics and pharmacokinetics of midazolam in the ball python (Python regius), and to evaluate the effects of midazolam and nitrous oxide (N2O) on the minimum anesthetic concentration of isoflurane (MACIso) in this species. The pharmacodynamics of midazolam were evaluated in a blinded, randomized, crossover study comparing two dosages (1 and 2 mg/kg intramuscular [IM]) in ten ball pythons. There were no significant differences between the two dosages for any parameter evaluated. Midazolam provided moderate sedation and muscle relaxation in all snakes starting 10-15 min post-injection and lasting up to 3-5 days, with a peak effect 60 min post-injection. Heart rates were significantly lower than baseline from 30 min to 5.3 days post-injection with both dosages. In an open trial using 9 ball pythons, the reversal effect of flumazenil was evaluated by administering 0.08 mg/kg IM 60 min after the administration of 1 mg/kg IM of midazolam. Sedation and muscle relaxation were fully reversed within 10 min of administration, but all snakes fully resedated within 3 h.