Tesis (4.391Mb)
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Chapter 11 Marine Fungi Associated with Antarctic Macroalgae
Chapter 11 Marine Fungi Associated with Antarctic Macroalgae Mayara B. Ogaki, Maria T. de Paula, Daniele Ruas, Franciane M. Pellizzari, César X. García-Laviña, and Luiz H. Rosa Abstract Fungi are well known for their important roles in terrestrial ecosystems, but filamentous and yeast forms are also active components of microbial communi- ties from marine ecosystems. Marine fungi are particularly abundant and relevant in coastal systems where they can be found in association with large organic substrata, like seaweeds. Antarctica is a rather unexplored region of the planet that is being influenced by strong and rapid climate change. In the past decade, several efforts have been made to get a thorough inventory of marine fungi from different environ- ments, with a particular emphasis on those associated with the large communities of seaweeds that abound in littoral and infralittoral ecosystems. The algicolous fungal communities obtained were characterized by a few dominant species and a large number of singletons, as well as a balance among endemic, indigenous, and cold- adapted cosmopolitan species. The long-term monitoring of this balance and the dynamics of richness, dominance, and distributional patterns of these algicolous fungal communities is proposed to understand and model the influence of climate change on the maritime Antarctic biota. In addition, several fungal isolates from marine Antarctic environments have shown great potential as producers of bioactive natural products and enzymes and may represent attractive sources of biotechno- logical products. M. B. Ogaki · M. T. de Paula · D. Ruas · L. H. Rosa (*) Departamento de Microbiologia, Universidade Federal de Minas Gerais, Belo Horizonte, MG, Brazil e-mail: [email protected] F. -

Diversity and Co-Occurrence Patterns of Microbial Communities in the Fertilized Eggs of Thitarodes Insect, the Implication on the Occurrence of Chinese Cordyceps
Diversity and Co-Occurrence Patterns of microbial Communities in the Fertilized Eggs of Thitarodes Insect, the Implication on the Occurrence of Chinese cordyceps Lian-Xian Guo Guangdong Medical University Xiao-Shan Liu Guangdong Medical University Zhan-Hua Mai Guangdong Medical University Yue-Hui Hong Guangdong Hospital of Traditional Chinese Medicine Qi-Jiong Zhu Guangdong Medical University Xu-Ming Guo Guangdong Medical University Huan-Wen Tang ( [email protected] ) Guangdong Medical University Research article Keywords: Bacterial, Fungal, Fertilized Eggs, Thitarodes, Chinese cordyceps, Wolbachia Posted Date: April 28th, 2020 DOI: https://doi.org/10.21203/rs.3.rs-24686/v1 License: This work is licensed under a Creative Commons Attribution 4.0 International License. Read Full License Page 1/20 Abstract Background The large-scale articial cultivation of Chinese cordyceps has not been widely implemented because the crucial factors triggering the occurrence of Chinese cordyceps have not been fully illuminated. Methods In this study, the bacterial and fungal structure of fertilized eggs in the host Thitarodes collected from 3 sampling sites with different occurrence rates of Chinese cordyceps (Sites A, B and C: high, low and null Chinese cordyceps, respectively) were analyzed by performing 16S RNA and ITS sequencing, respectively. And the intra-kingdom and inter-kingdom network were analyzed. Results For bacterial community, totally 4671 bacterial OTUs were obtained. α-diversity analysis revealed that the evenness of the eggs from site A was signicantly higher than that of sites B and C, and the dominance index of site A was signicantly lower than that of sites B and C ( P < 0.05). -

Lists of Names in Aspergillus and Teleomorphs As Proposed by Pitt and Taylor, Mycologia, 106: 1051-1062, 2014 (Doi: 10.3852/14-0
Lists of names in Aspergillus and teleomorphs as proposed by Pitt and Taylor, Mycologia, 106: 1051-1062, 2014 (doi: 10.3852/14-060), based on retypification of Aspergillus with A. niger as type species John I. Pitt and John W. Taylor, CSIRO Food and Nutrition, North Ryde, NSW 2113, Australia and Dept of Plant and Microbial Biology, University of California, Berkeley, CA 94720-3102, USA Preamble The lists below set out the nomenclature of Aspergillus and its teleomorphs as they would become on acceptance of a proposal published by Pitt and Taylor (2014) to change the type species of Aspergillus from A. glaucus to A. niger. The central points of the proposal by Pitt and Taylor (2014) are that retypification of Aspergillus on A. niger will make the classification of fungi with Aspergillus anamorphs: i) reflect the great phenotypic diversity in sexual morphology, physiology and ecology of the clades whose species have Aspergillus anamorphs; ii) respect the phylogenetic relationship of these clades to each other and to Penicillium; and iii) preserve the name Aspergillus for the clade that contains the greatest number of economically important species. Specifically, of the 11 teleomorph genera associated with Aspergillus anamorphs, the proposal of Pitt and Taylor (2014) maintains the three major teleomorph genera – Eurotium, Neosartorya and Emericella – together with Chaetosartorya, Hemicarpenteles, Sclerocleista and Warcupiella. Aspergillus is maintained for the important species used industrially and for manufacture of fermented foods, together with all species producing major mycotoxins. The teleomorph genera Fennellia, Petromyces, Neocarpenteles and Neopetromyces are synonymised with Aspergillus. The lists below are based on the List of “Names in Current Use” developed by Pitt and Samson (1993) and those listed in MycoBank (www.MycoBank.org), plus extensive scrutiny of papers publishing new species of Aspergillus and associated teleomorph genera as collected in Index of Fungi (1992-2104). -
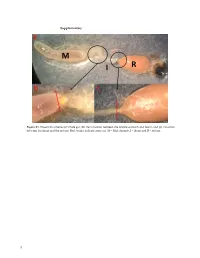
Supplementary File 1
Supplementary Figure S1. Dissection scheme (a) whole gut, (b) the transition between the middle stomach and ileum, and (c) transition between the ileum and the rectum. Red streaks indicate areas cut. M = Mid stomach, I = ileum and R = rectum. 1 Figure S2. Relative abundance of the 20 most abundant fungal OTUs across months per gut part. Taxonomic assignments are provided in Table S1. 2 Figure S3. Relative abundance of the bacterial OUTs (with abundance > 1%) across months per gut part. The color code is given at the genus level. 3 Table S1. Taxonomic assignments of fungal OTUs by Blast. OTU# Taxonomy by blast nt collection 11/11‐2020 Phylum Accesion# OTU_1 Aureobasidium pullulans Ascomycota MW085051 OTU_2 Meyerozyma guilliermondii Ascomycota MT988167 OTU_3 Starmerella apicola Ascomycota KY101940 OTU_4 Debaryomyces hansenii Ascomycota MW051606 OTU_5 Engyodontium sp. Ascomycota MN905797 Basidiomy‐ OTU_6 Mrakia sp. MT505696 cota Basidiomy‐ OTU_7 Tausonia pullulans MN900123 cota OTU_8 Unknown OTU_9 Unknown OTU_10 Penicillium corylophilum Ascomycota MT906500 OTU_11 Penicillium sp. Ascomycota MT993349 OTU_12 Cladosporium sp. Ascomycota MW077705 Phaffia rhodozyma/Xanthophyllomyces dendro‐ Basidiomy‐ OTU_13 KY104501/DQ904243 rhous cota Basidiomy‐ OTU_14 Filobasidium wieringae MN899199 cota OTU_15 Hanseniaspora uvarum Ascomycota MN556596 Basidiomy‐ OTU_16 Mrakia gelida MN460370 cota OTU_17 Uncultured fungus clone MK717974 OTU_18 Cladosporium allicinum Ascomycota MT974153 OTU_20 Taphrina carpini Ascomycota MK782181 Basidiomy‐ OTU_25 Papiliotrema -

00 PHD Beta 10
Identification and control of fungal contamination in ancient heritage documents Nuno Miguel da Costa Pinheiro Meneses Mesquita Setembro 2013 Dissertação apresentada à Universidade de Coimbra para a obtenção do grau de Doutor em Biologia, especialidade em Biologia Molecular, realizada sob a orientação científica do Professor Doutor António Manuel Santos Carriço Portugal e co-orientação da Professora Doutora Helena Maria de Oliveira Freitas do Departamento de Ciências da Vida da Faculdade de Ciências e Tecnologia da Universidade de Coimbra. This research was funded by Fundação para a Ciência e a Tecnologia (FCT) and the European Social Fund through the Programa Operacional Potencial Humano (POPH) of the National Strategic Reference Network, with the reference SFRH/BD/41287/2007. This work was also funded by Fundo Europeu de Desenvolvimento Regional (FEDER) through the Programa Operacional Fatores de Competitividade (POFC-COMPETE) and national funds, through FCT under the project with the reference PTDC/HAH/65262/2006. Acknowledgements To Fundação para a Ciência e a Tecnologia for sponsoring this work with a PhD grant (SFRH/BD/41287/2007). To my supervisor, Professor António Portugal, for his trust in having me as a PhD student; for his friendship, advice and utmost support in so many different matters. To Professor Helena Freitas, my co-supervisor, for the open-minded ideas and advice. To the Centre for Functional Ecology of the University of Coimbra, for allowing me to perform and develop my thesis. To Professor Teresa Gonçalves, for all the interesting conversations and support. To João Loureiro for his support and advice during the flow cytometry experiences. To my lab partners, in alphabetical order (to prevent malicious sample contaminations in the future): Diana, Elisa, Gabriela, Helena, Hugo, John, Sandra, Susana and Trovão. -

Phylogeny, Identification and Nomenclature of the Genus Aspergillus
available online at www.studiesinmycology.org STUDIES IN MYCOLOGY 78: 141–173. Phylogeny, identification and nomenclature of the genus Aspergillus R.A. Samson1*, C.M. Visagie1, J. Houbraken1, S.-B. Hong2, V. Hubka3, C.H.W. Klaassen4, G. Perrone5, K.A. Seifert6, A. Susca5, J.B. Tanney6, J. Varga7, S. Kocsube7, G. Szigeti7, T. Yaguchi8, and J.C. Frisvad9 1CBS-KNAW Fungal Biodiversity Centre, Uppsalalaan 8, NL-3584 CT Utrecht, The Netherlands; 2Korean Agricultural Culture Collection, National Academy of Agricultural Science, RDA, Suwon, South Korea; 3Department of Botany, Charles University in Prague, Prague, Czech Republic; 4Medical Microbiology & Infectious Diseases, C70 Canisius Wilhelmina Hospital, 532 SZ Nijmegen, The Netherlands; 5Institute of Sciences of Food Production National Research Council, 70126 Bari, Italy; 6Biodiversity (Mycology), Eastern Cereal and Oilseed Research Centre, Agriculture & Agri-Food Canada, Ottawa, ON K1A 0C6, Canada; 7Department of Microbiology, Faculty of Science and Informatics, University of Szeged, H-6726 Szeged, Hungary; 8Medical Mycology Research Center, Chiba University, 1-8-1 Inohana, Chuo-ku, Chiba 260-8673, Japan; 9Department of Systems Biology, Building 221, Technical University of Denmark, DK-2800 Kgs. Lyngby, Denmark *Correspondence: R.A. Samson, [email protected] Abstract: Aspergillus comprises a diverse group of species based on morphological, physiological and phylogenetic characters, which significantly impact biotechnology, food production, indoor environments and human health. Aspergillus was traditionally associated with nine teleomorph genera, but phylogenetic data suggest that together with genera such as Polypaecilum, Phialosimplex, Dichotomomyces and Cristaspora, Aspergillus forms a monophyletic clade closely related to Penicillium. Changes in the International Code of Nomenclature for algae, fungi and plants resulted in the move to one name per species, meaning that a decision had to be made whether to keep Aspergillus as one big genus or to split it into several smaller genera. -

Sensitization to Fungal Allergens: Resolved and Unresolved Issues
Allergology International 64 (2015) 321e331 Contents lists available at ScienceDirect Allergology International journal homepage: http://www.elsevier.com/locate/alit Invited review article Sensitization to fungal allergens: Resolved and unresolved issues * Yuma Fukutomi , Masami Taniguchi Clinical Research Center for Allergy and Rheumatology, Sagamihara National Hospital, Kanagawa, Japan article info abstract Article history: Exposure and sensitization to fungal allergens can promote the development and worsening of allergic Received 29 April 2015 diseases. Although numerous species of fungi have been associated with allergic diseases in the litera- Received in revised form ture, the significance of fungi from the genera Alternaria, Cladosporium, Penicillium, Aspergillus, and 1 May 2015 Malassezia has been well documented. However, it should be emphasized that the contribution of Accepted 7 May 2015 different fungal allergens to allergic diseases is not identical, but species-specific. Available online 9 June 2015 Alternaria and Cladosporium species are considered to be important outdoor allergens, and sensiti- zation and exposure to species of these genera is related to the development of asthma and rhinitis, as Keywords: Allergen well as epidemics of asthma exacerbation, including life-threatening asthma exacerbation. In contrast, Allergy xerophilic species of Penicillium and Aspergillus, excluding Aspergillus fumigatus, are implicated in allergic Cross-reactivity diseases as indoor allergens. A. fumigatus has a high capacity to colonize the bronchial tract of asthmatic Fungi patients, causing severe persistent asthma and low lung function, and sometimes leading to allergic Molecular-based allergy diagnostics bronchopulmonary aspergillosis. Malassezia are common commensals of healthy skin, although they are also associated with atopic dermatitis, especially on the head and neck, but not with respiratory allergies. -

Molecular Profiling of Indoor Microbial Communities in Moisture Damaged
View metadata, citation and similar papers at core.ac.uk brought to you by CORE provided by Helsingin yliopiston digitaalinen arkisto Molecular profi ling of indoor microbial communiƟ es in moisture damaged and non-damaged buildings Miia Pitkäranta Division of General Microbiology Faculty of Biological and Environmental Sciences University of Helsinki and DNA Sequencing and Genomics Laboratory InsƟ tute of Biotechnology University of Helsinki and Graduate School in Environmental Health (SYTYKE) Academic DissertaƟ on in General Microbiology To be presented for public examinaƟ on with the permission of the Faculty of Biological and Environmental Sciences of the University of Helsinki, in lecture hall Paatsama in the Animal Hospital building of University of Helsinki, on 20.1.2012 at 12 o’clock noon. Supervisors Docent Petri Auvinen Institute of Biotechnology University of Helsinki Helsinki, Finland Docent Helena Rintala Department of Environmental Health National Institute for Health and Welfare Kuopio, Finland Professor Martin Romantschuk Department of Environmental Sciences University of Helsinki Lahti, Finland Reviewers Professor Malcolm Richardson School of Medicine University of Manchester Manchester, UK Professor Kaarina Sivonen Department of Applied Microbiology and Chemistry University of Helsinki Helsinki, Finland Opponent Associate Professor James Scott Dalla Lana School of Public Health University of Toronto Toronto, Canada Custos Professor Jouko Rikkinen Department of Biosciences University of Helsinki Helsinki, Finland Layout: -

Classification of Aspergillus, Penicillium
available online at www.studiesinmycology.org STUDIES IN MYCOLOGY 95: 5–169 (2020). Classification of Aspergillus, Penicillium, Talaromyces and related genera (Eurotiales): An overview of families, genera, subgenera, sections, series and species J. Houbraken1*, S. Kocsube2, C.M. Visagie3, N. Yilmaz3, X.-C. Wang1,4, M. Meijer1, B. Kraak1, V. Hubka5, K. Bensch1, R.A. Samson1, and J.C. Frisvad6* 1Westerdijk Fungal Biodiversity Institute, Utrecht, The Netherlands; 2Department of Microbiology, Faculty of Science and Informatics, University of Szeged, Szeged, Hungary; 3Department of Biochemistry, Genetics and Microbiology, Forestry and Agricultural Biotechnology Institute (FABI), University of Pretoria, P. Bag X20, Hatfield, Pretoria, 0028, South Africa; 4State Key Laboratory of Mycology, Institute of Microbiology, Chinese Academy of Sciences, No. 3, 1st Beichen West Road, Chaoyang District, Beijing, 100101, China; 5Department of Botany, Charles University in Prague, Prague, Czech Republic; 6Department of Biotechnology and Biomedicine Technical University of Denmark, Søltofts Plads, B. 221, Kongens Lyngby, DK 2800, Denmark *Correspondence: J. Houbraken, [email protected]; J.C. Frisvad, [email protected] Abstract: The Eurotiales is a relatively large order of Ascomycetes with members frequently having positive and negative impact on human activities. Species within this order gain attention from various research fields such as food, indoor and medical mycology and biotechnology. In this article we give an overview of families and genera present in the Eurotiales and introduce an updated subgeneric, sectional and series classification for Aspergillus and Penicillium. Finally, a comprehensive list of accepted species in the Eurotiales is given. The classification of the Eurotiales at family and genus level is traditionally based on phenotypic characters, and this classification has since been challenged using sequence-based approaches. -

Phylogeny, Identification and Nomenclature of the Genus Aspergillus
Downloaded from orbit.dtu.dk on: Oct 07, 2021 Phylogeny, identification and nomenclature of the genus Aspergillus Samson, R.A.; Visagie, C.M.; Houbraken, J.; Hong, S. B.; Hubka, V.; Klaassen, C.H.W.; Perrone, G.; Seifert, K.A.; Susca, A.; Tanney, J.B. Total number of authors: 15 Published in: Studies in Mycology Link to article, DOI: 10.1016/j.simyco.2014.07.004 Publication date: 2014 Document Version Publisher's PDF, also known as Version of record Link back to DTU Orbit Citation (APA): Samson, R. A., Visagie, C. M., Houbraken, J., Hong, S. B., Hubka, V., Klaassen, C. H. W., Perrone, G., Seifert, K. A., Susca, A., Tanney, J. B., Varga, J., Kocsubé, S., Szigeti, G., Yaguchi, T., & Frisvad, J. C. (2014). Phylogeny, identification and nomenclature of the genus Aspergillus. Studies in Mycology, 78, 141-173. https://doi.org/10.1016/j.simyco.2014.07.004 General rights Copyright and moral rights for the publications made accessible in the public portal are retained by the authors and/or other copyright owners and it is a condition of accessing publications that users recognise and abide by the legal requirements associated with these rights. Users may download and print one copy of any publication from the public portal for the purpose of private study or research. You may not further distribute the material or use it for any profit-making activity or commercial gain You may freely distribute the URL identifying the publication in the public portal If you believe that this document breaches copyright please contact us providing details, and we will remove access to the work immediately and investigate your claim. -

Diversité Des Espèces De Levures Dans Des Levains Naturels Français
Diversité des espèces de levures dans des levains naturels français produits à partir de farine issue de l’Agriculture Biologique : une étude pilote pour analyser les pratiques boulangères et les patterns des communautés microbiennes Charlotte Urien To cite this version: Charlotte Urien. Diversité des espèces de levures dans des levains naturels français produits à partir de farine issue de l’Agriculture Biologique : une étude pilote pour analyser les pratiques boulangères et les patterns des communautés microbiennes. Ecologie, Environnement. Université Paris Sud - Paris XI, 2015. Français. NNT : 2015PA112007. tel-01249573 HAL Id: tel-01249573 https://tel.archives-ouvertes.fr/tel-01249573 Submitted on 4 Jan 2016 HAL is a multi-disciplinary open access L’archive ouverte pluridisciplinaire HAL, est archive for the deposit and dissemination of sci- destinée au dépôt et à la diffusion de documents entific research documents, whether they are pub- scientifiques de niveau recherche, publiés ou non, lished or not. The documents may come from émanant des établissements d’enseignement et de teaching and research institutions in France or recherche français ou étrangers, des laboratoires abroad, or from public or private research centers. publics ou privés. UNIVERSITÉ PARIS-SUD ÉCOLE DOCTORALE : Structure et Dynamique des Systèmes Vivants (anciennement ED 426 : Gènes, Génomes, Cellules) Laboratoire : UMR de Génétique Quantitative et Evolution – Le Moulon THÈSE DE DOCTORAT SCIENCES DE LA VIE ET DE LA SANTÉ par Charlotte URIEN Diversité des espèces -

Indoor Air Mycological Survey and Occupational Exposure in Libraries in Mato Grosso-Central Region—Brazil
Advances in Microbiology, 2018, 8, 324-353 http://www.scirp.org/journal/aim ISSN Online: 2165-3410 ISSN Print: 2165-3402 Indoor Air Mycological Survey and Occupational Exposure in Libraries in Mato Grosso-Central Region—Brazil Diniz Pereira Leite Júnior1,2,3, Ronaldo Sousa Pereira1, Washington Santos de Almeida1, Sara de Almeida Alves Simões1, Ana Caroline Akeme Yamamoto1, Janaina Vasconcellos Ribeiro de Souza1, Evelin Rodrigues Martins4, Fábio Alexandre Leal dos Santos1,3, Rosane Christine Hahn1,2* 1Faculty of Medicine, Post Graduate Course in Health Sciences - Federal University of Mato Grosso (UFMT), Cuiabá, MT, Brazil 2Specialized Medical Mycology Center, Federal University of Mato Grosso (UFMT), Cuiabá, MT, Brazil 3University Center of Várzea Grande (UNIVAG), Várzea Grande, MT, Brazil 4Faculty of Medicine of São José do Rio Preto (FAMERP), São Paulo, SP, Brazil How to cite this paper: Leite-Jr., D.P., Abstract Pereira, R.S., Almeida, W.S., Simões, S.A.A., Yamamoto, A.C.A., Souza, J.V.R., Martins, Background: Indoor air quality in environments where there is great circula- E.R., Leal-Santos, F.A. and Hahn, R.C. tion of people, posing risks to the health of its occupants, including allergic (2018) Indoor Air Mycological Survey and problems, infections and contaminations, can be aided by climatic factors, Occupational Exposure in Libraries in Mato Grosso-Central Region—Brazil. Advances chemicals and biological agents housed in these environments, influencing the in Microbiology, 8, 324-353. location and providing favorable conditions for the degradation of biblio- https://doi.org/10.4236/aim.2018.84022 graphic collections. The present study investigated the presence of fungi in indoor environments in seven public and private libraries in the central region Received: March 7, 2018 Accepted: April 23, 2018 of Brazil, Mato Grosso, and verified the impact on occupational health.