Coarse-Grained Modeling of the Protein Complexation Phenomena
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Molecular and Physiological Basis for Hair Loss in Near Naked Hairless and Oak Ridge Rhino-Like Mouse Models: Tracking the Role of the Hairless Gene
University of Tennessee, Knoxville TRACE: Tennessee Research and Creative Exchange Doctoral Dissertations Graduate School 5-2006 Molecular and Physiological Basis for Hair Loss in Near Naked Hairless and Oak Ridge Rhino-like Mouse Models: Tracking the Role of the Hairless Gene Yutao Liu University of Tennessee - Knoxville Follow this and additional works at: https://trace.tennessee.edu/utk_graddiss Part of the Life Sciences Commons Recommended Citation Liu, Yutao, "Molecular and Physiological Basis for Hair Loss in Near Naked Hairless and Oak Ridge Rhino- like Mouse Models: Tracking the Role of the Hairless Gene. " PhD diss., University of Tennessee, 2006. https://trace.tennessee.edu/utk_graddiss/1824 This Dissertation is brought to you for free and open access by the Graduate School at TRACE: Tennessee Research and Creative Exchange. It has been accepted for inclusion in Doctoral Dissertations by an authorized administrator of TRACE: Tennessee Research and Creative Exchange. For more information, please contact [email protected]. To the Graduate Council: I am submitting herewith a dissertation written by Yutao Liu entitled "Molecular and Physiological Basis for Hair Loss in Near Naked Hairless and Oak Ridge Rhino-like Mouse Models: Tracking the Role of the Hairless Gene." I have examined the final electronic copy of this dissertation for form and content and recommend that it be accepted in partial fulfillment of the requirements for the degree of Doctor of Philosophy, with a major in Life Sciences. Brynn H. Voy, Major Professor We have read this dissertation and recommend its acceptance: Naima Moustaid-Moussa, Yisong Wang, Rogert Hettich Accepted for the Council: Carolyn R. -

New Approach for Untangling the Role of Uncommon Calcium-Binding Proteins in the Central Nervous System
brain sciences Review New Approach for Untangling the Role of Uncommon Calcium-Binding Proteins in the Central Nervous System Krisztina Kelemen * and Tibor Szilágyi Department of Physiology, Doctoral School, Faculty of Medicine, George Emil Palade University of Medicine, Pharmacy, Science, and Technology of Targu Mures, 540142 Târgu Mures, , Romania; [email protected] * Correspondence: [email protected]; Tel.: +40-746-248064 Abstract: Although Ca2+ ion plays an essential role in cellular physiology, calcium-binding proteins (CaBPs) were long used for mainly as immunohistochemical markers of specific cell types in different regions of the central nervous system. They are a heterogeneous and wide-ranging group of proteins. Their function was studied intensively in the last two decades and a tremendous amount of informa- tion was gathered about them. Girard et al. compiled a comprehensive list of the gene-expression profiles of the entire EF-hand gene superfamily in the murine brain. We selected from this database those CaBPs which are related to information processing and/or neuronal signalling, have a Ca2+- buffer activity, Ca2+-sensor activity, modulator of Ca2+-channel activity, or a yet unknown function. In this way we created a gene function-based selection of the CaBPs. We cross-referenced these findings with publicly available, high-quality RNA-sequencing and in situ hybridization databases (Human Protein Atlas (HPA), Brain RNA-seq database and Allen Brain Atlas integrated into the HPA) and created gene expression heat maps of the regional and cell type-specific expression levels of the selected CaBPs. This represents a useful tool to predict and investigate different expression patterns and functions of the less-known CaBPs of the central nervous system. -

University of California, San Diego
UC San Diego UC San Diego Electronic Theses and Dissertations Title The post-terminal differentiation fate of RNAs revealed by next-generation sequencing Permalink https://escholarship.org/uc/item/7324r1rj Author Lefkowitz, Gloria Kuo Publication Date 2012 Peer reviewed|Thesis/dissertation eScholarship.org Powered by the California Digital Library University of California UNIVERSITY OF CALIFORNIA, SAN DIEGO The post-terminal differentiation fate of RNAs revealed by next-generation sequencing A dissertation submitted in partial satisfaction of the requirements for the degree Doctor of Philosophy in Biomedical Sciences by Gloria Kuo Lefkowitz Committee in Charge: Professor Benjamin D. Yu, Chair Professor Richard Gallo Professor Bruce A. Hamilton Professor Miles F. Wilkinson Professor Eugene Yeo 2012 Copyright Gloria Kuo Lefkowitz, 2012 All rights reserved. The Dissertation of Gloria Kuo Lefkowitz is approved, and it is acceptable in quality and form for publication on microfilm and electronically: __________________________________________________________________ __________________________________________________________________ __________________________________________________________________ __________________________________________________________________ __________________________________________________________________ Chair University of California, San Diego 2012 iii DEDICATION Ma and Ba, for your early indulgence and support. Matt and James, for choosing more practical callings. Roy, my love, for patiently sharing the ups and downs -

Transdifferentiation of Human Mesenchymal Stem Cells
Transdifferentiation of Human Mesenchymal Stem Cells Dissertation zur Erlangung des naturwissenschaftlichen Doktorgrades der Julius-Maximilians-Universität Würzburg vorgelegt von Tatjana Schilling aus San Miguel de Tucuman, Argentinien Würzburg, 2007 Eingereicht am: Mitglieder der Promotionskommission: Vorsitzender: Prof. Dr. Martin J. Müller Gutachter: PD Dr. Norbert Schütze Gutachter: Prof. Dr. Georg Krohne Tag des Promotionskolloquiums: Doktorurkunde ausgehändigt am: Hiermit erkläre ich ehrenwörtlich, dass ich die vorliegende Dissertation selbstständig angefertigt und keine anderen als die von mir angegebenen Hilfsmittel und Quellen verwendet habe. Des Weiteren erkläre ich, dass diese Arbeit weder in gleicher noch in ähnlicher Form in einem Prüfungsverfahren vorgelegen hat und ich noch keinen Promotionsversuch unternommen habe. Gerbrunn, 4. Mai 2007 Tatjana Schilling Table of contents i Table of contents 1 Summary ........................................................................................................................ 1 1.1 Summary.................................................................................................................... 1 1.2 Zusammenfassung..................................................................................................... 2 2 Introduction.................................................................................................................... 4 2.1 Osteoporosis and the fatty degeneration of the bone marrow..................................... 4 2.2 Adipose and bone -

Zimmer Cell Calcium 2013 Mammalian S100 Evolution.Pdf
Cell Calcium 53 (2013) 170–179 Contents lists available at SciVerse ScienceDirect Cell Calcium jo urnal homepage: www.elsevier.com/locate/ceca Evolution of the S100 family of calcium sensor proteins a,∗ b b,1 b Danna B. Zimmer , Jeannine O. Eubanks , Dhivya Ramakrishnan , Michael F. Criscitiello a Center for Biomolecular Therapeutics and Department of Biochemistry & Molecular Biology, University of Maryland School of Medicine, 108 North Greene Street, Baltimore, MD 20102, United States b Comparative Immunogenetics Laboratory, Department of Veterinary Pathobiology, College of Veterinary Medicine & Biomedical Sciences, Texas A&M University, College Station, TX 77843-4467, United States a r t i c l e i n f o a b s t r a c t 2+ Article history: The S100s are a large group of Ca sensors found exclusively in vertebrates. Transcriptomic and genomic Received 4 October 2012 data from the major radiations of mammals were used to derive the evolution of the mammalian Received in revised form 1 November 2012 S100s genes. In human and mouse, S100s and S100 fused-type proteins are in a separate clade from Accepted 3 November 2012 2+ other Ca sensor proteins, indicating that an ancient bifurcation between these two gene lineages Available online 14 December 2012 has occurred. Furthermore, the five genomic loci containing S100 genes have remained largely intact during the past 165 million years since the shared ancestor of egg-laying and placental mammals. Keywords: Nonetheless, interesting births and deaths of S100 genes have occurred during mammalian evolution. Mammals The S100A7 loci exhibited the most plasticity and phylogenetic analyses clarified relationships between Phylogenetic analyses the S100A7 proteins encoded in the various mammalian genomes. -

13993003.02041-2018.Full.Pdf
Early View Original article An atypical pulmonary fibrosis is associated with co-inheritance of mutations in the calcium binding protein genes S100A3 and S100A13 E.A. Al-Mutairy, F. Imtiaz, M. Khalid, S. Al Qattan, S. Saleh, L. Mahmoud, M. Al-Saif, L. Al-Haj, A. Al-Enazi, A.M. AlJebreen, S. Mohammed, A. Mobeireek, K. Alkattan, M.A. Chisti, I.G. Luzina, M. Al- Owain, I. Weheba, A. Abdelsayed, K. Ramzan, J.L. Janssen, W. Conca, A. Alaiya, K.S. Collison, B.F. Meyer, S.P. Atamas, K.S. Khabar, J.D. Hasday, F. Al-Mohanna Please cite this article as: Al-Mutairy EA, Imtiaz F, Khalid M, et al. An atypical pulmonary fibrosis is associated with co-inheritance of mutations in the calcium binding protein genes S100A3 and S100A13. Eur Respir J 2019; in press (https://doi.org/10.1183/13993003.02041- 2018). This manuscript has recently been accepted for publication in the European Respiratory Journal. It is published here in its accepted form prior to copyediting and typesetting by our production team. After these production processes are complete and the authors have approved the resulting proofs, the article will move to the latest issue of the ERJ online. Copyright ©ERS 2019 An atypical pulmonary fibrosis is associated with co-inheritance of mutations in the calcium binding protein genes S100A3 and S100A13 Al-Mutairy EA1,2,3, Imtiaz F4, Khalid M1, Al Qattan S2, Saleh S2, Mahmoud L5, Al-Saif M5, Al-Haj L5, Al-Enazi A2, AlJebreen AM6, Mohammed S7, Mobeireek A1, Alkattan K1,3, Chisti MA8, Luzina IG9,10, Al-Owain M3,11, Weheba I1,12 , Abdelsayed A1,13 -

Human S100A3 Low Endotoxin Circulex Product Data Sheet for Research Use Only, Not for Use in Diagnostic Procedures
TM Human S100A3 Low Endotoxin CircuLex Product Data Sheet For Research Use Only, Not for use in diagnostic procedures Human S100A3 Low Endotoxin Human, recombinant protein expressed in E. coli. Low Endotoxin & Sterilized Cat# CY-R2453 Amount: 100 µg ( µg/µl) Lot: Introduction: The protein S100A3, formerly called S100E, was recognized for the first time as the product of one of the tightest gene clusters discovered in the human genome located on chromosome 1q21. The S100A3 gene shows a low but general transcription level in diaphragm, heart, skeletal muscle, stomach, lung, liver, fat tissue, and placenta. S100A3 shows a remarkably narrow tissue- and cell-specific expression pattern. It is highly expressed in hair root cells and some astrocytomas. Because both cell types are characterized by high proliferation rates S100A3 is supposed to be involved in cell cycle progression. Within the S100 subfamily S100A3 is unique for the exceptionally high number of Cys residues. Despite the Cys frequency, S100A3 does not display the classical zinc-binding motifs seen in metallothioneins, DNA-binding proteins, or protein kinase C. Product Description: Full length of human S100A3, containing an N-terminal GST tag, expressed in E. coil. and purified by GSH agarose chromatography. Gene Information: The gene accession number is NM_006271. Gene Aliases: S100E Formulation: Recombinant human S100A3 is supplied frozen in 2X PBS (2X phosphate buffered saline) containing 50 % glycerol. Endotoxin Concentration: < 0.01 EU/µg as determined by Limulus Amebocyte Lysate (LAL) assay Cat#: CY-R2453 1 Version#: 120420 For Reference Purpose Only! TM Human S100A3 Low Endotoxin CircuLex Product Data Sheet For Research Use Only, Not for use in diagnostic procedures Molecular Weight: 34 kDa Recombinant human S100A3 demonstrates approximately 34 kDa band by Mw (kDa) SDS-PAGE analysis. -

Transcriptional Profile of Human Anti-Inflamatory Macrophages Under Homeostatic, Activating and Pathological Conditions
UNIVERSIDAD COMPLUTENSE DE MADRID FACULTAD DE CIENCIAS QUÍMICAS Departamento de Bioquímica y Biología Molecular I TESIS DOCTORAL Transcriptional profile of human anti-inflamatory macrophages under homeostatic, activating and pathological conditions Perfil transcripcional de macrófagos antiinflamatorios humanos en condiciones de homeostasis, activación y patológicas MEMORIA PARA OPTAR AL GRADO DE DOCTOR PRESENTADA POR Víctor Delgado Cuevas Directores María Marta Escribese Alonso Ángel Luís Corbí López Madrid, 2017 © Víctor Delgado Cuevas, 2016 Universidad Complutense de Madrid Facultad de Ciencias Químicas Dpto. de Bioquímica y Biología Molecular I TRANSCRIPTIONAL PROFILE OF HUMAN ANTI-INFLAMMATORY MACROPHAGES UNDER HOMEOSTATIC, ACTIVATING AND PATHOLOGICAL CONDITIONS Perfil transcripcional de macrófagos antiinflamatorios humanos en condiciones de homeostasis, activación y patológicas. Víctor Delgado Cuevas Tesis Doctoral Madrid 2016 Universidad Complutense de Madrid Facultad de Ciencias Químicas Dpto. de Bioquímica y Biología Molecular I TRANSCRIPTIONAL PROFILE OF HUMAN ANTI-INFLAMMATORY MACROPHAGES UNDER HOMEOSTATIC, ACTIVATING AND PATHOLOGICAL CONDITIONS Perfil transcripcional de macrófagos antiinflamatorios humanos en condiciones de homeostasis, activación y patológicas. Este trabajo ha sido realizado por Víctor Delgado Cuevas para optar al grado de Doctor en el Centro de Investigaciones Biológicas de Madrid (CSIC), bajo la dirección de la Dra. María Marta Escribese Alonso y el Dr. Ángel Luís Corbí López Fdo. Dra. María Marta Escribese -

Prediction of EF-Hand Calcium Binding Proteins and Analysis of Bacterial EF-Hand Proteins
Prediction of EF-hand Calcium Binding Proteins and Analysis of Bacterial EF-Hand Proteins Yubin Zhou, Wei Yang, Michael Kirberger, Hsiau-Wei Lee, Gayatri Ayalasomayajula, and Jenny J. Yang* Department of Chemistry, Georgia State University, Atlanta, GA 30303, USA *To whom correspondence should be addressed Jenny J. Yang Department of Chemistry, Georgia State University University Plaza, Atlanta, Georgia 30302 USA Tel: 404-651-4620 Fax: 404-651-2751 Email: [email protected] Running title: Prediction of EF-hand Ca(II)-binding proteins Key words: EF-hand; S100; pattern search; bacterial genomes; prediction; evolution 1 Abstract The EF-hand proteins with a helix-loop-helix Ca2+ binding motif are one of the largest protein families and are involved in numerous biological processes. To facilitate the understanding of the role of Ca2+ in biological systems using genomic information, we report herein our improvement on the pattern serach method for the identification of EF-hand and EF-like Ca2+-binding proteins. The canonical EF-hand patterns are modified to cater to different flanking structural elements. In addition, based on the conserved sequence of both the N- and C-terminal EF-hands within S100 and S100-like proteins, a new signature profile has been established to allow for the identification of pseudo EF-hand and S100 proteins from genomic information. The new patterns have a positive predictive value of 99% and a sensitivity of 96% for pseudo EF-hands. Furthermore, using the developed patterns, we have identified zero pseudo EF-hand motif and 467 canonical EF-hand Ca2+ binding motifs with diverse cellular functions in the bacteria genome. -
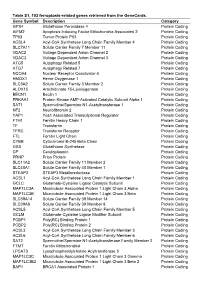
Table S1. 103 Ferroptosis-Related Genes Retrieved from the Genecards
Table S1. 103 ferroptosis-related genes retrieved from the GeneCards. Gene Symbol Description Category GPX4 Glutathione Peroxidase 4 Protein Coding AIFM2 Apoptosis Inducing Factor Mitochondria Associated 2 Protein Coding TP53 Tumor Protein P53 Protein Coding ACSL4 Acyl-CoA Synthetase Long Chain Family Member 4 Protein Coding SLC7A11 Solute Carrier Family 7 Member 11 Protein Coding VDAC2 Voltage Dependent Anion Channel 2 Protein Coding VDAC3 Voltage Dependent Anion Channel 3 Protein Coding ATG5 Autophagy Related 5 Protein Coding ATG7 Autophagy Related 7 Protein Coding NCOA4 Nuclear Receptor Coactivator 4 Protein Coding HMOX1 Heme Oxygenase 1 Protein Coding SLC3A2 Solute Carrier Family 3 Member 2 Protein Coding ALOX15 Arachidonate 15-Lipoxygenase Protein Coding BECN1 Beclin 1 Protein Coding PRKAA1 Protein Kinase AMP-Activated Catalytic Subunit Alpha 1 Protein Coding SAT1 Spermidine/Spermine N1-Acetyltransferase 1 Protein Coding NF2 Neurofibromin 2 Protein Coding YAP1 Yes1 Associated Transcriptional Regulator Protein Coding FTH1 Ferritin Heavy Chain 1 Protein Coding TF Transferrin Protein Coding TFRC Transferrin Receptor Protein Coding FTL Ferritin Light Chain Protein Coding CYBB Cytochrome B-245 Beta Chain Protein Coding GSS Glutathione Synthetase Protein Coding CP Ceruloplasmin Protein Coding PRNP Prion Protein Protein Coding SLC11A2 Solute Carrier Family 11 Member 2 Protein Coding SLC40A1 Solute Carrier Family 40 Member 1 Protein Coding STEAP3 STEAP3 Metalloreductase Protein Coding ACSL1 Acyl-CoA Synthetase Long Chain Family Member 1 Protein -

Gene Expression of Mouse S100A3, a Cysteine-Rich Calcium-Binding Protein, in Developing Hair Follicle
View metadata, citation and similar papers at core.ac.uk brought to you by CORE provided by Elsevier - Publisher Connector Gene Expression of Mouse S100A3, a Cysteine-Rich Calcium-Binding Protein, in Developing Hair Follicle Kenji Kizawa, Suguru Tsuchimoto,1 Keiko Hashimoto,2 and Hideyo Uchiwa Basic Research Laboratory, Kanebo Ltd, Odawara, Japan We have previously identified a cysteine-rich calcium layer. Expression was also observed to a lesser extent in binding protein S100A3 present in the cuticle of human differentiated cortical cells; however, expression was not hair fiber. In this study, we cloned a cDNA for mouse observed in any other component of the hair follicle or S100A3, identified its gene location, and elucidated the dorsal tissues. Immunohistochemical analysis showed expression profile throughout hair follicle development. that the S100A3 protein accumulated in cuticular and The mouse S100A3 gene was clustered with other S100 cortical cells undergoing terminal differentiation. These family members on chromosome 3, and specifically results indicate that the S100A3 gene is exclusively expressed in dorsal skin containing hair follicles. The expressed, and the translation product retained, in follicu- level of S100A3 mRNA was elevated during the anagen lar cells differentiating into major components of the phase of the hair growth cycle, and sharply declined from hair shaft. It seems likely that S100A3 plays an important the regression phase on. In situ hybridization revealed role in calcium-dependent processes leading to hair shaft that the S100A3 gene was prominently expressed in formation. Key words: cuticle/differentiation/hair cycle/S100 cuticular cells of the hair follicle, and mRNA levels were protein. -

Human Epidermal Differentiation Complex in a Single 2.5 Mbp Long Continuum of Overlapping DNA Cloned in Bacteria Integrating Physical and Transcript Maps
View metadata, citation and similar papers at core.ac.uk brought to you by CORE provided by Elsevier - Publisher Connector Human Epidermal Differentiation Complex in a Single 2.5 Mbp Long Continuum of Overlapping DNA Cloned in Bacteria Integrating Physical and Transcript Maps Andrew P. South, Adriana Cabral,* Jane H. Ives, Colin H. James, Ghazala Mirza,† Ingo Marenholz,‡ Dietmar Mischke,‡ Claude Backendorf,* Jiannis Ragoussis,† and Dean Nizetic Center for Applied Molecular Biology, School of Pharmacy, University of London, London, U.K.; *Department of Molecular Genetics, Leiden Institute of Chemistry, University of Leiden, The Netherlands; †St Thomas’ Hospitals (UMDS), Guy’s Hospital, London, U.K.; ‡Institute for Immunogenetics Charite, Humbolt University, Berlin, Germany Terminal differentiation of keratinocytes involves the duction roles in the differentiation of epidermis and sequential expression of several major proteins which other tissues. In order to provide a bacterial clone can be identified in distinct cellular layers within the resource that will enable further studies of genomic mammalian epidermis and are characteristic for the structure, transcriptional regulation, function and maturation state of the keratinocyte. Many of the evolution of the epidermal differentiation complex, as corresponding genes are clustered in one specific well as the identification of novel genes, we have human chromosomal region 1q21. It is rare in the constructed a single 2.45 Mbp long continuum of genome to find in such close proximity the genes genomic DNA cloned as 45 p1 artificial chromosomes, belonging to at least three structurally different famil- three bacterial artificial chromosomes, and 34 cosmid ies, yet sharing spatial and temporal expression speci- clones.