דוקטור לפילוסופיה Doctor of Philosophy מוגשת למועצה המדעית של Submitted to the Scientific Council of the מכון ויצמן למדע Weizmann Institute of Science
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Analysis of Trans Esnps Infers Regulatory Network Architecture
Analysis of trans eSNPs infers regulatory network architecture Anat Kreimer Submitted in partial fulfillment of the requirements for the degree of Doctor of Philosophy in the Graduate School of Arts and Sciences COLUMBIA UNIVERSITY 2014 © 2014 Anat Kreimer All rights reserved ABSTRACT Analysis of trans eSNPs infers regulatory network architecture Anat Kreimer eSNPs are genetic variants associated with transcript expression levels. The characteristics of such variants highlight their importance and present a unique opportunity for studying gene regulation. eSNPs affect most genes and their cell type specificity can shed light on different processes that are activated in each cell. They can identify functional variants by connecting SNPs that are implicated in disease to a molecular mechanism. Examining eSNPs that are associated with distal genes can provide insights regarding the inference of regulatory networks but also presents challenges due to the high statistical burden of multiple testing. Such association studies allow: simultaneous investigation of many gene expression phenotypes without assuming any prior knowledge and identification of unknown regulators of gene expression while uncovering directionality. This thesis will focus on such distal eSNPs to map regulatory interactions between different loci and expose the architecture of the regulatory network defined by such interactions. We develop novel computational approaches and apply them to genetics-genomics data in human. We go beyond pairwise interactions to define network motifs, including regulatory modules and bi-fan structures, showing them to be prevalent in real data and exposing distinct attributes of such arrangements. We project eSNP associations onto a protein-protein interaction network to expose topological properties of eSNPs and their targets and highlight different modes of distal regulation. -

Exploring Autophagy with Gene Ontology
Autophagy ISSN: 1554-8627 (Print) 1554-8635 (Online) Journal homepage: https://www.tandfonline.com/loi/kaup20 Exploring autophagy with Gene Ontology Paul Denny, Marc Feuermann, David P. Hill, Ruth C. Lovering, Helene Plun- Favreau & Paola Roncaglia To cite this article: Paul Denny, Marc Feuermann, David P. Hill, Ruth C. Lovering, Helene Plun- Favreau & Paola Roncaglia (2018) Exploring autophagy with Gene Ontology, Autophagy, 14:3, 419-436, DOI: 10.1080/15548627.2017.1415189 To link to this article: https://doi.org/10.1080/15548627.2017.1415189 © 2018 The Author(s). Published by Informa UK Limited, trading as Taylor & Francis Group. View supplementary material Published online: 17 Feb 2018. Submit your article to this journal Article views: 1097 View Crossmark data Full Terms & Conditions of access and use can be found at https://www.tandfonline.com/action/journalInformation?journalCode=kaup20 AUTOPHAGY, 2018 VOL. 14, NO. 3, 419–436 https://doi.org/10.1080/15548627.2017.1415189 RESEARCH PAPER - BASIC SCIENCE Exploring autophagy with Gene Ontology Paul Denny a,†,§, Marc Feuermann b,§, David P. Hill c,f,§, Ruth C. Lovering a,§, Helene Plun-Favreau d and Paola Roncaglia e,f,§ aFunctional Gene Annotation, Institute of Cardiovascular Science, University College London, London, UK; bSIB Swiss Institute of Bioinformatics, Geneva, Switzerland; cThe Jackson Laboratory, Bar Harbor, ME, USA; dDepartment of Molecular Neuroscience, UCL Institute of Neurology, London, UK; eEuropean Bioinformatics Institute (EMBL-EBI), European Molecular Biology Laboratory, Wellcome Genome Campus, Hinxton, Cambridge, UK; fThe Gene Ontology Consortium ABSTRACT ARTICLE HISTORY Autophagy is a fundamental cellular process that is well conserved among eukaryotes. It is one of the Received 18 May 2017 strategies that cells use to catabolize substances in a controlled way. -

Datasheet: MCA5780GA Product Details
Datasheet: MCA5780GA Description: MOUSE ANTI WIPI2 Specificity: WIPI2 Format: Purified Product Type: Monoclonal Antibody Clone: 2A2 Isotype: IgG1 Quantity: 0.1 mg Product Details Applications This product has been reported to work in the following applications. This information is derived from testing within our laboratories, peer-reviewed publications or personal communications from the originators. Please refer to references indicated for further information. For general protocol recommendations, please visit www.bio-rad-antibodies.com/protocols. Yes No Not Determined Suggested Dilution Flow Cytometry Immunohistology - Frozen Immunohistology - Paraffin ELISA Immunoprecipitation Western Blotting Immunofluorescence Where this product has not been tested for use in a particular technique this does not necessarily exclude its use in such procedures. Suggested working dilutions are given as a guide only. It is recommended that the user titrates the product for use in their own system using the appropriate negative/positive controls. Target Species Human Species Cross Reacts with: Mouse Reactivity N.B. Antibody reactivity and working conditions may vary between species. Product Form Purified IgG - liquid Buffer Solution Phosphate buffered saline Preservative 0.09% Sodium Azide (NaN ) Stabilisers 3 Carrier Free Yes Approx. Protein IgG concentration 1.0 mg/ml Concentrations Immunogen Synthetic peptide corresponding to the C-terminus of WIPI2b (CSALRLDEDSEHPPMILRTD) Page 1 of 3 External Database Links UniProt: Q9Y4P8 Related reagents Q80W47 Related reagents Entrez Gene: 26100 WIPI2 Related reagents 74781 Wipi2 Related reagents Specificity Mouse anti Human WIPI2 antibody, clone 2A2 recognies WD repeat domain phosphoinositide- interacting protein 2 (WIPI-2), also known as WIPI49-like protein 2. WIPI2 is a 454 amino acid ~54 kDa autophagosomal marker containing three WD repeats. -

NRF1) Coordinates Changes in the Transcriptional and Chromatin Landscape Affecting Development and Progression of Invasive Breast Cancer
Florida International University FIU Digital Commons FIU Electronic Theses and Dissertations University Graduate School 11-7-2018 Decipher Mechanisms by which Nuclear Respiratory Factor One (NRF1) Coordinates Changes in the Transcriptional and Chromatin Landscape Affecting Development and Progression of Invasive Breast Cancer Jairo Ramos [email protected] Follow this and additional works at: https://digitalcommons.fiu.edu/etd Part of the Clinical Epidemiology Commons Recommended Citation Ramos, Jairo, "Decipher Mechanisms by which Nuclear Respiratory Factor One (NRF1) Coordinates Changes in the Transcriptional and Chromatin Landscape Affecting Development and Progression of Invasive Breast Cancer" (2018). FIU Electronic Theses and Dissertations. 3872. https://digitalcommons.fiu.edu/etd/3872 This work is brought to you for free and open access by the University Graduate School at FIU Digital Commons. It has been accepted for inclusion in FIU Electronic Theses and Dissertations by an authorized administrator of FIU Digital Commons. For more information, please contact [email protected]. FLORIDA INTERNATIONAL UNIVERSITY Miami, Florida DECIPHER MECHANISMS BY WHICH NUCLEAR RESPIRATORY FACTOR ONE (NRF1) COORDINATES CHANGES IN THE TRANSCRIPTIONAL AND CHROMATIN LANDSCAPE AFFECTING DEVELOPMENT AND PROGRESSION OF INVASIVE BREAST CANCER A dissertation submitted in partial fulfillment of the requirements for the degree of DOCTOR OF PHILOSOPHY in PUBLIC HEALTH by Jairo Ramos 2018 To: Dean Tomás R. Guilarte Robert Stempel College of Public Health and Social Work This dissertation, Written by Jairo Ramos, and entitled Decipher Mechanisms by Which Nuclear Respiratory Factor One (NRF1) Coordinates Changes in the Transcriptional and Chromatin Landscape Affecting Development and Progression of Invasive Breast Cancer, having been approved in respect to style and intellectual content, is referred to you for judgment. -

WIPI2 Antibody A
Revision 1 C 0 2 - t WIPI2 Antibody a e r o t S Orders: 877-616-CELL (2355) [email protected] Support: 877-678-TECH (8324) 7 6 Web: [email protected] 5 www.cellsignal.com 8 # 3 Trask Lane Danvers Massachusetts 01923 USA For Research Use Only. Not For Use In Diagnostic Procedures. Applications: Reactivity: Sensitivity: MW (kDa): Source: UniProt ID: Entrez-Gene Id: WB H M R Endogenous 49 Rabbit Q9Y4P8 26100 Product Usage Information positive autophagosomes during autophagy; this translocation may be used as an autophagy marker (12). Application Dilution 1. Reggiori, F. and Klionsky, D.J. (2002) Eukaryot Cell 1, 11-21. 2. Codogno, P. and Meijer, A.J. (2005) Cell Death Differ 12 Suppl 2, 1509-18. Western Blotting 1:1000 3. Levine, B. and Yuan, J. (2005) J Clin Invest 115, 2679-88. 4. Corvera, S. (2001) Traffic 2, 859-66. Storage 5. Yan, Y. and Backer, J.M. (2007) Biochem Soc Trans 35, 239-41. 6. Krick, R. et al. (2006) FEBS Lett 580, 4632-8. Supplied in 10 mM sodium HEPES (pH 7.5), 150 mM NaCl, 100 µg/ml BSA and 50% 7. Strømhaug, P.E. et al. (2004) Mol Biol Cell 15, 3553-66. glycerol. Store at –20°C. Do not aliquot the antibody. 8. Obara, K. et al. (2008) J Biol Chem 283, 23972-80. 9. Jeffries, T.R. et al. (2004) Mol Biol Cell 15, 2652-63. Specificity / Sensitivity 10. Proikas-Cezanne, T. et al. (2007) FEBS Lett 581, 3396-404. 11. Polson, H.E. et al. -

The Emerging Roles of Mtorc1 in Macromanaging Autophagy
cancers Review The Emerging Roles of mTORC1 in Macromanaging Autophagy Akpedje S. Dossou and Alakananda Basu * Department of Microbiology, Immunology and Genetics, University of North Texas Health Science Center, Fort Worth, TX 76107, USA; [email protected] * Correspondence: [email protected]; Tel.: +1-817-735-2487 Received: 25 August 2019; Accepted: 23 September 2019; Published: 24 September 2019 Abstract: Autophagy is a process of self-degradation that enables the cell to survive when faced with starvation or stressful conditions. The mechanistic target of rapamycin (mTOR), also known as the mammalian target of rapamycin, plays a critical role in maintaining a balance between cellular anabolism and catabolism. mTOR complex 1 (mTORC1) was unveiled as a master regulator of autophagy since inhibition of mTORC1 was required to initiate the autophagy process. Evidence has emerged in recent years to indicate that mTORC1 also directly regulates the subsequent steps of the autophagy process, including the nucleation, autophagosome elongation, autophagosome maturation and termination. By phosphorylating select protein targets of the autophagy core machinery and/or their regulators, mTORC1 can alter their functions, increase their proteasomal degradation or modulate their acetylation status, which is a key switch of the autophagy process. Moreover, it phosphorylates and alters the subcellular localization of transcription factors to suppress the expression of genes needed for autophagosome formation and lysosome biogenesis. The purpose of this review article is to critically analyze current literatures to provide an integrated view of how mTORC1 regulates various steps of the autophagy process. Keywords: macroautophagy; autophagy regulation; mTORC1 substrates; AMPK; ULK1; autophagy initiation; nucleation; elongation; autophagosome maturation; transcriptional regulation 1. -

Whole-Exome Sequencing Points to Considerable Genetic Heterogeneity of Cerebral Palsy
Molecular Psychiatry (2015) 20, 176–182 © 2015 Macmillan Publishers Limited All rights reserved 1359-4184/15 www.nature.com/mp IMMEDIATE COMMUNICATION Whole-exome sequencing points to considerable genetic heterogeneity of cerebral palsy G McMichael1, MN Bainbridge2, E Haan3,4, M Corbett1,4, A Gardner1,4, S Thompson4,5, BWM van Bon3,6, CL van Eyk1, J Broadbent1, C Reynolds1,MEO’Callaghan1, LS Nguyen4, DL Adelson7, R Russo8, S Jhangiani2, H Doddapaneni2, DM Muzny2, RA Gibbs2, J Gecz1,4,9,10 and AH MacLennan1,9,10 Cerebral palsy (CP) is a common, clinically heterogeneous group of disorders affecting movement and posture. Its prevalence has changed little in 50 years and the causes remain largely unknown. The genetic contribution to CP causation has been predicted to be ~ 2%. We performed whole-exome sequencing of 183 cases with CP including both parents (98 cases) or one parent (67 cases) and 18 singleton cases (no parental DNA). We identified and validated 61 de novo protein-altering variants in 43 out of 98 (44%) case-parent trios. Initial prioritization of variants for causality was by mutation type, whether they were known or predicted to be deleterious and whether they occurred in known disease genes whose clinical spectrum overlaps CP. Further, prioritization used two multidimensional frameworks—the Residual Variation Intolerance Score and the Combined Annotation-dependent Depletion score. Ten de novo mutations in three previously identified disease genes (TUBA1A (n =2),SCN8A (n =1)andKDM5C (n = 1)) and in six novel candidate CP genes (AGAP1, JHDM1D, MAST1, NAA35, RFX2 and WIPI2) were predicted to be potentially pathogenic for CP. -

393LN V 393P 344SQ V 393P Probe Set Entrez Gene
393LN v 393P 344SQ v 393P Entrez fold fold probe set Gene Gene Symbol Gene cluster Gene Title p-value change p-value change chemokine (C-C motif) ligand 21b /// chemokine (C-C motif) ligand 21a /// chemokine (C-C motif) ligand 21c 1419426_s_at 18829 /// Ccl21b /// Ccl2 1 - up 393 LN only (leucine) 0.0047 9.199837 0.45212 6.847887 nuclear factor of activated T-cells, cytoplasmic, calcineurin- 1447085_s_at 18018 Nfatc1 1 - up 393 LN only dependent 1 0.009048 12.065 0.13718 4.81 RIKEN cDNA 1453647_at 78668 9530059J11Rik1 - up 393 LN only 9530059J11 gene 0.002208 5.482897 0.27642 3.45171 transient receptor potential cation channel, subfamily 1457164_at 277328 Trpa1 1 - up 393 LN only A, member 1 0.000111 9.180344 0.01771 3.048114 regulating synaptic membrane 1422809_at 116838 Rims2 1 - up 393 LN only exocytosis 2 0.001891 8.560424 0.13159 2.980501 glial cell line derived neurotrophic factor family receptor alpha 1433716_x_at 14586 Gfra2 1 - up 393 LN only 2 0.006868 30.88736 0.01066 2.811211 1446936_at --- --- 1 - up 393 LN only --- 0.007695 6.373955 0.11733 2.480287 zinc finger protein 1438742_at 320683 Zfp629 1 - up 393 LN only 629 0.002644 5.231855 0.38124 2.377016 phospholipase A2, 1426019_at 18786 Plaa 1 - up 393 LN only activating protein 0.008657 6.2364 0.12336 2.262117 1445314_at 14009 Etv1 1 - up 393 LN only ets variant gene 1 0.007224 3.643646 0.36434 2.01989 ciliary rootlet coiled- 1427338_at 230872 Crocc 1 - up 393 LN only coil, rootletin 0.002482 7.783242 0.49977 1.794171 expressed sequence 1436585_at 99463 BB182297 1 - up 393 -

WIPI2 Depletion Inhibits the Growth of Hepatocellular Carcinoma Cells Through the AMPK Signaling Pathway
ONCOLOGY REPORTS 43: 1467-1478, 2020 WIPI2 depletion inhibits the growth of hepatocellular carcinoma cells through the AMPK signaling pathway CHUNSHENG LIU1*, FENG LI2*, XIAONING LI1, MINGGANG CAO3, GANG FENG1, XIANWEN YUAN4,5 and XIAOLEI SHI4 1Department of Laboratory Medicine, Yijishan Hospital of Wannan Medical College, Wuhu, Anhui 241000; 2Department of Gastroenterology, The First People's Hospital of Anqing, Anqing, Anhui 246000; 3Department of Medicine, Anhui College of Traditional Chinese Medicine, Wuhu, Anhui 241000; 4Department of Hepatobiliary Surgery, The Affiliated Drum Tower Hospital of Nanjing University Medical School, Nanjing, Jiangsu 210008; 5State Key Laboratory of Pharmaceutical Biotechnology, Jiangsu Key Laboratory of Molecular Medicine and School of Medicine, Nanjing University, Nanjing, Jiangsu 210093, P.R. China Received November 4, 2019; Accepted February 12, 2020 DOI: 10.3892/or.2020.7531 Abstract. WD-repeat domain phosphoinositide-interacting both in HCC Huh7 and Hep3B cells. In order to explore the protein 2 (WIPI2) is a protein that regulates the assembly mechanisms of WIPI2 in HCC, WIPI2 was depleted in HCC of multiprotein complexes by presenting a beta-propeller cell lines and a gene microarray was constructed. The bioinfor- platform for simultaneous and reversible protein-protein matic analysis showed that WIPI2 regulated the proliferation interactions. This study was designed to investigate the asso- of HCC cells mainly through the AMPK signaling pathway. ciation between the expression of WIPI2 and the growth of Further analysis indicated that the downstream factors of the hepatocellular carcinoma (HCC). Publicly-available data from AMPK signaling pathway were downregulated after WIPI2 the UALCAN platform revealed that WIPI2 is upregulated depletion. -
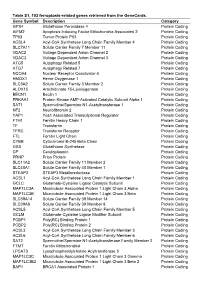
Table S1. 103 Ferroptosis-Related Genes Retrieved from the Genecards
Table S1. 103 ferroptosis-related genes retrieved from the GeneCards. Gene Symbol Description Category GPX4 Glutathione Peroxidase 4 Protein Coding AIFM2 Apoptosis Inducing Factor Mitochondria Associated 2 Protein Coding TP53 Tumor Protein P53 Protein Coding ACSL4 Acyl-CoA Synthetase Long Chain Family Member 4 Protein Coding SLC7A11 Solute Carrier Family 7 Member 11 Protein Coding VDAC2 Voltage Dependent Anion Channel 2 Protein Coding VDAC3 Voltage Dependent Anion Channel 3 Protein Coding ATG5 Autophagy Related 5 Protein Coding ATG7 Autophagy Related 7 Protein Coding NCOA4 Nuclear Receptor Coactivator 4 Protein Coding HMOX1 Heme Oxygenase 1 Protein Coding SLC3A2 Solute Carrier Family 3 Member 2 Protein Coding ALOX15 Arachidonate 15-Lipoxygenase Protein Coding BECN1 Beclin 1 Protein Coding PRKAA1 Protein Kinase AMP-Activated Catalytic Subunit Alpha 1 Protein Coding SAT1 Spermidine/Spermine N1-Acetyltransferase 1 Protein Coding NF2 Neurofibromin 2 Protein Coding YAP1 Yes1 Associated Transcriptional Regulator Protein Coding FTH1 Ferritin Heavy Chain 1 Protein Coding TF Transferrin Protein Coding TFRC Transferrin Receptor Protein Coding FTL Ferritin Light Chain Protein Coding CYBB Cytochrome B-245 Beta Chain Protein Coding GSS Glutathione Synthetase Protein Coding CP Ceruloplasmin Protein Coding PRNP Prion Protein Protein Coding SLC11A2 Solute Carrier Family 11 Member 2 Protein Coding SLC40A1 Solute Carrier Family 40 Member 1 Protein Coding STEAP3 STEAP3 Metalloreductase Protein Coding ACSL1 Acyl-CoA Synthetase Long Chain Family Member 1 Protein -

Mouse Wipi2 Conditional Knockout Project (CRISPR/Cas9)
https://www.alphaknockout.com Mouse Wipi2 Conditional Knockout Project (CRISPR/Cas9) Objective: To create a Wipi2 conditional knockout Mouse model (C57BL/6J) by CRISPR/Cas-mediated genome engineering. Strategy summary: The Wipi2 gene (NCBI Reference Sequence: NM_178398 ; Ensembl: ENSMUSG00000029578 ) is located on Mouse chromosome 5. 12 exons are identified, with the ATG start codon in exon 1 and the TGA stop codon in exon 12 (Transcript: ENSMUST00000036872). Exon 2 will be selected as conditional knockout region (cKO region). Deletion of this region should result in the loss of function of the Mouse Wipi2 gene. To engineer the targeting vector, homologous arms and cKO region will be generated by PCR using BAC clone RP23-324G18 as template. Cas9, gRNA and targeting vector will be co-injected into fertilized eggs for cKO Mouse production. The pups will be genotyped by PCR followed by sequencing analysis. Note: Exon 2 starts from about 5.62% of the coding region. The knockout of Exon 2 will result in frameshift of the gene. The size of intron 1 for 5'-loxP site insertion: 8369 bp, and the size of intron 2 for 3'-loxP site insertion: 17563 bp. The size of effective cKO region: ~583 bp. The cKO region does not have any other known gene. Page 1 of 7 https://www.alphaknockout.com Overview of the Targeting Strategy Wildtype allele gRNA region 5' gRNA region 3' 1 2 12 Targeting vector Targeted allele Constitutive KO allele (After Cre recombination) Legends Exon of mouse Wipi2 Homology arm cKO region loxP site Page 2 of 7 https://www.alphaknockout.com Overview of the Dot Plot Window size: 10 bp Forward Reverse Complement Sequence 12 Note: The sequence of homologous arms and cKO region is aligned with itself to determine if there are tandem repeats. -

Large-Scale Inference of Gene Function Through Phylogenetic Annotation Of
Database, 2016, 1–11 doi: 10.1093/database/baw155 Original article Original article Large-scale inference of gene function through phylogenetic annotation of Gene Ontology terms: case study of the apoptosis and autophagy cellular processes Marc Feuermann1,†, Pascale Gaudet2,*,†, Huaiyu Mi3, Suzanna E. Lewis4 and Paul D. Thomas3 1Swiss-Prot group, 2CALIPHO group, SIB Swiss Institute of Bioinformatics, Centre Medical Universitaire, Switzerland 1 rue Michel Servet, 1211 Geneva 4, Switzerland, 3Division of Bioinformatics, Department of Preventive Medicine, Keck School of Medicine of USC, University of Southern California, Los Angeles, CA, USA and 4Lawrence Berkeley National Laboratory, Genomics Division, Berkeley, CA, USA *Corresponding author: Email: [email protected] †These authors contributed equally to this work. Correspondence may also be addressed to Paul D. Thomas. Email: [email protected] Citation details: Feuermann,M., Gaudet,P., Mi,H. et al. Large-scale inference of gene function through phylogenetic anno- tation of gene ontology terms: case study of the apoptosis and autophagy cellular processes. Database (2016) Vol. 2016: article ID baw155; doi:10.1093/database/baw155. Received 26 July 2016; Revised 10 October 2016; Accepted 1 November 2016 Abstract We previously reported a paradigm for large-scale phylogenomic analysis of gene fami- lies that takes advantage of the large corpus of experimentally supported Gene Ontology (GO) annotations. This ‘GO Phylogenetic Annotation’ approach integrates GO annota- tions from evolutionarily related genes across 100 different organisms in the context of a gene family tree, in which curators build an explicit model of the evolution of gene functions. GO Phylogenetic Annotation models the gain and loss of functions in a gene family tree, which is used to infer the functions of uncharacterized (or incompletely char- acterized) gene products, even for human proteins that are relatively well studied.