André Guollo
Total Page:16
File Type:pdf, Size:1020Kb

Load more
Recommended publications
-

Mirnas Generated from Meg3-Mirg Locus Are Downregulated During Aging
www.aging-us.com AGING 2021, Vol. 13, No. 12 Research Paper miRNAs generated from Meg3-Mirg locus are downregulated during aging Ana-Mihaela Lupan1,*, Evelyn-Gabriela Rusu1,*, Mihai Bogdan Preda1, Catalina Iolanda Marinescu1, Cristina Ivan2, Alexandrina Burlacu1 1Laboratory of Stem Cell Biology, Institute of Cellular Biology and Pathology “Nicolae Simionescu”, Bucharest 050568, Romania 2Department of Experimental Therapeutics, Division of Basic Science Research, The University of Texas MD Anderson Cancer Center, Houston, TX 77030, USA *Equal contribution Correspondence to: Alexandrina Burlacu; email: [email protected] Keywords: aging, miRNA, Meg3-Mirg locus, cardiac fibroblasts, heart ventricles Received: October 31, 2020 Accepted: June 2, 2021 Published: June 22, 2021 Copyright: © 2021 Lupan et al. This is an open access article distributed under the terms of the Creative Commons Attribution License (CC BY 3.0), which permits unrestricted use, distribution, and reproduction in any medium, provided the original author and source are credited. ABSTRACT Aging determines a multilevel functional decline and increases the risk for cardiovascular pathologies. MicroRNAs are recognized as fine tuners of all cellular functions, being involved in various cardiac diseases. The heart is one of the most affected organs in aged individuals, however little is known about the extent and robustness to which miRNA profiles are modulated in cardiac cells during aging. This paper provides a comprehensive characterization of the aging-associated miRNA profile in the murine cardiac fibroblasts, which are increasingly recognized for their active involvement in the cardiac physiology and pathology. Next- generation sequencing of cardiac fibroblasts isolated from young and old mice revealed that an important fraction of the miRNAs generated by the Meg3-Mirg locus was downregulated during aging. -

Hypopituitarism May Be an Additional Feature of SIM1 and POU3F2 Containing 6Q16 Deletions in Children with Early Onset Obesity
Open Access Journal of Pediatric Endocrinology Case Report Hypopituitarism may be an Additional Feature of SIM1 and POU3F2 Containing 6q16 Deletions in Children with Early Onset Obesity Rutteman B1, De Rademaeker M2, Gies I1, Van den Bogaert A2, Zeevaert R1, Vanbesien J1 and De Abstract Schepper J1* Over the past decades, 6q16 deletions have become recognized as a 1Department of Pediatric Endocrinology, UZ Brussel, frequent cause of the Prader-Willi-like syndrome. Involvement of SIM1 in the Belgium deletion has been linked to the development of obesity. Although SIM1 together 2Department of Genetics, UZ Brussel, Belgium with POU3F2, which is located close to the SIM1 locus, are involved in pituitary *Corresponding author: De Schepper J, Department development and function, pituitary dysfunction has not been reported frequently of Pediatric Endocrinology, UZ Brussel, Belgium in cases of 6q16.1q16.3 deletion involving both genes. Here we report on a case of a girl with typical Prader-Willi-like symptoms including early-onset Received: December 23, 2016; Accepted: February 21, hyperphagic obesity, hypotonia, short hands and feet and neuro-psychomotor 2017; Published: February 22, 2017 development delay. Furthermore, she suffered from central diabetes insipidus, central hypothyroidism and hypocortisolism. Her genetic defect is a 6q16.1q16.3 deletion, including SIM1 and POU3F2. We recommend searching for a 6q16 deletion in children with early onset hyperphagic obesity associated with biological signs of hypopituitarism and/or polyuria-polydipsia syndrome. The finding of a combined SIM1 and POU3F2 deletion should prompt monitoring for the development of hypopituitarism, if not already present at diagnosis. Keywords: 6q16 deletion; SIM1; POU3F2; Prader-Willi-like syndrome; Hypopituitarism Introduction insipidus and of some specific pituitary hormone deficiencies in children with a combined SIM1 and POU3F2 deficiency. -

High-Throughput Sequencing of Human Plasma RNA by Using Thermostable Group II Intron Reverse Transcriptases
Downloaded from rnajournal.cshlp.org on September 27, 2021 - Published by Cold Spring Harbor Laboratory Press High-throughput sequencing of human plasma RNA by using thermostable group II intron reverse transcriptases YIDAN QIN,1,2,3 JUN YAO,1,2,3 DOUGLAS C. WU,1,2 RYAN M. NOTTINGHAM,1,2 SABINE MOHR,1,2 SCOTT HUNICKE-SMITH,1 and ALAN M. LAMBOWITZ1,2 1Institute for Cellular and Molecular Biology, University of Texas at Austin, Austin, Texas 78712, USA 2Department of Molecular Biosciences, University of Texas at Austin, Austin, Texas 78712, USA ABSTRACT Next-generation RNA-sequencing (RNA-seq) has revolutionized transcriptome profiling, gene expression analysis, and RNA-based diagnostics. Here, we developed a new RNA-seq method that exploits thermostable group II intron reverse transcriptases (TGIRTs) and used it to profile human plasma RNAs. TGIRTs have higher thermostability, processivity, and fidelity than conventional reverse transcriptases, plus a novel template-switching activity that can efficiently attach RNA-seq adapters to target RNA sequences without RNA ligation. The new TGIRT-seq method enabled construction of RNA-seq libraries from <1 ng of plasma RNA in <5 h. TGIRT-seq of RNA in 1-mL plasma samples from a healthy individual revealed RNA fragments mapping to a diverse population of protein-coding gene and long ncRNAs, which are enriched in intron and antisense sequences, as well as nearly all known classes of small ncRNAs, some of which have never before been seen in plasma. Surprisingly, many of the small ncRNA species were present as full-length transcripts, suggesting that they are protected from plasma RNases in ribonucleoprotein (RNP) complexes and/or exosomes. -

Molecular Reprogramming in Tomato Pollen During Development And
Molecular reprogramming in tomato pollen during development and heat stress Dissertation zur Erlangung des Doktorgrades der Naturwissenschaften vorgelegt beim Fachbereich Biowissenschaften (FB15) der Goethe-Universität Frankfurt am Main von Mario Keller geboren in Frankfurt am Main (Hessen) Frankfurt am Main 2018 (D30) Vom Fachbereich Biowissenschaften (FB15) der Goethe-Universität als Dissertation angenommen. Dekan: Prof. Dr. Sven Klimpel Gutachter: Prof. Dr. Enrico Schleiff, Jun. Prof. Dr. Michaela Müller-McNicoll Datum der Disputation: 25.03.2019 Index of contents Index of contents Index of contents ....................................................................................................................................... i Index of figures ........................................................................................................................................ iii Index of tables ......................................................................................................................................... iv Index of supplemental material ................................................................................................................ v Index of supplemental figures ............................................................................................................... v Index of supplemental tables ................................................................................................................ v Abbreviations .......................................................................................................................................... -
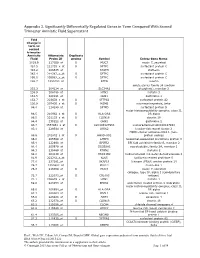
Appendix 2. Significantly Differentially Regulated Genes in Term Compared with Second Trimester Amniotic Fluid Supernatant
Appendix 2. Significantly Differentially Regulated Genes in Term Compared With Second Trimester Amniotic Fluid Supernatant Fold Change in term vs second trimester Amniotic Affymetrix Duplicate Fluid Probe ID probes Symbol Entrez Gene Name 1019.9 217059_at D MUC7 mucin 7, secreted 424.5 211735_x_at D SFTPC surfactant protein C 416.2 206835_at STATH statherin 363.4 214387_x_at D SFTPC surfactant protein C 295.5 205982_x_at D SFTPC surfactant protein C 288.7 1553454_at RPTN repetin solute carrier family 34 (sodium 251.3 204124_at SLC34A2 phosphate), member 2 238.9 206786_at HTN3 histatin 3 161.5 220191_at GKN1 gastrokine 1 152.7 223678_s_at D SFTPA2 surfactant protein A2 130.9 207430_s_at D MSMB microseminoprotein, beta- 99.0 214199_at SFTPD surfactant protein D major histocompatibility complex, class II, 96.5 210982_s_at D HLA-DRA DR alpha 96.5 221133_s_at D CLDN18 claudin 18 94.4 238222_at GKN2 gastrokine 2 93.7 1557961_s_at D LOC100127983 uncharacterized LOC100127983 93.1 229584_at LRRK2 leucine-rich repeat kinase 2 HOXD cluster antisense RNA 1 (non- 88.6 242042_s_at D HOXD-AS1 protein coding) 86.0 205569_at LAMP3 lysosomal-associated membrane protein 3 85.4 232698_at BPIFB2 BPI fold containing family B, member 2 84.4 205979_at SCGB2A1 secretoglobin, family 2A, member 1 84.3 230469_at RTKN2 rhotekin 2 82.2 204130_at HSD11B2 hydroxysteroid (11-beta) dehydrogenase 2 81.9 222242_s_at KLK5 kallikrein-related peptidase 5 77.0 237281_at AKAP14 A kinase (PRKA) anchor protein 14 76.7 1553602_at MUCL1 mucin-like 1 76.3 216359_at D MUC7 mucin 7, -

Karla Alejandra Vizcarra Zevallos Análise Da Função De Genes
Karla Alejandra Vizcarra Zevallos Análise da função de genes candidatos à manutenção da inativação do cromossomo X em humanos Dissertação apresentada ao Pro- grama de Pós‐Graduação Inter- unidades em Biotecnologia USP/ Instituto Butantan/ IPT, para obtenção do Título de Mestre em Ciências. São Paulo 2017 Karla Alejandra Vizcarra Zevallos Análise da função de genes candidatos à manutenção da inativação do cromossomo X em humanos Dissertação apresentada ao Pro- grama de Pós‐Graduação Inter- unidades em Biotecnologia do Instituto de Ciências Biomédicas USP/ Instituto Butantan/ IPT, para obtenção do Título de Mestre em Ciências. Área de concentração: Biotecnologia Orientadora: Profa. Dra. Lygia da Veiga Pereira Carramaschi Versão corrigida. A versão original eletrônica encontra-se disponível tanto na Biblioteca do ICB quanto na Biblioteca Digital de Teses e Dissertações da USP (BDTD) São Paulo 2017 UNIVERSIDADE DE SÃO PAULO Programa de Pós-Graduação Interunidades em Biotecnologia Universidade de São Paulo, Instituto Butantan, Instituto de Pesquisas Tecnológicas Candidato(a): Karla Alejandra Vizcarra Zevallos Título da Dissertação: Análise da função de genes candidatos à manutenção da inativação do cromossomo X em humanos Orientador: Profa. Dra. Lygia da Veiga Pereira Carramaschi A Comissão Julgadora dos trabalhos de Defesa da Dissertação de Mestrado, em sessão pública realizada a ........./......../.........., considerou o(a) candidato(a): ( ) Aprovado(a) ( ) Reprovado(a) Examinador(a): Assinatura: .............................................................................. -

Full Text (PDF)
Published OnlineFirst November 11, 2014; DOI: 10.1158/0008-5472.CAN-14-1041 Cancer Tumor and Stem Cell Biology Research Loss of Estrogen-Regulated microRNA Expression Increases HER2 Signaling and Is Prognostic of Poor Outcome in Luminal Breast Cancer Shannon T. Bailey1,2,3, Thomas Westerling1,2,3, and Myles Brown1,2,3 Abstract Among the genes regulated by estrogen receptor (ER) are Genome Atlas (TCGA). Among luminal tumors, these miRNAs miRNAs that play a role in breast cancer signaling pathways. To are expressed at higher levels in luminal A versus B tumors. determine whether miRNAs are involved in ER-positive breast Although their expression is uniformly low in luminal B tumors, cancer progression to hormone independence, we profiled the they are lost only in a subset of luminal A patients. Interestingly, expression of 800 miRNAs in the estrogen-dependent human this subset with low expression of these miRNAs had worse breast cancer cell line MCF7 and its estrogen-independent deriv- overall survival compared with luminal A patients with high ative MCF7:2A (MCF7:2A) using NanoString. We found 78 expression. We confirmed that miR125b directly targets HER2 miRNAs differentially expressed between the two cell lines, and that let-7c also regulates HER2 protein expression. In addi- including a cluster comprising let-7c, miR99a, and miR125b, tion, HER2 protein expression and activity are negatively corre- which is encoded in an intron of the long noncoding RNA lated with let-7c expression in TCGA. In summary, we identified LINC00478. These miRNAs are ER targets in MCF7 cells, and an ER-regulated miRNA cluster that regulates HER2, is lost with nearby ER binding and their expression are significantly progression to estrogen independence, and may serve as a bio- þ decreased in MCF7:2A cells. -

Micrornas in Prion Diseases—From Molecular Mechanisms to Insights in Translational Medicine
cells Review MicroRNAs in Prion Diseases—From Molecular Mechanisms to Insights in Translational Medicine Danyel Fernandes Contiliani 1,2, Yasmin de Araújo Ribeiro 1,2, Vitor Nolasco de Moraes 1,2 and Tiago Campos Pereira 1,2,* 1 Graduate Program of Genetics, Department of Genetics, Faculty of Medicine of Ribeirao Preto, University of Sao Paulo, Av. Bandeirantes, Ribeirao Preto 3900, Brazil; [email protected] (D.F.C.); [email protected] (Y.d.A.R.); [email protected] (V.N.d.M.) 2 Department of Biology, Faculty of Philosophy, Sciences and Letters, University of Sao Paulo, Av. Bandeirantes, Ribeirao Preto 3900, Brazil * Correspondence: [email protected]; Tel.: +55-16-3315-3818 Abstract: MicroRNAs (miRNAs) are small non-coding RNA molecules able to post-transcriptionally regulate gene expression via base-pairing with partially complementary sequences of target tran- scripts. Prion diseases comprise a singular group of neurodegenerative conditions caused by endoge- nous, misfolded pathogenic (prion) proteins, associated with molecular aggregates. In humans, classical prion diseases include Creutzfeldt–Jakob disease, fatal familial insomnia, Gerstmann– Sträussler–Scheinker syndrome, and kuru. The aim of this review is to present the connections between miRNAs and prions, exploring how the interaction of both molecular actors may help understand the susceptibility, onset, progression, and pathological findings typical of such disorders, as well as the interface with some prion-like disorders, such as Alzheimer’s. Additionally, due to the inter-regulation of prions and miRNAs in health and disease, potential biomarkers for non-invasive miRNA-based diagnostics, as well as possible miRNA-based therapies to restore the levels of dereg- Citation: Contiliani, D.F.; Ribeiro, Y.d.A.; de Moraes, V.N.; Pereira, T.C. -

Improving the Detection of Genomic Rearrangements in Short Read Sequencing Data
Improving the detection of genomic rearrangements in short read sequencing data Daniel Lee Cameron orcid.org/0000-0002-0951-7116 Doctor of Philosophy July 2017 Department of Medical Biology, University of Melbourne Bioinformatics Division, Walter and Eliza Hall Institute of Medical Research Submitted in total fulfilment of the requirements of the degree of Doctor of Philosophy Produced on archival quality paper Page i Abstract Genomic rearrangements, also known as structural variants, play a significant role in the development of cancer and in genetic disorders. With the bulk of genetic studies focusing on single nucleotide variants and small insertions and deletions, structural variants are often overlooked. In part this is due to the increased complexity of analysis required, but is also due to the increased difficulty of detection. The identification of genomic rearrangements using massively parallel sequencing remains a major challenge. To address this, I have developed the Genome Rearrangement Identification Software Suite (GRIDSS). In this thesis, I show that high sensitivity and specificity can be achieved by performing reference-guided assembly prior to variant calling and incorporating the results of this assembly into the variant calling process itself. Utilising a novel genome-wide break-end assembly approach, GRIDSS halves the false discovery rate compared to other recent methods on human cell line data. A comparison of assembly approaches reveals that incorporating read alignment information into a positional de Bruijn graph improves assembly quality compared to traditional de Bruijn graph assembly. To characterise the performance of structural variant calling software, I have performed the most comprehensive benchmarking of structural variant calling software to date. -

Timo Lassmann, Yoshiko Maida 2, Yasuhiro Tomaru, Mami Yasukawa
Int. J. Mol. Sci. 2015, 16, 1192-1208; doi:10.3390/ijms16011192 OPEN ACCESS International Journal of Molecular Sciences ISSN 1422-0067 www.mdpi.com/journal/ijms Article Telomerase Reverse Transcriptase Regulates microRNAs Timo Lassmann 1,†, Yoshiko Maida 2,†, Yasuhiro Tomaru 1, Mami Yasukawa 2, Yoshinari Ando 1, Miki Kojima 1, Vivi Kasim 2, Christophe Simon 1, Carsten O. Daub 1, Piero Carninci 1, Yoshihide Hayashizaki 1,* and Kenkichi Masutomi 2,3,* 1 RIKEN Omics Science Center, RIKEN Yokohama Institute, 1-7-22 Suehiro-cho, Tsurumi-ku, Yokohama 230-0045, Japan; E-Mails: [email protected] (T.L.); [email protected] (Y.T.); [email protected] (Y.A.); [email protected] (M.K.); [email protected] (C.S.); [email protected] (C.O.D.); [email protected] (P.C.) 2 Cancer Stem Cell Project, National Cancer Center Research Institute, 5-1-1 Tsukiji, Chuo-ku, Tokyo 104-0045, Japan; E-Mails: [email protected] (Y.M.); [email protected] (M.Y.); [email protected] (V.K.) 3 Precursory Research for Embryonic Science and Technology (PREST), Japan Science and Technology Agency, 4-1-8 Honcho Kawaguchi, Saitama 332-0012, Japan † These authors contributed equally to this work. * Authors to whom correspondence should be addressed; E-Mails: [email protected] (Y.H.); [email protected] (K.M.); Tel.: +81-45-503-9248 (Y.H.); +81-3-3547-5173 (K.M.); Fax: +81-45-503-9216 (Y.H.); +81-3-3547-5123 (K.M.). Academic Editor: Martin Pichler Received: 22 November 2014 / Accepted: 26 December 2014 / Published: 6 January 2015 Abstract: MicroRNAs are small non-coding RNAs that inhibit the translation of target mRNAs. -

Charakterisierung Interindividueller Strahlenempfindlichkeit in Zellen Aus Tumorpatienten
Charakterisierung interindividueller Strahlenempfindlichkeit in Zellen aus Tumorpatienten Anne Maria Dietz Charakterisierung interindividueller Strahlenempfindlichkeit in Zellen aus Tumorpatienten Dissertation an der Fakultät für Biologie der Ludwig-Maximilians-Universität München zur Erlangung des Doktorgrades der Naturwissenschaften Angefertigt an der Klinik und Poliklinik für Strahlentherapie und Radioonkolgie in der Arbeitsgruppe Molekulare Strahlenbiologie unter der Leitung von PD. Dr. Anna Friedl Vorgelegt von Anne Maria Dietz München, den 03. Dezember 2014 Erstgutachter: PD Dr. Anna A. Friedl Zweitgutachter: Prof. Dr. Peter Becker Tag der mündlichen Prüfung: 05.05.2015 Meinem Vater Inhaltsverzeichnis Inhaltsverzeichnis 1 Zusammenfassung ........................................................................................................................... 5 2 Einleitung ......................................................................................................................................... 7 2.1 Individuelle Strahlenempfindlichkeit ...................................................................................... 7 2.2 Das Chromosomeninstabilitätssyndrom Ataxia Telangiectasia .............................................. 9 2.3 Biomarker-Entdeckung im Zeitalter der Omics-Technologien .............................................. 12 2.3.1 Charakterisierung eines validen Biomarkers ................................................................. 12 2.3.2 Biomarker für Strahlenempfindlichkeit ........................................................................ -

Insights Into Transcription Through Nascent RNA Sequencing
Insights into transcription through nascent RNA sequencing by Artur Botelho Veloso A dissertation submitted in partial fulfillment of the requirements for the degree of Doctor of Philosophy (Bioinformatics) in The University of Michigan 2014 Doctoral Committee: Professor Mats E.D. Ljungman, Chair Associate Professor Scott E. Barolo Professor Daniel M. Burns Jr. Assistant Professor Hui Jiang Professor Kerby A. Shedden Associate Professor Thomas E. Wilson c Artur B Veloso 2014 All Rights Reserved Para os meus pais, L´eae Rog´erio. ii ACKNOWLEDGEMENTS At´eonde eu me lembro, o meu interesse em ci^enciae pesquisa me acompanhou durante toda a minha vida. Quando crian¸caeu me sentia fascinado pelo mundo cient´ıficoe almejava o dia em que eu faria parte desse mundo. Durante a ´ultima d´ecadaeu tive a oportunidade de perseguir esse sonho, mas isso foi feita a duras penas. A minha decis~aode mudar de pa´ıse deixar fam´ıliae amigos para tr´asn~ao foi tomada facilmente. Eu n~aoa teria tomado, no entanto, se n~aotivesse recebido tanto apoio dessas mesmas pessoas. Esse processo n~aofoi f´acilpara mim, assim como n~aofoi f´acilpara eles. Por isso, eu primeiramente gostaria de agradecer `as pessoas que tem me apoiado durante toda minha vida. Minha m~aee meu pai, L´eae Rog´erio,foram fundamentais em meu desenvolvimento. Al´emdas qualidades b´asicas que bons pais ensinam a filhos, eles me ajudaram a desenvolver um forte sentido de independ^encia.Essa independ^enciafoi fundamental na minha decis~aode estudar em outro pa´ıs. Outro aspecto importante nessa decis~aofoi a minha ambi¸c~ao.Uma das pessoas que tiveram o maior impacto nessa caracter´ıstica foi meu irm~ao,Cristiano.