Instructions to RFLP Blind Test Application
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

An Annotated Checklist of the Shorefishes of the Canary Islands
AMERICAN MUSEUM Novitates PUBLISHED BY THE AMERICAN MUSEUM OF NATURAL HISTORY CENTRAL PARK WEST AT 79TH STREET, NEW YORK, N.Y. 10024 Number 2824, pp. 1-49, figs. 1-5 August 7, 1985 An Annotated Checklist of the Shorefishes of the Canary Islands JAMES K. DOOLEY,' JAMES VAN TASSELL,2 AND ALBERTO BRITO3 ABSTRACT The inshore canarian fish fauna includes 217 The fish fauna contains elements from the Med- species from 67 families. Fifteen new records (in- iterranean-Atlantic and West African areas, but cluding two undescribed species) and numerous does not exhibit any clear transition. Three en- rare species have been included. The number of demic species of fishes have been confirmed. The fishes documented from the Canary Islands and families with the greatest diversification include: nearby waters total approximately 400 species. Sparidae (21 species), Scorpaenidae (1 1), Gobiidae This figure includes some 200 pelagic, deepwater, (1 1), Blenniidae (10), Serranidae (9), Carangidae and elasmobranch species not treated in this study. (9), Muraenidae (7), and Labridae (7). RESUMEN La fauna ictiologica de las aguas costeras se las en el presente trabajo. La fauna contiene elemen- Islas Canarias comprende 217 especies de 67 fa- tos de las regiones Atlantico-Mediterranea y Oeste milias. Se incluyen quince citas nuevas (incluyen Africana, pero no muestra una clara transicion. dos especies no describen) y numerosas especies Tres especie endemica existe. Las familias con ma- raras. El nu'mero de peces de las aguas canarias se yor diversificacion son: Sparidae (21 especies), eleva aproximadamente a 400 especies. Este nui- Scorpaenidae (1 1), Gobiidae (1 1), Blenniidae (10), mero incluye casi 200 especies pelagicas, de aguas Serranidae (9), Carangidae (9), Muraenidae (7), y profundas y elasmobranquios que no se discuten Labridae (7). -

229 Index of Scientific and Vernacular Names
previous page 229 INDEX OF SCIENTIFIC AND VERNACULAR NAMES EXPLANATION OF THE SYSTEM Type faces used: Italics : Valid scientific names (genera and species) Italics : Synonyms * Italics : Misidentifications (preceded by an asterisk) ROMAN (saps) : Family names Roman : International (FAO) names of species 230 Page Page A African red snapper ................................................. 79 Abalistes stellatus ............................................... 42 African sawtail catshark ......................................... 144 Abámbolo ............................................................... 81 African sicklefìsh ...................................................... 62 Abámbolo de bajura ................................................ 81 African solenette .................................................... 111 Ablennes hians ..................................................... 44 African spadefish ..................................................... 63 Abuete cajeta ........................................................ 184 African spider shrimp ............................................. 175 Abuete de Angola ................................................. 184 African spoon-nose eel ............................................ 88 Abuete negro ........................................................ 184 African squid .......................................................... 199 Abuete real ........................................................... 183 African striped grunt ................................................ -

Zoogeography of Digenetic Trematodes from West African Marine Fishes1
192 PROCEEDINGS OF THE HELMINTHOLOGICAL SOCIETY Zoogeography of Digenetic Trematodes from West African Marine Fishes1 JACOB H. FISCHTHAL Department of Biological Sciences, State University of New York at Binghamton, Binghamton, New York 13901. ABSTRACT: Of the 107 species of trematodes found in West African (Mauritania to Gabon) marine fishes, 100 are allocated to 64 genera in 24 families while seven are immature didymozoids. Many of these genera are located in most of the world's seas with the exception of the polar seas; only five are en- demic to West Africa. The data for the 41 species known from West Africa and elsewhere, and those morphologically closest to the 55 endemic species, indicate that they are very widely distributed, particularly in the Western and North Atlantic, and Mediterranean. Historical and present- day events concerning physical and biological environmental factors and their effects on actual and po- tential hosts as well as on life cycle stages of the trematodes have resulted in the geographical distribution reported. The distribution of marine fishes has been emphasized to explain in part the trematode distribu- tion. Studies on the geographical distribution of (Gulf of Guinea from 5° S to 15° N) and digenetic trematodes of marine fishes in various warm temperate Mauritania have been pre- seas have been presented by Manter (1955, sented by Ekman (1953), Buchanan (1958), 1963, 1967), Szidat (1961), and Lebedev Longhurst (1962), and Ingham (1970). (1969), but West African waters were not included as sufficient data were not available Zoogeographical Distribution until more recently. The digenetic trematodes Of the 107 species of trematodes found in of West African marine fishes (mainly shore West African fishes, 100 are allocated to 64 and shelf inhabitants) have been reported by genera in 24 families while seven are immature Dollfus (1929, 1937a, b, 1946, 1951, 1960), didymozoids of unknown generic status (Ap- Dollfus and Capron (1958), Thomas (1959, pendix I). -

Otolith Atlas for the Western Mediterranean, North and Central Eastern Atlantic
SCIENTIA MARINA 72S1 July 2008, 7-198, Barcelona (Spain) ISSN: 0214-8358 Otolith atlas for the western Mediterranean, north and central eastern Atlantic VICTOR M. TUSET 1, ANTONI LOMBARTE 2 and CARLOS A. ASSIS 3 1 Instituto Canario de Ciencias Marinas, Departamento de Biología Pesquera, P.O. Box. 56, E-35200 Telde (Las Palmas), Canary Islands, Spain. E-mail: [email protected] 2 Institut de Ciències del Mar-CSIC, Departament de Recursos Marins Renovables, Passeig Marítim 37-49, Barcelona 08003, Catalonia, Spain. 3 Instituto de Oceanografia e Departamento de Biologia Animal, Faculdade de Ciências da Universidade de Lisboa, Campo Grande 1749-016, Lisboa, Portugal. SUMMARY: The sagittal otolith of 348 species, belonging to 99 families and 22 orders of marine Teleostean fishes from the north and central eastern Atlantic and western Mediterranean were described using morphological and morphometric characters. The morphological descriptions were based on the otolith shape, outline and sulcus acusticus features. The mor- phometric parameters determined were otolith length (OL, mm), height (OH, mm), perimeter (P; mm) and area (A; mm2) and were expressed in terms of shape indices as circularity (P2/A), rectangularity (A/(OL×OH)), aspect ratio (OH/OL; %) and OL/fish size. The present Atlas provides information that complements the characterization of some ichthyologic taxa. In addition, it constitutes an important instrument for species identification using sagittal otoliths collected in fossiliferous layers, in archaeological sites or in feeding remains of bony fish predators. Keywords: otolith, sagitta, morphology, morphometry, western Mediterranean, north eastern Atlantic, central eastern Atlantic. RESUMEN: Otolitos de peces del mediterráneo occidental y del atlántico central y nororiental. -

Identification of the Sole Resources of the Gambia
Identification of the Sole Resources of The Gambia Gambia-Senegal Sustainable Fisheries Program (Ba Nafaa) December 2011 This publication is available electronically on the Coastal Resources Center’s website at http://www.crc.uri.edu. For more information contact: Coastal Resources Center, University of Rhode Island, Narragansett Bay Campus, South Ferry Road, Narragansett, Rhode Island 02882, USA. Tel: 401) 874-6224; Fax: 401) 789-4670; Email: [email protected] The BaNafaa project is implemented by the Coastal Resources Center of the University of Rhode Island and the World Wide Fund for Nature-West Africa Marine Ecoregion (WWF-WAMER) in partnership with the Department of Fisheries and the Ministry of Fisheries, Water Resources and National Assembly Matters. Citation: Coastal Resources Center, 2011. Identification of the Sole Resources of The Gambia. Coastal Resources Center, University of Rhode Island, pp.11 Disclaimer: This report was made possible by the generous support of the American people through the United States Agency for International Development (USAID). The contents are the responsibility of the authors and do not necessarily reflect the views of USAID or the United States Government. Cooperative Agreement # 624-A-00-09- 00033-00. Cover Photo: Coastal Resources Center/URI Fisheries Center Photo Credit: Coastal Resources Center/URI Fisheries Center 2 The Sole Resources Proper identification of the species is critical for resource management. There are four major families of flatfish with representative species found in the Gambian nearshore waters: Soleidae, Cynoglossidae, Psettododae and Paralichthyidae. The species below have been confirmed through literature review, and through discussions with local fishermen, processors and the Gambian Department of Fisheries. -
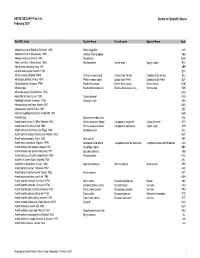
ASFIS ISSCAAP Fish List February 2007 Sorted on Scientific Name
ASFIS ISSCAAP Fish List Sorted on Scientific Name February 2007 Scientific name English Name French name Spanish Name Code Abalistes stellaris (Bloch & Schneider 1801) Starry triggerfish AJS Abbottina rivularis (Basilewsky 1855) Chinese false gudgeon ABB Ablabys binotatus (Peters 1855) Redskinfish ABW Ablennes hians (Valenciennes 1846) Flat needlefish Orphie plate Agujón sable BAF Aborichthys elongatus Hora 1921 ABE Abralia andamanika Goodrich 1898 BLK Abralia veranyi (Rüppell 1844) Verany's enope squid Encornet de Verany Enoploluria de Verany BLJ Abraliopsis pfefferi (Verany 1837) Pfeffer's enope squid Encornet de Pfeffer Enoploluria de Pfeffer BJF Abramis brama (Linnaeus 1758) Freshwater bream Brème d'eau douce Brema común FBM Abramis spp Freshwater breams nei Brèmes d'eau douce nca Bremas nep FBR Abramites eques (Steindachner 1878) ABQ Abudefduf luridus (Cuvier 1830) Canary damsel AUU Abudefduf saxatilis (Linnaeus 1758) Sergeant-major ABU Abyssobrotula galatheae Nielsen 1977 OAG Abyssocottus elochini Taliev 1955 AEZ Abythites lepidogenys (Smith & Radcliffe 1913) AHD Acanella spp Branched bamboo coral KQL Acanthacaris caeca (A. Milne Edwards 1881) Atlantic deep-sea lobster Langoustine arganelle Cigala de fondo NTK Acanthacaris tenuimana Bate 1888 Prickly deep-sea lobster Langoustine spinuleuse Cigala raspa NHI Acanthalburnus microlepis (De Filippi 1861) Blackbrow bleak AHL Acanthaphritis barbata (Okamura & Kishida 1963) NHT Acantharchus pomotis (Baird 1855) Mud sunfish AKP Acanthaxius caespitosa (Squires 1979) Deepwater mud lobster Langouste -

Age, Growth, and Reproductive Biology of Blackcheek Tonguefish, Symphurus Plagiusa (Cynoglossidae: Pleuronectiformes), in Chesapeake Bay, Virginia
W&M ScholarWorks Dissertations, Theses, and Masters Projects Theses, Dissertations, & Master Projects 1996 Age, Growth, and Reproductive Biology of Blackcheek Tonguefish, Symphurus plagiusa (Cynoglossidae: Pleuronectiformes), in Chesapeake Bay, Virginia Mark Richard Terwilliger College of William and Mary - Virginia Institute of Marine Science Follow this and additional works at: https://scholarworks.wm.edu/etd Part of the Fresh Water Studies Commons, Oceanography Commons, and the Zoology Commons Recommended Citation Terwilliger, Mark Richard, "Age, Growth, and Reproductive Biology of Blackcheek Tonguefish, Symphurus plagiusa (Cynoglossidae: Pleuronectiformes), in Chesapeake Bay, Virginia" (1996). Dissertations, Theses, and Masters Projects. Paper 1539617701. https://dx.doi.org/doi:10.25773/v5-msx0-vs25 This Thesis is brought to you for free and open access by the Theses, Dissertations, & Master Projects at W&M ScholarWorks. It has been accepted for inclusion in Dissertations, Theses, and Masters Projects by an authorized administrator of W&M ScholarWorks. For more information, please contact [email protected]. Age, growth, and reproductive biology of blackcheek tonguefish, Symphurus plagiusa (Cynoglossidae: Pleuronectiformes), in Chesapeake Bay, Virginia A Thesis Presented to The Faculty of the School of Marine Science The College of William and Mary In Partial Fulfillment of the Requirements for the Degree of Master of Arts By Mark R. Terwilliger 1996 This thesis is submitted in partical fulfillment of the requirements for the degree of Master of Arts Mark R. Terwi Approved, December 1996 Thomas A. Munroe, Ph.D. National Marine Fisheries Service National Systematics Laboratory National Museum of Natural History Washington, D.C. 20560 / John A. Musick, Ph.D. H - , L , j M ' j \ Y. -

2003. Fish Biodiversity: Local Studies As Basis for Global Inferences
Fish Biodiversity: Local Studies as Basis for Global Inferences. M.L.D. Palomares, B. Samb, T. Diouf, J.M. Vakily and D. Pauly (Eds.) ACP – EU Fisheries Research Report NO. 14 ACP-EU Fisheries Research Initiative Fish Biodiversity: Local Studies as Basis for Global Inferences Edited by Maria Lourdes D. Palomares Fisheries Centre, University of British Columbia, Vancouver, Canada Birane Samb Centre de Recherches Océanographiques de Dakar-Thiaroye, Sénégal Taïb Diouf Centre de Recherches Océanographiques de Dakar-Thiaroye, Sénégal Jan Michael Vakily Joint Research Center, Ispra, Italy and Daniel Pauly Fisheries Centre, University of British Columbia, Vancouver, Canada Brussels December 2003 ACP-EU Fisheries Research Report (14) – Page 2 Fish Biodiversity: Local Studies as Basis for Global Inferences. M.L.D. Palomares, B. Samb, T. Diouf, J.M. Vakily and D. Pauly (eds.) The designations employed and the presentation of material in this publication do not imply the expression of any opinion whatsoever on the part of the European Commission concerning the legal status of any country, territory, city or area or of its authorities, or concerning the delimitation of frontiers or boundaries. Copyright belongs to the European Commission. Nevertheless, permission is hereby granted for reproduction in whole or part for educational, scientific or development related purposes, except those involving commercial sale on any medium whatsoever, provided that (1) full citation of the source is given and (2) notification is given in writing to the European Commission, Directorate General for Research, INCO-Programme, 8 Square de Meeûs, B-1049 Brussels, Belgium. Copies are available free of charge upon request from the Information Desks of the Directorate General for Development, 200 rue de la Loi, B-1049 Brussels, Belgium, and of the INCO-Programme of the Directorate General for Research, 8 Square de Meeûs, B-1049 Brussels, Belgium, E-mail: [email protected]. -

A Revision of Irvine's Marine Fishes of Tropical West Africa
A Revision of Irvine’s Marine Fishes of Tropical West Africa by ALASDAIR J. EDWARDS Tropical Coastal Management Studies, Department of Marine Sciences and Coastal Management, University of Newcastle, Newcastle upon Tyne NE1 7RU, United Kingdom ANTHONY C. GILL Department of Zoology, The Natural History Museum, Cromwell Road, London SW7 5BD, United Kingdom and PARCY O. ABOHWEYERE, Nigerian Institute for Oceanography and Marine Research, Wilmot Point Road, Bar-Beach, P.M.B. 12729, Victoria Island, Lagos, Nigeria TABLE OF CONTENTS INTRODUCTION .......................................................................................................................1 SHARKS AND RAYS (EUSELACHII) .......................................................................................4 Sixgill and Sevengill sharks (Hexanchidae) ......................................................................................4 Sand tiger sharks (Odontaspididae) .................................................................................................5 Mackerel sharks (Lamnidae) .........................................................................................................5 Thresher Sharks (Alopiidae) ..........................................................................................................7 Nurse sharks (Ginglymostomatidae) ...............................................................................................7 Requiem sharks (Carcharhinidae) ...................................................................................................8 -
Fishery Co-Management Plan for the Gambia Sole Complex
Fishery Co-Management Plan For The Gambia Sole Complex (focus on artisanal fisheries sub-sector) January 2012 Ministry of Fisheries, Water Resources and National Assembly Matters TABLE OF CONTENTS Page Co-Management Agreement ....................................................................................................... iv Chapter 1: Introduction ...............................................................................................................1 1.1 Principles for Management of the Fishery ............................................................................ 2 1.2 The Process of Developing the Sole Management Plan ....................................................... 3 1.3 Contents of the Sole Plan ...................................................................................................... 4 Chapter 2: The Resources ............................................................................................................5 2.1 The Species ........................................................................................................................... 5 2.2 Distribution and Migration ................................................................................................... 5 2.3 Growth and Spawning........................................................................................................... 7 Chapter 3: The Fishery ................................................................................................................9 3.1 History of Exploitation ........................................................................................................ -

A Phylogenetic Assessment of Flatfish (Order Pleuronectiformes)
A phylogenetic assessment of flatfish (Order Pleuronectiformes) intrarelationships based on molecular evidence Lisa Byrne Thesis submitted to the Office of Graduate Studies in partial fulfillment of the requirements for the degree of Master of Science in Biology Department of Biology Faculty of Science University of Ottawa © Lisa Byrne, Ottawa, Canada, 2018 This work is dedicated to my son Hunter. I have embodied many qualities while undertaking this project. Perseverance. Resilience. Following a path that you truly believe in. I hope that I am able to cultivate in you some of these values as we navigate your upbringing. This thesis is also dedicated in loving memory of Uncle Georgie. Always ready with canoe paddles, a campfire or a homemade fishing rod, and the general advice to go outside and play. Summers at Lac Labelle piqued my biological curiosity at an early age. My life is much richer for having had the luxury of these experiences. ii Acknowledgements I would like to thank my supervisor, François Chapleau, for giving me the opportunity to pursue graduate studies in systematic biology. His guidance, patience, and zest for zoology is greatly appreciated. I would like to extend my sincerest thanks to Stéphane Aris-Brosou for providing a wealth of expertise that made this project possible. This work could not have been completed without his encouragement, editing and enthusiasm. I would also like to thank the other members of my committee, Keith Seifert and Julian Starr for their time, skill, and helpful suggestions. I would also extend my gratitude to Claude Paquette in the financial aid department for helping me time and again find creative funding opportunities during the course of my studies. -

A Systematic Monograph of the Tongue Soles of the Genus Cynoglossus Hamilton-Buchanan (Pisces: Cynoglossidae)
A Systematic Monograph of the Tongue Soles of the Genus Cynoglossus Hamilton-Buchanan (Pisces: Cynoglossidae) A. G. K..MENON SMITHSONIAN CONTRIBUTIONS TO ZOOLOGY • NUMBER 238 SERIES PUBLICATIONS OF THE SMITHSONIAN INSTITUTION Emphasis upon publication as a means of "diffusing knowledge" was expressed by the first Secretary of the Smithsonian. In his formal plan for the Institution, Joseph Henry outlined a program that included the following statement: "It is proposed to publish a series of reports, giving an account of the new discoveries in science, and of the changes made from year to year in all branches of knowledge." This theme of basic research has been adhered to through the years by thousands of titles issued in series publications under the Smithsonian imprint, commencing with Smithsonian Contributions to Knowledge in 1848 and continuing with the following active series: Smithsonian Contributions to Anthropology Smithsonian Contributions to Astrophysics Smithsonian Contributions to Botany Smithsonian Contributions to the Earth Sciences Smithsonian Contributions to Paleobiology Smithsonian Contributions to Zoology Smithsonian Studies in Air and Space Smithsonian Studies in History and Technology In these series, the Institution publishes small papers and full-scale monographs that report the research and collections of its various museums and bureaux or of professional colleagues in the world cf science and scholarship. The publications are distributed by mailing lists to libraries, universities, and similar institutions throughout the world. Papers or monographs submitted for series publication are received by the Smithsonian Institution Press, subject to its own review for format and style, only through departments of the various Smithsonian museums or bureaux, where the manuscripts are given substantive review.