These De Doctorat De L'universite Paris-Saclay
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

In Vitro Regeneration of the Croatian Endemic Species Iris Adriatica Trinajstić Ex Mitić
ACTA BIOLOGICA CRACOVIENSIA Series Botanica 51/2: 7–12, 2009 IN VITRO REGENERATION OF THE CROATIAN ENDEMIC SPECIES IRIS ADRIATICA TRINAJSTIĆ EX MITIĆ SNJEŽANA KEREŠA1*, ANITA MIHOVILOVIĆ1, MIRNA ĆURKOVIĆ-PERICA2, BOŽENA MITIĆ2, MARIJANA BARIĆ1, INES VRŠEK1 AND STEFANO MARCHETTI3 1Department of Plant Breeding, Genetics and Biometrics, Faculty of Aqriculture, University of Zagreb, Svetošimunska 25, 10000 Zagreb, Croatia 2Department of Botany and Botanical Garden, Faculty of Science, University of Zagreb, Marulićev trg 20/II, 10000 Zagreb, Croatia 3Department of Agriculture and Environmental Sciences, University of Udine, Via delle Scienze 208, 33100 Udine, Italy Received December 15, 2008; revision accepted May 29, 2009 Plant regeneration via somatic embryogenesis and organogenesis was achieved in leaf base and ovary culture of the Croatian endemic Iris adriatica Trinajstić ex Mitić. Callus induction from leaf base explants occurred in the dark on three media with MS mineral solution containing 4.52 μM dichlorophenoxyacetic acid (2,4-D), 4.83 μM naphthaleneacetic acid (NAA), 0.46 μM kinetin (Kin), 5% sucrose and 200 mg L-1 casein hydrolysate. The media differed only in vitamin and/or proline content. Calli from ovary culture were achieved on MS medium contain- ing 45.25 μM 2,4-D. The mean percentage of callus induction from leaf base explants was 18.9%, with no sig- nificant differences between media, and 27.3% from ovary sections. All embryogenic calli were formed on MS media containing 0.45 μM 2,4-D, 4.44 μM benzyladenine (BA) and 0.49 μM indole-3-butyric acid (IBA) under low light intensity (25 μE m-2s-1). -

Pdf (306.01 K)
REVIEW ARTICLE RECORDS OF PHARMACEUTICAL AND BIOMEDICAL SCIENCES Review article on chemical constituents and biological activity of Thymelaea hirsuta. Ahmed M Badawya, Hashem A Hassaneanb, Amany K. Ibrahimb, Eman S. Habibb, Safwat A. Ahmedb* aDepartment of Pharmacognosy, Faculty of Pharmacy, Sinai University, El-Arish, Egypt, b Department of Pharmacognosy, Faculty of Pharmacy, Suez Canal University, Ismailia, Egypt 41522. Abstract Received on: 07.04. 2019 Thymelaea hirsuta a perennial, evergreen and dioecious shrub, which is native Revised on: 30. 04. 2019 to North Africa. T. hirsuta is a widespread invasive weed and is commonly known as “Methnane”. Along the history, T. hirsuta, family Thymelaeaceae, Accepted on: 10. 04. 2019 has been recognized as an important medicinal plant. Much research has been carried out on the medical applications of Methnane. The choice of the plant was based on the good previous biological study of T. hirsuta plant extract to Correspondence Author: use as anticancer, hepatoprotective and anti-diapetic. Several species of Tel:+ 01092638387 Thymelaeaceae have been the subject of numerous phytochemical studies. Initially, interest may have been due to the marked toxicity of these plants, but E-mail address: the widespread use of some species medicinally has certainly played a part in [email protected] sustaining this interest. Keywords: Thymelaea hirsuta , Chemical constituents, Biological activity 1.Introduction: Near East: Lebanon and Palestine. The choice of the plant was based on the good previous Thymelaea hirsuta a perennial, evergreen and biological study of T. hirsuta plant extract to use dioecious shrub, which is native to North Africa. T. as anticancer, hepatoprotective and anti-diabetic. -

Scrub Mint Dicerandra Frutescens Shinners
Scrub Mint Dicerandra frutescens Shinners he scrub mint is a small, fragrant shrub that inhabits Federal Status: Endangered (Nov. 1, 1985) the scrub of central peninsular Florida. It bears a Critical Habitat: None Designated Tstrong resemblance to another Dicerandra species, Florida Status: Endangered Garretts mint, but can be differentiated by its scent, the Recovery Plan Status: Revision (May 18, 1999) color of its flowers, and the size of its leaves. Loss of habitat due to residential and agricultural development (particularly Geographic Coverage: Rangewide for citrus groves), as well as fire suppression in tracts of remaining habitat, are the principle threats to this plant. This account represents a revision of the existing Figure 1. County distribution of scrub mint. recovery plan for the scrub mint (FWS 1987). Description The scrub mint is a dense or straggly, low-growing shrub (Kral, 1983). It reaches 50 cm in height and grows from a deep, stout, spreading-branching taproot. Its branches are mostly spreading, and sometimes are prostrate. Its shoots have two forms, one which is strictly leafy and overwintering, and another which is flowering and dies back after fruiting. The leaves vary in shape. They can be narrowly oblong- elliptic, linear-elliptic, or linear-oblanceolate (Kral 1983). The upper surface of the leaves is dark green, with the midrib slightly impressed. The lower surface is slightly paler, with the midrib slightly raised. They are 1.5 to 2.5 cm long, 2 to 3 mm wide, subsessile, flattish but somewhat fleshy, narrowly or broadly rounded at the apical end, have entire margins, and are not revolute. -

Legumes of the North-Central States: C
LEGUMES OF THE NORTH-CENTRAL STATES: C-ALEGEAE by Stanley Larson Welsh A Dissertation Submitted, to the Graduate Faculty in Partial Fulfillment of The Requirements for the Degree of DOCTOR OF PHILOSOPHY Major Subject: Systematic Botany Approved: Signature was redacted for privacy. Signature was redacted for privacy. artment Signature was redacted for privacy. Dean of Graduat College Iowa State University Of Science and Technology Ames, Iowa I960 ii TABLE OF CONTENTS Page ACKNOWLEDGMENTS iii INTRODUCTION 1 HISTORICAL ACCOUNT 3 MATERIALS AND METHODS 8 TAXONOMIC AND NOMENCLATURE TREATMENT 13 REFERENCES 158 APPENDIX A 176 APPENDIX B 202 iii ACKNOWLEDGMENTS The writer wishes to express his deep gratitude to Professor Duane Isely for assistance in the selection of the problem and for the con structive criticisms and words of encouragement offered throughout the course of this investigation. Support through the Iowa Agricultural Experiment Station and through the Industrial Science Research Institute made possible the field work required in this problem. Thanks are due to the curators of the many herbaria consulted during this investigation. Special thanks are due the curators of the Missouri Botanical Garden, U. S. National Museum, University of Minnesota, North Dakota Agricultural College, University of South Dakota, University of Nebraska, and University of Michigan. The cooperation of the librarians at Iowa State University is deeply appreciated. Special thanks are due Dr. G. B. Van Schaack of the Missouri Botanical Garden library. His enthusiastic assistance in finding rare botanical volumes has proved invaluable in the preparation of this paper. To the writer's wife, Stella, deepest appreciation is expressed. Her untiring devotion, work, and cooperation have made this work possible. -

Natural Resource Condition Assessment for Stones River National Battlefield
National Park Service U.S. Department of the Interior Natural Resource Stewardship and Science Natural Resource Condition Assessment for Stones River National Battlefield Natural Resource Report NPS/STRI/NRR—2016/1141 ON THIS PAGE Native warm season grass, located south of Stones River National Battlefield visitor center Photograph by: Jeremy Aber, MTSU Geospatial Research Center ON THE COVER Karst topography in the cedar forest at the “Slaughter Pen,” Stones River National Battlefield Photograph by: Jeremy Aber, MTSU Geospatial Research Center Natural Resource Condition Assessment for Stones River National Battlefield Natural Resource Report NPS/STRI/NRR—2016/1141 Henrique Momm Zada Law Siti Nur Hidayati Jeffrey Walck Kim Sadler Mark Abolins Lydia Simpson Jeremy Aber Geospatial Research Center Department of Geosciences Middle Tennessee State University Murfreesboro, Tennessee 37132 February 2016 U.S. Department of the Interior National Park Service Natural Resource Stewardship and Science Fort Collins, Colorado The National Park Service, Natural Resource Stewardship and Science office in Fort Collins, Colorado, publishes a range of reports that address natural resource topics. These reports are of interest and applicability to a broad audience in the National Park Service and others in natural resource management, including scientists, conservation and environmental constituencies, and the public. The Natural Resource Report Series is used to disseminate comprehensive information and analysis about natural resources and related topics concerning lands managed by the National Park Service. The series supports the advancement of science, informed decision-making, and the achievement of the National Park Service mission. The series also provides a forum for presenting more lengthy results that may not be accepted by publications with page limitations. -

Extrapolating Demography with Climate, Proximity and Phylogeny: Approach with Caution
! ∀#∀#∃ %& ∋(∀∀!∃ ∀)∗+∋ ,+−, ./ ∃ ∋∃ 0∋∀ /∋0 0 ∃0 . ∃0 1##23%−34 ∃−5 6 Extrapolating demography with climate, proximity and phylogeny: approach with caution Shaun R. Coutts1,2,3, Roberto Salguero-Gómez1,2,3,4, Anna M. Csergő3, Yvonne M. Buckley1,3 October 31, 2016 1. School of Biological Sciences. Centre for Biodiversity and Conservation Science. The University of Queensland, St Lucia, QLD 4072, Australia. 2. Department of Animal and Plant Sciences, University of Sheffield, Western Bank, Sheffield, UK. 3. School of Natural Sciences, Zoology, Trinity College Dublin, Dublin 2, Ireland. 4. Evolutionary Demography Laboratory. Max Planck Institute for Demographic Research. Rostock, DE-18057, Germany. Keywords: COMPADRE Plant Matrix Database, comparative demography, damping ratio, elasticity, matrix population model, phylogenetic analysis, population growth rate (λ), spatially lagged models Author statement: SRC developed the initial concept, performed the statistical analysis and wrote the first draft of the manuscript. RSG helped develop the initial concept, provided code for deriving de- mographic metrics and phylogenetic analysis, and provided the matrix selection criteria. YMB helped develop the initial concept and advised on analysis. All authors made substantial contributions to editing the manuscript and further refining ideas and interpretations. 1 Distance and ancestry predict demography 2 ABSTRACT Plant population responses are key to understanding the effects of threats such as climate change and invasions. However, we lack demographic data for most species, and the data we have are often geographically aggregated. We determined to what extent existing data can be extrapolated to predict pop- ulation performance across larger sets of species and spatial areas. We used 550 matrix models, across 210 species, sourced from the COMPADRE Plant Matrix Database, to model how climate, geographic proximity and phylogeny predicted population performance. -

How Illinois Kicked the Exotic Habit
HOW ILLINOIS KICKED THE EXOTIC HABIT Francis M. Harty Illinois Department of Conservation 2005 Round Bam Road Champagne, IL 61821 Introduction For the purpose of this paper, an exotic species is defined as "a plant or animal not native to North America." The history of folly surrounding the premeditated and accidental introduction of exotic animals has been well-documented (DeVos et al. 1956, Elton 1958, Hall 1963, Laycock 1966, Ehrenfeld 1970, Bratton 1974/1975, Howe and Bratton 1976, Moyle 1976, Courtenay 1978, Coblentz 1978, Iverson 1978, Weller 1981, Bratton 1982, Vale 1982, and Savidge 1987). In 1963, Dr. E. Raymond Hall wrote, "Introducing exotic species of vertebrates is unscientific, economically wasteful, politically shortsighted, and biologically wrong." Naturalizing exotic species are living time bombs, but no one knows for sure how much time we have. For example, the ring necked pheasant (Phasianus colchicus), touted as the Midwestern example of a good exotic introduction, has recently developed a nefarious relationship with the greater prairie chicken (Tympanuchus cupido) in Illinois. Parasitism of prairie chicken nests by hen pheasants and harassment of displaying male chickens by cock pheasants are contributing to the decline of prairie chickens in Illinois (Vance and Westemeier 1979). The interspecific competition between the exotic pheasant (which is expanding its range in Illinois) and the native prairie chicken (which is an endangered species in Illinois) may be the final factor causing the extirpation of the prairie chicken from Illinois; it has already been extirpated from neighboring Indiana. In 1953, Klimstra and Hankla wrote, "In connection with the development of a pheasant adapted to southern conditions, the compatibility of pheasants and quail (Colinus virginianus) needs to be evaluated. -
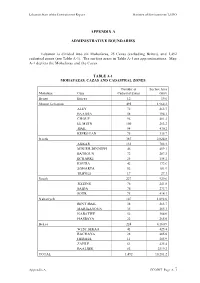
Appendix a Administrative Boundaries
Lebanon State of the Environment Report Ministry of Environment/LEDO APPENDIX A ADMINISTRATIVE BOUNDARIES Lebanon is divided into six Mohafazas, 25 Cazas (excluding Beirut), and 1,492 cadastral zones (see Table A-1). The surface areas in Table A-1 are approximations. Map A-1 depicts the Mohafazas and the Cazas. TABLE A-1 MOHAFAZAS, CAZAS AND CADASTRAL ZONES Number of Surface Area Mohafaza Caza Cadastral Zones (km2) Beirut Beirut 12 19.6 Mount Lebanon 495 1,968.3 ALEY 72 263.7 BAABDA 58 194.3 CHOUF 96 481.2 EL METN 100 263.2 JBAIL 94 430.2 KESROUAN 75 335.7 North 387 2,024.8 AKKAR 133 788.4 MINIEH-DINNIEH 46 409.1 BATROUN 72 287.3 BCHARRE 25 158.2 KOURA 42 172.6 ZGHARTA 52 181.9 TRIPOLI 17 27.3 South 227 929.6 JEZZINE 76 241.8 SAIDA 76 273.7 SOUR 75 414.1 Nabatiyeh 147 1,098.0 BENT JBAIL 38 263.7 MARJAAYOUN 35 265.3 NABATIYE 52 304.0 HASBAYA 22 265.0 Bekaa 224 4,160.9 WEST BEKAA 41 425.4 RACHAYA 28 485.0 HERMEL 11 505.9 ZAHLE 61 425.4 BAALBEK 83 2319.2 TOTAL 1,492 10,201.2 Appendix A. ECODIT Page A. 1 Lebanon State of the Environment Report Ministry of Environment/LEDO MAP A-1 ADMINISTRATIVE BOUNDARIES (MOHAFAZAS AND CAZAS) AKKAR Tripoli North #Y Lebanon HERMEL KOURA MINIEH-DINNIEH ZGHARTA BCHARRE BATROUN BAALBEK BATROUN Mount Bekaa Lebanon KESROUAN Beirut METN #Y BAABDA ZAHLE ALEY CHOUF WEST BEKAA Saida #Y JEZZINE RACHAYA SAIDA South NABATIYEH Lebanon HASBAYA Tyre Nabatiyeh #Y MARJAYOUN TYRE BINT JBEIL Appendix A. -

Towards an Updated Checklist of the Libyan Flora
Towards an updated checklist of the Libyan flora Article Published Version Creative Commons: Attribution 3.0 (CC-BY) Open access Gawhari, A. M. H., Jury, S. L. and Culham, A. (2018) Towards an updated checklist of the Libyan flora. Phytotaxa, 338 (1). pp. 1-16. ISSN 1179-3155 doi: https://doi.org/10.11646/phytotaxa.338.1.1 Available at http://centaur.reading.ac.uk/76559/ It is advisable to refer to the publisher’s version if you intend to cite from the work. See Guidance on citing . Published version at: http://dx.doi.org/10.11646/phytotaxa.338.1.1 Identification Number/DOI: https://doi.org/10.11646/phytotaxa.338.1.1 <https://doi.org/10.11646/phytotaxa.338.1.1> Publisher: Magnolia Press All outputs in CentAUR are protected by Intellectual Property Rights law, including copyright law. Copyright and IPR is retained by the creators or other copyright holders. Terms and conditions for use of this material are defined in the End User Agreement . www.reading.ac.uk/centaur CentAUR Central Archive at the University of Reading Reading’s research outputs online Phytotaxa 338 (1): 001–016 ISSN 1179-3155 (print edition) http://www.mapress.com/j/pt/ PHYTOTAXA Copyright © 2018 Magnolia Press Article ISSN 1179-3163 (online edition) https://doi.org/10.11646/phytotaxa.338.1.1 Towards an updated checklist of the Libyan flora AHMED M. H. GAWHARI1, 2, STEPHEN L. JURY 2 & ALASTAIR CULHAM 2 1 Botany Department, Cyrenaica Herbarium, Faculty of Sciences, University of Benghazi, Benghazi, Libya E-mail: [email protected] 2 University of Reading Herbarium, The Harborne Building, School of Biological Sciences, University of Reading, Whiteknights, Read- ing, RG6 6AS, U.K. -

PDF Document
Cyclamen Notes by Wilhelm (Bill) Bischoff Flowers of Atlantis? Page 2 Cyclamen Blooming Times Page 4 Cyclamen Species, Subspecies, Page 5 Forma, & Varieties in Alphabetical Order Cyclamen Descriptions Page 6 (photos referenced are not included) Wilhelm (Bill) Bischoff is available for lectures & garden tours for Cyclamen & Hardy Orchids 604-589-6134 wbischoff @ shaw.ca The Flowers of Atlantis? By Wilhelm (Bill) Bischoff / member BC Council of Garden Clubs If you can accept that the island called Santorini in the central Mediterranean, also known as Thira / Tera, is the original Island of Atlantis; if you also can agree that this Island had a terrific volcanic explosion more than 3,000 years ago, than I can share with you an equally fantastic botanical story with you. That today’s Thira is the remnant of an exploded volcano is quite evident when one looks at a map of this region of the Mediterranean. Located as part of the Aegean Islands, just north of Crete, it shows the unmistakable shape of a water filled volcanic caldera with a center-cone island. Scientists have identified volcanic ash taken from the bottom of the Mediterranean Sea, close to the Lebanese coast, as originating from Thira. The time frame of some 3300 years ago also coincides with the beginning of a rather tumultuous time in this part of the ancient world, the end of the “Bronze Age”. The possible cause of that could well have been a natural disaster, in the very heart of the ancient world as we know it. Now that I have your attention and possibly have whetted your curiosity, let me introduce you to one of the small wonders of this very ancient world, the beautiful Cyclamen, all 22 species of them. -

Banks in Lebanon
932-933.qxd 14/01/2011 09:13 Õ Page 2 AL BAYAN BUSINESS GUIDE USEFUL NUMBERS Airport International Calls (100) Ports - Information (1) 628000-629065/6 Beirut (1) 580211/2/3/4/5/6 - 581400 - ADMINISTRATION (1) 629125/130 Internal Security Forces (112) Byblos (9) 540054 - Customs (1) 629160 Chika (6) 820101 National Defense (1701) (1702) Jounieh (9) 640038 Civil Defence (125) Saida (7) 752221 Tripoli (6) 600789 Complaints & Review (119) Ogero (1515) Tyr (7) 741596 Consumer Services Protection (1739) Police (160) Water Beirut (1) 386761/2 Red Cross (140) Dbaye (4) 542988- 543471 Electricity (145) (1707) Barouk (5) 554283 Telephone Repairs (113) Jounieh (9) 915055/6 Fire Department (175) Metn (1) 899416 Saida (7) 721271 General Security (1717) VAT (1710) Tripoli (6) 601276 Tyr (7) 740194 Information (120) Weather (1718) Zahle (8) 800235/722 ASSOCIATIONS, SYNDICATES & OTHER ORGANIZATIONS - MARBLE AND CEMENT (1)331220 KESRWAN (9)926135 BEIRUT - PAPER & PACKAGING (1)443106 NORTH METN (4)926072-920414 - PHARMACIES (1)425651-426041 - ACCOUNTANTS (1)616013/131- (3)366161 SOUTH METN (5)436766 - PLASTIC PRODUCERS (1)434126 - ACTORS (1)383407 - LAWYERS - PORT EMPLOYEES (1) 581284 - ADVERTISING (1)894545 - PRESS (1)865519-800351 ALEY (5)554278 - AUDITOR (1)322075 BAABDA (5)920616-924183 - ARTIST (1)383401 - R.D.C.L. (BUSINESSMEN) (1)320450 DAIR AL KAMAR (5)510244 - BANKS (1)970500 - READY WEAR (3)879707-(3)236999 - CARS DRIVERS (1)300448 - RESTAURANTS & CAFE (1)363040 JBEIL (9)541640 - CHEMICAL (1)499851/46 - TELEVISIONS (5)429740 JDEIDET EL METN (1)892548 - CONTRACTORS (5)454769 - TEXTILLES (5)450077-456151 JOUNIEH (9)915051-930750 - TOURISM JOURNALISTS (1)349251 - DENTISTS (1)611222/555 - SOCKS (9)906135 - TRADERS (1)347997-345735 - DOCTORS (1)610710 - TANNERS (9)911600 - ENGINEERS (1)850111 - TRADERS & IND. -

Extracts Against the Maggots of the Housefly, Musca Domestica (Diptera: Muscidae) and of the Bluebottle Fly, Calliphoravomitoria (Diptera: Calliphoridae)
Volume-6, Issue-3, July-Sept-2016 Coden: IJPAJX-CAS-USA, Copyrights@2016 ISSN-2231-4490 Received: 28th March 2016 Revised: 16th June 2016 Accepted: 28th July 2016 DOI: 10.21276/Ijpaes http://dx.doi.org/10.21276/ijpaes Research article EFFECT OF CYCLAMEN LIBANOTICUM (MYRSINACEAE) EXTRACTS AGAINST THE MAGGOTS OF THE HOUSEFLY, MUSCA DOMESTICA (DIPTERA: MUSCIDAE) AND OF THE BLUEBOTTLE FLY, CALLIPHORAVOMITORIA (DIPTERA: CALLIPHORIDAE) Fadel Dania*, Hamze Mhammada, Chahine Halaa, Rizk Helenea, Aoun George a aLebanese University, Faculty of Agriculture, Department of Plant Production. Dekwaneh–Lebanon ABSTRACT: The objective of this study was to evaluate the larvicidal effect of the tuber extracts isolated from Cyclamen libanoticum against Musca domestica and Calliphora Vomitoria.For this reason, twenty young(second-third) instars of the Musca domestica and Calliphora Vomitoria were exposed separately to various concentrations (0.05 g/ml to 0.2 g/ml) of the extracts. Mortality was recorded after 10, 30, 60, 120, 180 and 240 min) of exposure. The results showed that the young larvae of Musca domestica were more susceptible to the extracts in comparison with Calliphora Vomitoria. A comparison of LC50 and LC90 values has shown that Cyclamen libanoticum wasvery active against both larval flies. Conclusions: This study is the first to report on the larvicidal activity of the extracts of Cyclamen libanoticum against Musca domestica and Calliphora Vomitoria. More studies are needed to isolate and identify the active components involved, their mode of action, and effects on other flies and pest species. Key words: Cyclamen libanoticum, Musca domestica, Calliphora Vomitoria, larvicidal activity, maggots *Corresponding author: Fadel Dani, Lebanese University, Faculty of Agriculture, Department of Plant Production.