Dissertation
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

(12) Patent Application Publication (10) Pub. No.: US 2012/0264.634 A1 Amersdorfer Et Al
US 20120264.634A1 (19) United States (12) Patent Application Publication (10) Pub. No.: US 2012/0264.634 A1 Amersdorfer et al. (43) Pub. Date: Oct. 18, 2012 (54) MARKER SEQUENCES FOR PANCREATIC Publication Classification CANCER DISEASES, PANCREATIC (51) Int. Cl. CARCINOMIA AND USE THEREOF C40B 30/04 (2006.01) GOIN 2L/64 (2006.01) (75) Inventors: Peter Amersdorfer, Graz (AT); GOIN 27/72 (2006.01) Annabel Höpfner, Dortmund (DE); C07K I4/435 (2006.01) Angelika Lueking, Bochum (DE) C40B 40/06 (2006.01) C40B 40/10 (2006.01) CI2N 5/09 (2010.01) (73) Assignee: PROTAGEN Aktiengesellschaft, C7H 2L/04 (2006.01) Dortmund (DE) GOIN 33/574 (2006.01) GOIN 27/62 (2006.01) (21) Appl. No.: 13/498,964 (52) U.S. Cl. ........... 506/9:436/501; 435/6.14; 435/7.92; 506/16:506/18: 435/2:536/23.1; 530/350 (22) PCT Filed: Sep. 29, 2010 (57) ABSTRACT The present invention relates to novel marker sequences for (86). PCT No.: PCT/EP2010/064510 pancreatic cancer diseases, pancreatic carcinoma and the diagnostic use thereof together with a method for Screening of S371 (c)(1), potential active Substances for pancreatic cancer diseases, (2), (4) Date: Jun. 22, 2012 pancreatic carcinoma by means of these marker sequences. Furthermore, the invention relates to a diagnostic device con (30) Foreign Application Priority Data taining Such marker sequences for pancreatic cancer diseases, pancreatic carcinoma, in particular a protein biochip and the Sep. 29, 2009 (EP) .................................. O9171690.2 use thereof. Patent Application Publication Oct. 18, 2012 US 2012/0264.634 A1 US 2012/0264.634 A1 Oct. -

Table SI. Genes Upregulated ≥ 2-Fold by MIH 2.4Bl Treatment Affymetrix ID
Table SI. Genes upregulated 2-fold by MIH 2.4Bl treatment Fold UniGene ID Description Affymetrix ID Entrez Gene Change 1558048_x_at 28.84 Hs.551290 231597_x_at 17.02 Hs.720692 238825_at 10.19 93953 Hs.135167 acidic repeat containing (ACRC) 203821_at 9.82 1839 Hs.799 heparin binding EGF like growth factor (HBEGF) 1559509_at 9.41 Hs.656636 202957_at 9.06 3059 Hs.14601 hematopoietic cell-specific Lyn substrate 1 (HCLS1) 202388_at 8.11 5997 Hs.78944 regulator of G-protein signaling 2 (RGS2) 213649_at 7.9 6432 Hs.309090 serine and arginine rich splicing factor 7 (SRSF7) 228262_at 7.83 256714 Hs.127951 MAP7 domain containing 2 (MAP7D2) 38037_at 7.75 1839 Hs.799 heparin binding EGF like growth factor (HBEGF) 224549_x_at 7.6 202672_s_at 7.53 467 Hs.460 activating transcription factor 3 (ATF3) 243581_at 6.94 Hs.659284 239203_at 6.9 286006 Hs.396189 leucine rich single-pass membrane protein 1 (LSMEM1) 210800_at 6.7 1678 translocase of inner mitochondrial membrane 8 homolog A (yeast) (TIMM8A) 238956_at 6.48 1943 Hs.741510 ephrin A2 (EFNA2) 242918_at 6.22 4678 Hs.319334 nuclear autoantigenic sperm protein (NASP) 224254_x_at 6.06 243509_at 6 236832_at 5.89 221442 Hs.374076 adenylate cyclase 10, soluble pseudogene 1 (ADCY10P1) 234562_x_at 5.89 Hs.675414 214093_s_at 5.88 8880 Hs.567380; far upstream element binding protein 1 (FUBP1) Hs.707742 223774_at 5.59 677825 Hs.632377 small nucleolar RNA, H/ACA box 44 (SNORA44) 234723_x_at 5.48 Hs.677287 226419_s_at 5.41 6426 Hs.710026; serine and arginine rich splicing factor 1 (SRSF1) Hs.744140 228967_at 5.37 -

Rabbit Anti-CDCA5/FITC Conjugated Antibody
SunLong Biotech Co.,LTD Tel: 0086-571- 56623320 Fax:0086-571- 56623318 E-mail:[email protected] www.sunlongbiotech.com Rabbit Anti-CDCA5/FITC Conjugated antibody SL7717R-FITC Product Name: Anti-CDCA5/FITC Chinese Name: FITC标记的细胞分裂周期相关蛋白5抗体 Alias: Cell division cycle associated protein 5; MGC16386; p35; Sororin; CDCA5_HUMAN. Organism Species: Rabbit Clonality: Polyclonal React Species: Human,Mouse,Rat,Dog,Pig,Cow,Horse,Rabbit, Flow-Cyt=1:50-200IF=1:50-200 Applications: not yet tested in other applications. optimal dilutions/concentrations should be determined by the end user. Molecular weight: 28kDa Form: Lyophilized or Liquid Concentration: 1mg/ml immunogen: KLH conjugated synthetic peptide derived from human CDCA5 Lsotype: IgG Purification: affinity purified by Protein A Storage Buffer: 0.01M TBS(pH7.4) with 1% BSA, 0.03% Proclin300 and 50% Glycerol. Store at -20 °C for one year. Avoid repeated freeze/thaw cycles. The lyophilized antibodywww.sunlongbiotech.com is stable at room temperature for at least one month and for greater than a year Storage: when kept at -20°C. When reconstituted in sterile pH 7.4 0.01M PBS or diluent of antibody the antibody is stable for at least two weeks at 2-4 °C. background: Sororin, also designated cell division cycle-associated protein 5 (CDCA5) or p35, functions as a regulator of sister chromatid cohesion during mitosis. It interacts with the APC/C complex and is found in a complex consisting of cohesion components SCC- 112, MC1L1, SMC3L1, RAD21 and APRIN. The deduced human and mouse Sororin Product Detail: proteins consist of 252 and 264 amino acid residues, respectively, and both contain a KEN box for APC-dependent ubiquitination. -

Análise Correlacional Entre a Expressão Dos Fatores De Splicing E a Ocorrência De Splicing Alternativo Em Tecidos Humanos E De Camundongos
ANÁLISE CORRELACIONAL ENTRE A EXPRESSÃO DOS FATORES DE SPLICING E A OCORRÊNCIA DE SPLICING ALTERNATIVO EM TECIDOS HUMANOS E DE CAMUNDONGOS JULIO CÉSAR NUNES Dissertação apresentada à Fundação Antônio Prudente para a obtenção do título de Mestre em Ciências Área de Concentração: Oncologia Orientador: Dr. Sandro José de Souza São Paulo 2008 Livros Grátis http://www.livrosgratis.com.br Milhares de livros grátis para download. FICHA CATALOGRÁFICA Preparada pela Biblioteca da Fundação Antônio Prudente Nunes, Julio César Análise correlacional entre a expressão dos fatores de splicing e a ocorrência de splicing alternativo em tecidos humanos e de camundongos / Julio César Nunes – São Paulo, 2008. 79p. Dissertação (Mestrado) - Fundação Antônio Prudente. Curso de Pós-Graduação em Ciências - Área de concentração: Oncologia. Orientador: Sandro José Souza Descritores: 1. SPLICING ALTERNATIVO 2. BIOLOGIA MOLECULAR COMPUTACIONAL 3. CÂNCER 4. GENOMICA. AGRADECIMENTOS Agradeço à FAPESP e CAPES pela bolsa de Mestrado. Ao Sandro José de Souza agradeço toda orientação e conhecimento oferecido. Meus especiais agradecimentos ao Pedro Alexandre Favoretto Galante que dedicou atenção a minha formação no processo de Pós-Graduação na Fundação Antônio Prudente, bem como pela sua oficiosa co-orientação ao projeto de pesquisa. À grande família e amigos pela dedicação e incentivo a minha formação acadêmica. À Fundação Antônio Prudente, Hospital do Câncer e Instituto Ludwig de Pesquisa sobre o Câncer dedico os meus nobres agradecimentos finais. RESUMO Nunes JC. Análise correlacional entre a expressão dos fatores de splicing e a ocorrência de splicing alternativo em tecidos humanos e de camundongos. São Paulo; 2007. [Dissertacão de Mestrado - Fundação Antônio Prudente] Splicing alternativo desempenha uma significante função no aumento da complexidade genômica, produzindo um extenso número de mRNA e isoformas protéicas. -

Low Tolerance for Transcriptional Variation at Cohesin Genes Is Accompanied by Functional Links to Disease-Relevant Pathways
bioRxiv preprint doi: https://doi.org/10.1101/2020.04.11.037358; this version posted April 13, 2020. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY-NC-ND 4.0 International license. Title Low tolerance for transcriptional variation at cohesin genes is accompanied by functional links to disease-relevant pathways Authors William Schierdingǂ1, Julia Horsfieldǂ2,3, Justin O’Sullivan1,3,4 ǂTo whom correspondence should be addressed. 1 Liggins Institute, The University of Auckland, Auckland, New Zealand 2 Department of Pathology, Dunedin School of Medicine, University of Otago, Dunedin, New Zealand 3 The Maurice Wilkins Centre for Biodiscovery, The University of Auckland, Auckland, New Zealand 4 MRC Lifecourse Epidemiology Unit, University of Southampton Acknowledgements This work was supported by a Royal Society of New Zealand Marsden Grant to JH and JOS (16-UOO- 072), and WS was supported by the same grant. Contributions WS planned the study, performed analyses, and drafted the manuscript. JH and JOS revised the manuscript. Competing interests None declared. bioRxiv preprint doi: https://doi.org/10.1101/2020.04.11.037358; this version posted April 13, 2020. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY-NC-ND 4.0 International license. Abstract Variants in DNA regulatory elements can alter the regulation of distant genes through spatial- regulatory connections. -

Gene Regulation by Cohesin in Cancer: Is the Ring an Unexpected Party to Proliferation?
Published OnlineFirst September 22, 2011; DOI: 10.1158/1541-7786.MCR-11-0382 Molecular Cancer Review Research Gene Regulation by Cohesin in Cancer: Is the Ring an Unexpected Party to Proliferation? Jenny M. Rhodes, Miranda McEwan, and Julia A. Horsfield Abstract Cohesin is a multisubunit protein complex that plays an integral role in sister chromatid cohesion, DNA repair, and meiosis. Of significance, both over- and underexpression of cohesin are associated with cancer. It is generally believed that cohesin dysregulation contributes to cancer by leading to aneuploidy or chromosome instability. For cancers with loss of cohesin function, this idea seems plausible. However, overexpression of cohesin in cancer appears to be more significant for prognosis than its loss. Increased levels of cohesin subunits correlate with poor prognosis and resistance to drug, hormone, and radiation therapies. However, if there is sufficient cohesin for sister chromatid cohesion, overexpression of cohesin subunits should not obligatorily lead to aneuploidy. This raises the possibility that excess cohesin promotes cancer by alternative mechanisms. Over the last decade, it has emerged that cohesin regulates gene transcription. Recent studies have shown that gene regulation by cohesin contributes to stem cell pluripotency and cell differentiation. Of importance, cohesin positively regulates the transcription of genes known to be dysregulated in cancer, such as Runx1, Runx3, and Myc. Furthermore, cohesin binds with estrogen receptor a throughout the genome in breast cancer cells, suggesting that it may be involved in the transcription of estrogen-responsive genes. Here, we will review evidence supporting the idea that the gene regulation func- tion of cohesin represents a previously unrecognized mechanism for the development of cancer. -
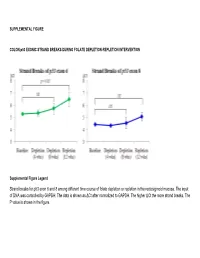
Strand Breaks for P53 Exon 6 and 8 Among Different Time Course of Folate Depletion Or Repletion in the Rectosigmoid Mucosa
SUPPLEMENTAL FIGURE COLON p53 EXONIC STRAND BREAKS DURING FOLATE DEPLETION-REPLETION INTERVENTION Supplemental Figure Legend Strand breaks for p53 exon 6 and 8 among different time course of folate depletion or repletion in the rectosigmoid mucosa. The input of DNA was controlled by GAPDH. The data is shown as ΔCt after normalized to GAPDH. The higher ΔCt the more strand breaks. The P value is shown in the figure. SUPPLEMENT S1 Genes that were significantly UPREGULATED after folate intervention (by unadjusted paired t-test), list is sorted by P value Gene Symbol Nucleotide P VALUE Description OLFM4 NM_006418 0.0000 Homo sapiens differentially expressed in hematopoietic lineages (GW112) mRNA. FMR1NB NM_152578 0.0000 Homo sapiens hypothetical protein FLJ25736 (FLJ25736) mRNA. IFI6 NM_002038 0.0001 Homo sapiens interferon alpha-inducible protein (clone IFI-6-16) (G1P3) transcript variant 1 mRNA. Homo sapiens UDP-N-acetyl-alpha-D-galactosamine:polypeptide N-acetylgalactosaminyltransferase 15 GALNTL5 NM_145292 0.0001 (GALNT15) mRNA. STIM2 NM_020860 0.0001 Homo sapiens stromal interaction molecule 2 (STIM2) mRNA. ZNF645 NM_152577 0.0002 Homo sapiens hypothetical protein FLJ25735 (FLJ25735) mRNA. ATP12A NM_001676 0.0002 Homo sapiens ATPase H+/K+ transporting nongastric alpha polypeptide (ATP12A) mRNA. U1SNRNPBP NM_007020 0.0003 Homo sapiens U1-snRNP binding protein homolog (U1SNRNPBP) transcript variant 1 mRNA. RNF125 NM_017831 0.0004 Homo sapiens ring finger protein 125 (RNF125) mRNA. FMNL1 NM_005892 0.0004 Homo sapiens formin-like (FMNL) mRNA. ISG15 NM_005101 0.0005 Homo sapiens interferon alpha-inducible protein (clone IFI-15K) (G1P2) mRNA. SLC6A14 NM_007231 0.0005 Homo sapiens solute carrier family 6 (neurotransmitter transporter) member 14 (SLC6A14) mRNA. -

Downloaded Per Proteome Cohort Via the Web- Site Links of Table 1, Also Providing Information on the Deposited Spectral Datasets
www.nature.com/scientificreports OPEN Assessment of a complete and classifed platelet proteome from genome‑wide transcripts of human platelets and megakaryocytes covering platelet functions Jingnan Huang1,2*, Frauke Swieringa1,2,9, Fiorella A. Solari2,9, Isabella Provenzale1, Luigi Grassi3, Ilaria De Simone1, Constance C. F. M. J. Baaten1,4, Rachel Cavill5, Albert Sickmann2,6,7,9, Mattia Frontini3,8,9 & Johan W. M. Heemskerk1,9* Novel platelet and megakaryocyte transcriptome analysis allows prediction of the full or theoretical proteome of a representative human platelet. Here, we integrated the established platelet proteomes from six cohorts of healthy subjects, encompassing 5.2 k proteins, with two novel genome‑wide transcriptomes (57.8 k mRNAs). For 14.8 k protein‑coding transcripts, we assigned the proteins to 21 UniProt‑based classes, based on their preferential intracellular localization and presumed function. This classifed transcriptome‑proteome profle of platelets revealed: (i) Absence of 37.2 k genome‑ wide transcripts. (ii) High quantitative similarity of platelet and megakaryocyte transcriptomes (R = 0.75) for 14.8 k protein‑coding genes, but not for 3.8 k RNA genes or 1.9 k pseudogenes (R = 0.43–0.54), suggesting redistribution of mRNAs upon platelet shedding from megakaryocytes. (iii) Copy numbers of 3.5 k proteins that were restricted in size by the corresponding transcript levels (iv) Near complete coverage of identifed proteins in the relevant transcriptome (log2fpkm > 0.20) except for plasma‑derived secretory proteins, pointing to adhesion and uptake of such proteins. (v) Underrepresentation in the identifed proteome of nuclear‑related, membrane and signaling proteins, as well proteins with low‑level transcripts. -

SWITCH 1/DYAD Is a Novel WINGS APART-LIKE Antagonist That
bioRxiv preprint doi: https://doi.org/10.1101/551317; this version posted February 15, 2019. The copyright holder for this preprint (which was not certified by peer review) is the author/funder. All rights reserved. No reuse allowed without permission. Title: SWITCH 1/DYAD is a novel WINGS APART-LIKE antagonist that maintains sister chromatid cohesion in meiosis Authors: Chao Yang1, Yuki Hamamura1, Kostika Sofroni1, Franziska Böwer1, Sara Christina Stolze2, Hirofumi Nakagami2 and Arp Schnittger1,* Affiliations: 1 Department of Developmental Biology, University of Hamburg, 22609 Hamburg, Germany. 2 Max-Planck-Institute for Plant Breeding Research, 50829 Cologne, Germany. *Correspondence to: [email protected] Mitosis and meiosis both rely on cohesin, which embraces the sister chromatids and plays a crucial role for the faithful distribution of chromosomes to daughter cells. Prior to the cleavage by Separase at anaphase onset, cohesin is largely removed from chromosomes by the non- proteolytic action of WINGS APART-LIKE (WAPL), a mechanism referred to as the prophase pathway. To prevent the premature loss of sister chromatid cohesion, WAPL is inhibited in early mitosis by Sororin. However, Sororin homologs have only been found to function as WAPL inhibitors during mitosis in vertebrates and Drosophila. Here we show that SWITCH 1/DYAD defines a novel WAPL antagonist that acts in meiosis of Arabidopsis. Crucially, SWI1 becomes dispensable for sister chromatid cohesion in the absence of WAPL. Despite the lack of any seQuence similarities, we found that SWI1 is regulated and functions in a similar 1 bioRxiv preprint doi: https://doi.org/10.1101/551317; this version posted February 15, 2019. -

Variation in Protein Coding Genes Identifies Information Flow
bioRxiv preprint doi: https://doi.org/10.1101/679456; this version posted June 21, 2019. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY-NC-ND 4.0 International license. Animal complexity and information flow 1 1 2 3 4 5 Variation in protein coding genes identifies information flow as a contributor to 6 animal complexity 7 8 Jack Dean, Daniela Lopes Cardoso and Colin Sharpe* 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 Institute of Biological and Biomedical Sciences 25 School of Biological Science 26 University of Portsmouth, 27 Portsmouth, UK 28 PO16 7YH 29 30 * Author for correspondence 31 [email protected] 32 33 Orcid numbers: 34 DLC: 0000-0003-2683-1745 35 CS: 0000-0002-5022-0840 36 37 38 39 40 41 42 43 44 45 46 47 48 49 Abstract bioRxiv preprint doi: https://doi.org/10.1101/679456; this version posted June 21, 2019. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY-NC-ND 4.0 International license. Animal complexity and information flow 2 1 Across the metazoans there is a trend towards greater organismal complexity. How 2 complexity is generated, however, is uncertain. Since C.elegans and humans have 3 approximately the same number of genes, the explanation will depend on how genes are 4 used, rather than their absolute number. -

Análise Correlacional Entre a Expressão Dos Fatores De Splicing E a Ocorrência De Splicing Alternativo Em Tecidos Humanos E De Camundongos
ANÁLISE CORRELACIONAL ENTRE A EXPRESSÃO DOS FATORES DE SPLICING E A OCORRÊNCIA DE SPLICING ALTERNATIVO EM TECIDOS HUMANOS E DE CAMUNDONGOS JULIO CÉSAR NUNES Dissertação apresentada à Fundação Antônio Prudente para a obtenção do título de Mestre em Ciências Área de Concentração: Oncologia Orientador: Dr. Sandro José de Souza São Paulo 2008 FICHA CATALOGRÁFICA Preparada pela Biblioteca da Fundação Antônio Prudente Nunes, Julio César Análise correlacional entre a expressão dos fatores de splicing e a ocorrência de splicing alternativo em tecidos humanos e de camundongos / Julio César Nunes – São Paulo, 2008. 79p. Dissertação (Mestrado) - Fundação Antônio Prudente. Curso de Pós-Graduação em Ciências - Área de concentração: Oncologia. Orientador: Sandro José Souza Descritores: 1. SPLICING ALTERNATIVO 2. BIOLOGIA MOLECULAR COMPUTACIONAL 3. CÂNCER 4. GENOMICA. AGRADECIMENTOS Agradeço à FAPESP e CAPES pela bolsa de Mestrado. Ao Sandro José de Souza agradeço toda orientação e conhecimento oferecido. Meus especiais agradecimentos ao Pedro Alexandre Favoretto Galante que dedicou atenção a minha formação no processo de Pós-Graduação na Fundação Antônio Prudente, bem como pela sua oficiosa co-orientação ao projeto de pesquisa. À grande família e amigos pela dedicação e incentivo a minha formação acadêmica. À Fundação Antônio Prudente, Hospital do Câncer e Instituto Ludwig de Pesquisa sobre o Câncer dedico os meus nobres agradecimentos finais. RESUMO Nunes JC. Análise correlacional entre a expressão dos fatores de splicing e a ocorrência de splicing alternativo em tecidos humanos e de camundongos. São Paulo; 2007. [Dissertacão de Mestrado - Fundação Antônio Prudente] Splicing alternativo desempenha uma significante função no aumento da complexidade genômica, produzindo um extenso número de mRNA e isoformas protéicas. -

Dissertation
DISSERTATION submitted to the Combined Faculties for the Natural Sciences and for Mathematics of the Ruperto-Carola University of Heidelberg, Germany for the degree of Doctor of Natural Sciences presented by Syafrizayanti born in Padang, Indonesia Oral examination: March 11, 2014 Production of Personalized Protein Microarrays Optimized production of protein microarrays and the establishment of processes for the representation of protein conformations that occur in individual patients Referees: Prof. Dr. Stefan Wiemann Prof. Dr. Stefan Wölfl Thesis declaration I hereby declare that this thesis has been written only by the undersigned and without any assistance from third parties. Furthermore, I confirm that no sources have been used in the preparation of this thesis other than those indicated in the thesis itself. Heidelberg, 2014 Syafrizayanti Part of this work has been accepted for publication: Syafrizayanti, Betzen C, Hoheisel JD, Kastelic D. Methods for analysing and quantifying protein-protein interactions. Exp. Rev. Prot. (2014) in press. Another manuscript will be submitted while this thesis is evaluated: Syafrizayanti, Lueong SS, Hoheisel JD. Production of personalized protein microarrays (2014). Lueong SS, Syafrizayanti, Hoheisel JD. Production of functional protein microarrays from cDNA products and functional applications (2014). Poster presentations Syafrizayanti, Lueong S, Hoheisel JD, Personalised proteomics by means of individualised protein microarrays, HUPO 12th Annual World Congress, Yokohama, September 2013 Bilen S,