Pseudomonas Aeruginosa Diego Carriel
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Screening and Identification of Key Biomarkers in Clear Cell Renal Cell Carcinoma Based on Bioinformatics Analysis
bioRxiv preprint doi: https://doi.org/10.1101/2020.12.21.423889; this version posted December 23, 2020. The copyright holder for this preprint (which was not certified by peer review) is the author/funder. All rights reserved. No reuse allowed without permission. Screening and identification of key biomarkers in clear cell renal cell carcinoma based on bioinformatics analysis Basavaraj Vastrad1, Chanabasayya Vastrad*2 , Iranna Kotturshetti 1. Department of Biochemistry, Basaveshwar College of Pharmacy, Gadag, Karnataka 582103, India. 2. Biostatistics and Bioinformatics, Chanabasava Nilaya, Bharthinagar, Dharwad 580001, Karanataka, India. 3. Department of Ayurveda, Rajiv Gandhi Education Society`s Ayurvedic Medical College, Ron, Karnataka 562209, India. * Chanabasayya Vastrad [email protected] Ph: +919480073398 Chanabasava Nilaya, Bharthinagar, Dharwad 580001 , Karanataka, India bioRxiv preprint doi: https://doi.org/10.1101/2020.12.21.423889; this version posted December 23, 2020. The copyright holder for this preprint (which was not certified by peer review) is the author/funder. All rights reserved. No reuse allowed without permission. Abstract Clear cell renal cell carcinoma (ccRCC) is one of the most common types of malignancy of the urinary system. The pathogenesis and effective diagnosis of ccRCC have become popular topics for research in the previous decade. In the current study, an integrated bioinformatics analysis was performed to identify core genes associated in ccRCC. An expression dataset (GSE105261) was downloaded from the Gene Expression Omnibus database, and included 26 ccRCC and 9 normal kideny samples. Assessment of the microarray dataset led to the recognition of differentially expressed genes (DEGs), which was subsequently used for pathway and gene ontology (GO) enrichment analysis. -

A Review on Agmatinase Inhibitors
www.ijcrt.org © 2020 IJCRT | Volume 8, Issue 12 December 2020 | ISSN: 2320-2882 A review on Agmatinase inhibitors 1Sunnica Biswas, 2Mr. R. T. Lohiya, 3Dr. Milind Umekar *1&2 Department Of Pharmaceutical Chemistry Smt. Kishoriti Bhoyar College of Pharmacy, Kamptee, Dst. Nagpur, 441002 Abstract : Agmatine is the product of arginine decarboxylation and can be hydrolyzed by agmatinase to putrescine, the precursor for biosynthesis of higher polyamines, spermidine, and spermine. Besides being an intermediate in polyamine metabolism, recent findings indicate that agmatine may play important regulatory roles in mammals. Agmatine, 4-aminobutyl guanidine, has recently been found in various mammalian organs and is thought to act as a neurotransmitter or neuromodulatory agent. The present study is to do a review on agmatine and its synthesized analogues till now for agmatinase inhibitory action. Agmatinase is a binuclear manganese metalloenzyme and belongs to the ureohydrolase superfamily that includes arginase, formiminoglutamase, and proclavaminate amidinohydrolase. Compared with a wealth of structural information available for arginases, no three dimensional structure of agmatinase has been reported. Agmatinase is an enzyme which blocks the mammalian agmatine which is ultimately responsible for the agmatine degradation in the body. Agmatinase is an enzyme which regulates the half life of agmatine in the brain. Hence a selective inhibitor of brain agmatinase is required. Several derivatives of agmatine are synthesized previously for agmatinase inhibitory activity but none of them showed selective inhibition. PZC (Piperazinecarboxamidine) is a derivative of agmatine or guanidine is expected to show selective inhibition of human agmatinase. A detailed review is carried out in order to understand the agmatinase inhibitor. -

Screening of a Composite Library of Clinically Used Drugs and Well
HHS Public Access Author manuscript Author ManuscriptAuthor Manuscript Author Pharmacol Manuscript Author Res. Author Manuscript Author manuscript; available in PMC 2017 November 01. Published in final edited form as: Pharmacol Res. 2016 November ; 113(Pt A): 18–37. doi:10.1016/j.phrs.2016.08.016. Screening of a composite library of clinically used drugs and well-characterized pharmacological compounds for cystathionine β-synthase inhibition identifies benserazide as a drug potentially suitable for repurposing for the experimental therapy of colon cancer Nadiya Druzhynaa, Bartosz Szczesnya, Gabor Olaha, Katalin Módisa,b, Antonia Asimakopoulouc,d, Athanasia Pavlidoue, Petra Szoleczkya, Domokos Geröa, Kazunori Yanagia, Gabor Töröa, Isabel López-Garcíaa, Vassilios Myrianthopoulose, Emmanuel Mikrose, John R. Zatarainb, Celia Chaob, Andreas Papapetropoulosd,e, Mark R. Hellmichb,f, and Csaba Szaboa,f,* aDepartment of Anesthesiology, The University of Texas Medical Branch, Galveston, TX, USA bDepartment of Surgery, The University of Texas Medical Branch, Galveston, TX, USA cLaboratory of Molecular Pharmacology, Department of Pharmacy, University of Patras, Greece dCenter of Clinical, Experimental Surgery & Translational Research, Biomedical Research Foundation of the Academy of Athens, Greece eNational and Kapodistrian University of Athens, School of Pharmacy, Athens, Greece fCBS Therapeutics Inc., Galveston, TX, USA Abstract Cystathionine-β-synthase (CBS) has been recently identified as a drug target for several forms of cancer. Currently no -

Computational Prediction and Character Ization of Genomic Islands: Insights Into Bacterial Pathogenicity
COMPUTATIONAL PREDICTION AND CHARACTER IZATION OF GENOMIC ISLANDS: INSIGHTS INTO BACTERIAL PATHOGENICITY by Morgan Gavel Ira Langille B.Sc. University of New Brunswick, 2004 B.CS University of New Brunswick, 2004 THESIS SUBMITTED IN PARTIAL FULFILLMENT OF THE REQUIREMENTS FOR THE DEGREE OF DOCTOR OF PHILOSOPHY In the Department of Molecular Biology and Biochemistry © Morgan Gavel Ira Langille 2009 SIMON FRASER UNIVERSITY Summer 2009 All rights reserved. This work may not be reproduced in whole or in part, by photocopy or other means, without permission of the author. APPROVAL Name: Morgan Gavel Ira Langille Degree: Doctor of Philosophy Title of Thesis: Computational prediction and characterization of genomic islands: insights into bacterial pathogenicity Examining Committee: Chair: Dr. Paul C.H. Li Associate Professor, Department of Chemistry ______________________________________ Dr. Fiona S.L. Brinkman Senior Supervisor Associate Professor of Molecular Biology and Biochemistry ______________________________________ Dr. David L. Baillie Supervisor Professor of Molecular Biology and Biochemistry ______________________________________ Dr. Frederic F. Pio Supervisor Assistant Professor of Molecular Biology and Biochemistry ______________________________________ Dr. Jack Chen Internal Examiner Associate Professor of Molecular Biology and Biochemistry ______________________________________ Dr. Steven Hallam External Examiner Assistant Professor of Microbiology and Immunology, University of British Columbia Date Defended/Approved: Thursday April 16, 2009 ii Declaration of Partial Copyright Licence The author, whose copyright is declared on the title page of this work, has granted to Simon Fraser University the right to lend this thesis, project or extended essay to users of the Simon Fraser University Library, and to make partial or single copies only for such users or in response to a request from the library of any other university, or other educational institution, on its own behalf or for one of its users. -

Pyoverdine Production in the Pathogen Pseudomonas Aeruginosa: a Study on Cooperative Interactions Among Individuals and Its Role for Virulence
Ludwig-Maximilans-Universtiät München Pyoverdine production in the pathogen Pseudomonas aeruginosa: a study on cooperative interactions among individuals and its role for virulence Dissertation der Fakultät für Biologie der Ludwig-Maximilians-Universität München vorgelegt von Michael Weigert aus Straubing April 2017 Gutachter: 1. Prof. Kirsten Jung 2. Prof. Kai Papenfort Tag der Abgabe: 03.04.2017 Tag der mündlichen Prüfung: 09.06.2017 I Contents ContentsII 1 PrefaceV 1.1 Eidesstattliche Erklärung/Statutory Declaration . .V 1.2 Publications and Manuscripts Originating From this Thesis . .VI 1.3 Contributions to Publications and Manuscripts Presented in this Thesis . VII 1.4 Abbreviations . .IX 1.5 Summary . 10 1.6 Zusammenfassung . 12 2 Introduction 15 2.1 Cooperation in Bacteria . 16 2.1.1 Bacterial Cooperation and the Problem of Cheating . 17 2.2 Quorum Sensing . 18 2.3 Siderophores . 19 2.3.1 The Siderophores of Pseudomonas aeruginosa ........ 19 2.3.2 Pyoverdine and Virulence . 20 2.3.3 Regulation of Pyoverdine-Expression . 22 2.4 New Approaches to Fight Multi-Drug-Resistant Pathogens . 24 2.4.1 The Problem of Antibiotic Resistance . 24 2.4.2 Anti-Virulence Treatments . 26 II 2.4.3 The Evolution of Resistance to Anti-Virulence Treatments 29 2.5 Evolution Proof Drugs . 30 2.5.1 Targeting a Secreted Virulence Factor . 31 2.5.2 Targeting a Cooperatively Shared Virulence Factor . 32 2.6 Aims of this Thesis . 32 3 Gallium-Mediated Siderophore Quenching as an Evolutionarily Ro- bust Antibacterial Treatment 35 3.1 Supporting Material . 48 4 Manipulating Virulence Factor Availability Can Have Complex Con- sequences for Infections 51 4.1 Supporting Material . -

(Batch Learning Self-Organizing Maps), to the Microbiome Analysis of Ticks
Title A novel approach, based on BLSOMs (Batch Learning Self-Organizing Maps), to the microbiome analysis of ticks Nakao, Ryo; Abe, Takashi; Nijhof, Ard M; Yamamoto, Seigo; Jongejan, Frans; Ikemura, Toshimichi; Sugimoto, Author(s) Chihiro The ISME Journal, 7(5), 1003-1015 Citation https://doi.org/10.1038/ismej.2012.171 Issue Date 2013-03 Doc URL http://hdl.handle.net/2115/53167 Type article (author version) File Information ISME_Nakao.pdf Instructions for use Hokkaido University Collection of Scholarly and Academic Papers : HUSCAP A novel approach, based on BLSOMs (Batch Learning Self-Organizing Maps), to the microbiome analysis of ticks Ryo Nakao1,a, Takashi Abe2,3,a, Ard M. Nijhof4, Seigo Yamamoto5, Frans Jongejan6,7, Toshimichi Ikemura2, Chihiro Sugimoto1 1Division of Collaboration and Education, Research Center for Zoonosis Control, Hokkaido University, Kita-20, Nishi-10, Kita-ku, Sapporo, Hokkaido 001-0020, Japan 2Nagahama Institute of Bio-Science and Technology, Nagahama, Shiga 526-0829, Japan 3Graduate School of Science & Technology, Niigata University, 8050, Igarashi 2-no-cho, Nishi- ku, Niigata 950-2181, Japan 4Institute for Parasitology and Tropical Veterinary Medicine, Freie Universität Berlin, Königsweg 67, 14163 Berlin, Germany 5Miyazaki Prefectural Institute for Public Health and Environment, 2-3-2 Gakuen Kibanadai Nishi, Miyazaki 889-2155, Japan 6Utrecht Centre for Tick-borne Diseases (UCTD), Department of Infectious Diseases and Immunology, Faculty of Veterinary Medicine, Utrecht University, Yalelaan 1, 3584 CL Utrecht, The Netherlands 7Department of Veterinary Tropical Diseases, Faculty of Veterinary Science, University of Pretoria, Private Bag X04, 0110 Onderstepoort, South Africa aThese authors contributed equally to this work. Keywords: BLSOMs/emerging diseases/metagenomics/microbiomes/symbionts/ticks Running title: Tick microbiomes revealed by BLSOMs Subject category: Microbe-microbe and microbe-host interactions Abstract Ticks transmit a variety of viral, bacterial and protozoal pathogens, which are often zoonotic. -

Agmatinase Sirna (H): Sc-60060
SANTA CRUZ BIOTECHNOLOGY, INC. Agmatinase siRNA (h): sc-60060 BACKGROUND STORAGE AND RESUSPENSION Agmatinase (also known as agmatine ureohydrolase) results from the decar- Store lyophilized siRNA duplex at -20° C with desiccant. Stable for at least boxylation of L-arginine by arginine decarboxylase to form a metabolic inter- one year from the date of shipment. Once resuspended, store at -20° C, mediate in the biosynthesis of putresine and higher polyamines (spermidine avoid contact with RNAses and repeated freeze thaw cycles. and spermine). Agmatinase has been shown to play a role in several important Resuspend lyophilized siRNA duplex in 330 µl of the RNAse-free water biochemical processes in humans, ranging from effects on the central nervous provided. Resuspension of the siRNA duplex in 330 µl of RNAse-free water system to cell proliferation in cancer and viral replication. Agmatinase cat- makes a 10 µM solution in a 10 µM Tris-HCl, pH 8.0, 20 mM NaCl, 1 mM alyzes the hydrolysis of agmatine to putresine and urea and is a major target EDTA buffered solution. for drug therapy. Human Agmatinase retains about 30% identity to bacterial agmatinases and less than 20% identity to mammalian arginases. Residues APPLICATIONS required for binding of Mn2+ at the active site in bacterial Agmatinase and other members of the arginase superfamily are fully conserved in human Agmatinase siRNA (h) is recommended for the inhibition of Agmatinase Agmatinase. Agmatinase mRNA is most abundant in human liver and kidney, expression in human cells. but is also expressed in several other tissues, including skeletal muscle and brain. -
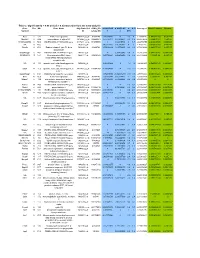
Table 2. Significant
Table 2. Significant (Q < 0.05 and |d | > 0.5) transcripts from the meta-analysis Gene Chr Mb Gene Name Affy ProbeSet cDNA_IDs d HAP/LAP d HAP/LAP d d IS Average d Ztest P values Q-value Symbol ID (study #5) 1 2 STS B2m 2 122 beta-2 microglobulin 1452428_a_at AI848245 1.75334941 4 3.2 4 3.2316485 1.07398E-09 5.69E-08 Man2b1 8 84.4 mannosidase 2, alpha B1 1416340_a_at H4049B01 3.75722111 3.87309653 2.1 1.6 2.84852656 5.32443E-07 1.58E-05 1110032A03Rik 9 50.9 RIKEN cDNA 1110032A03 gene 1417211_a_at H4035E05 4 1.66015788 4 1.7 2.82772795 2.94266E-05 0.000527 NA 9 48.5 --- 1456111_at 3.43701477 1.85785922 4 2 2.8237185 9.97969E-08 3.48E-06 Scn4b 9 45.3 Sodium channel, type IV, beta 1434008_at AI844796 3.79536664 1.63774235 3.3 2.3 2.75319499 1.48057E-08 6.21E-07 polypeptide Gadd45gip1 8 84.1 RIKEN cDNA 2310040G17 gene 1417619_at 4 3.38875643 1.4 2 2.69163229 8.84279E-06 0.0001904 BC056474 15 12.1 Mus musculus cDNA clone 1424117_at H3030A06 3.95752801 2.42838452 1.9 2.2 2.62132809 1.3344E-08 5.66E-07 MGC:67360 IMAGE:6823629, complete cds NA 4 153 guanine nucleotide binding protein, 1454696_at -3.46081884 -4 -1.3 -1.6 -2.6026947 8.58458E-05 0.0012617 beta 1 Gnb1 4 153 guanine nucleotide binding protein, 1417432_a_at H3094D02 -3.13334396 -4 -1.6 -1.7 -2.5946297 1.04542E-05 0.0002202 beta 1 Gadd45gip1 8 84.1 RAD23a homolog (S. -

Asparaginase and Glutaminase Activities of Micro-Organisms
Journal of General MicrobiologjI (1973),76,85-99 85 Printed in Great Britain Asparaginase and Glutaminase Activities of Micro-organisms By A. IMADA, S. IGARASI, K. NAKAHAMA AND M. ISONO Microbiological Research Laboratories, Central Research Dillision, Takeda Chemical Industries, JlisB, Osaka, Japan (Received 14 September 1972; revised 28 November 1972) SUMMARY L-Asparaginase and L-glutaminase activities were detected in many micro- organisms and the distribution of these activities was found to be related to the classification of micro-organisms. Among 464 bacteria, the activities occurred in many Gram-negative bacteria and in a few Gram-positive bacteria. Most members of the family Enterobacteri- aceae possessed L-asparaginase. L-Asparaginase and L-glutaminase occurred together in a large proportion of pseudomonads. Among Gram-positive bacteria many strains of Bacillus pumilus showed strong L-asparaginase activity. Amidase activities were also observed in several strains in other families. L-Asparaginase activity was not detected in culture filtrates of 261 strains of species of the genera Streptomyces and Nocardia, but L-asparaginase and L- glutaminase were detected when these organisms were sonicated. The amidase activities in culture filtrates of 4158 fungal strains were tested. All the strains of Fusarium species formed L-asparaginase. Organisms of the genera Hjyomyces and Nectria, which are regarded as the perfect stage of the genus Fusarium, also formed L-asparaginase. Several Penicillium species formed L-asparaginase. Two organisms of the family Moniliaceae formed L-glutaminase together with L-asparaginase, and a few ascomycetous fungi formed L-asparaginase or L-glutaminase. Among I 326 yeasts, L-asparaginase or L-glutaminase occurred frequently in certain serological groups of yeasts : VI (Hansenula) group, Cryptococcus group and Rhodotorula group. -

Downloaded 13 April 2017); Using Diamond
bioRxiv preprint doi: https://doi.org/10.1101/347021; this version posted June 14, 2018. The copyright holder for this preprint (which was not certified by peer review) is the author/funder. All rights reserved. No reuse allowed without permission. 1 2 3 4 5 Re-evaluating the salty divide: phylogenetic specificity of 6 transitions between marine and freshwater systems 7 8 9 10 Sara F. Pavera, Daniel J. Muratorea, Ryan J. Newtonb, Maureen L. Colemana# 11 a 12 Department of the Geophysical Sciences, University of Chicago, Chicago, Illinois, USA 13 b School of Freshwater Sciences, University of Wisconsin Milwaukee, Milwaukee, Wisconsin, USA 14 15 Running title: Marine-freshwater phylogenetic specificity 16 17 #Address correspondence to Maureen Coleman, [email protected] 18 bioRxiv preprint doi: https://doi.org/10.1101/347021; this version posted June 14, 2018. The copyright holder for this preprint (which was not certified by peer review) is the author/funder. All rights reserved. No reuse allowed without permission. 19 Abstract 20 Marine and freshwater microbial communities are phylogenetically distinct and transitions 21 between habitat types are thought to be infrequent. We compared the phylogenetic diversity of 22 marine and freshwater microorganisms and identified specific lineages exhibiting notably high or 23 low similarity between marine and freshwater ecosystems using a meta-analysis of 16S rRNA 24 gene tag-sequencing datasets. As expected, marine and freshwater microbial communities 25 differed in the relative abundance of major phyla and contained habitat-specific lineages; at the 26 same time, however, many shared taxa were observed in both environments. 27 Betaproteobacteria and Alphaproteobacteria sequences had the highest similarity between 28 marine and freshwater sample pairs. -

Supplementary Table S1. Table 1. List of Bacterial Strains Used in This Study Suppl
Supplementary Material Supplementary Tables: Supplementary Table S1. Table 1. List of bacterial strains used in this study Supplementary Table S2. List of plasmids used in this study Supplementary Table 3. List of primers used for mutagenesis of P. intermedia Supplementary Table 4. List of primers used for qRT-PCR analysis in P. intermedia Supplementary Table 5. List of the most highly upregulated genes in P. intermedia OxyR mutant Supplementary Table 6. List of the most highly downregulated genes in P. intermedia OxyR mutant Supplementary Table 7. List of the most highly upregulated genes in P. intermedia grown in iron-deplete conditions Supplementary Table 8. List of the most highly downregulated genes in P. intermedia grown in iron-deplete conditions Supplementary Figures: Supplementary Figure 1. Comparison of the genomic loci encoding OxyR in Prevotella species. Supplementary Figure 2. Distribution of SOD and glutathione peroxidase genes within the genus Prevotella. Supplementary Table S1. Bacterial strains Strain Description Source or reference P. intermedia V3147 Wild type OMA14 isolated from the (1) periodontal pocket of a Japanese patient with periodontitis V3203 OMA14 PIOMA14_I_0073(oxyR)::ermF This study E. coli XL-1 Blue Host strain for cloning Stratagene S17-1 RP-4-2-Tc::Mu aph::Tn7 recA, Smr (2) 1 Supplementary Table S2. Plasmids Plasmid Relevant property Source or reference pUC118 Takara pBSSK pNDR-Dual Clonetech pTCB Apr Tcr, E. coli-Bacteroides shuttle vector (3) plasmid pKD954 Contains the Porpyromonas gulae catalase (4) -

Progress Toward Understanding the Contribution of Alkali Generation in Dental Biofilms to Inhibition of Dental Caries
International Journal of Oral Science (2012) 4, 135–140 ß 2012 WCSS. All rights reserved 1674-2818/12 www.nature.com/ijos REVIEW Progress toward understanding the contribution of alkali generation in dental biofilms to inhibition of dental caries Ya-Ling Liu1, Marcelle Nascimento2 and Robert A Burne1 Alkali production by oral bacteria is believed to have a major impact on oral microbial ecology and to be inibitory to the initiation and progression of dental caries. A substantial body of evidence is beginning to accumulate that indicates the modulation of the alkalinogenic potential of dental biofilms may be a promising strategy for caries control. This brief review highlights recent progress toward understanding molecular genetic and physiologic aspects of important alkali-generating pathways in oral bacteria, and the role of alkali production in the ecology of dental biofilms in health and disease. International Journal of Oral Science (2012) 4, 135–140; doi:10.1038/ijos.2012.54; published online 21 September 2012 Keywords: arginine; biofilm; dental caries; microbial ecology; urea INTRODUCTION via the arginine deiminase system (ADS).15–18 Urea is provided con- Dental biofilms, the microbial communities that colonize the surfaces tinuously in salivary secretions and gingival exudates at concentra- of the teeth, exist in a dynamic equilibrium with host defenses and are tions roughly equivalent to those in serum, which range from about 3 generally compatible with the integrity of the tissues they colonize.1–4 to10 mmol?L21 in healthy humans. Urea is rapidly converted to A strong correlation is evident between the compositional and meta- ammonia and CO2 by bacterial ureases (Figure 1), which are produced bolic changes of the dental biofilms and the transition from oral health by a small subset of oral bacteria that includes Streptococcus salivarius, 2,5 to disease states, including dental caries and periodontal disease.