Progetto Equinbio
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

List of Horse Breeds 1 List of Horse Breeds
List of horse breeds 1 List of horse breeds This page is a list of horse and pony breeds, and also includes terms used to describe types of horse that are not breeds but are commonly mistaken for breeds. While there is no scientifically accepted definition of the term "breed,"[1] a breed is defined generally as having distinct true-breeding characteristics over a number of generations; its members may be called "purebred". In most cases, bloodlines of horse breeds are recorded with a breed registry. However, in horses, the concept is somewhat flexible, as open stud books are created for developing horse breeds that are not yet fully true-breeding. Registries also are considered the authority as to whether a given breed is listed as Light or saddle horse breeds a "horse" or a "pony". There are also a number of "color breed", sport horse, and gaited horse registries for horses with various phenotypes or other traits, which admit any animal fitting a given set of physical characteristics, even if there is little or no evidence of the trait being a true-breeding characteristic. Other recording entities or specialty organizations may recognize horses from multiple breeds, thus, for the purposes of this article, such animals are classified as a "type" rather than a "breed". The breeds and types listed here are those that already have a Wikipedia article. For a more extensive list, see the List of all horse breeds in DAD-IS. Heavy or draft horse breeds For additional information, see horse breed, horse breeding and the individual articles listed below. -
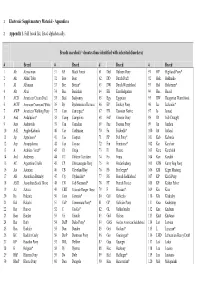
Electronic Supplementary Material - Appendices
1 Electronic Supplementary Material - Appendices 2 Appendix 1. Full breed list, listed alphabetically. Breeds searched (* denotes those identified with inherited disorders) # Breed # Breed # Breed # Breed 1 Ab Abyssinian 31 BF Black Forest 61 Dul Dülmen Pony 91 HP Highland Pony* 2 Ak Akhal Teke 32 Boe Boer 62 DD Dutch Draft 92 Hok Hokkaido 3 Al Albanian 33 Bre Breton* 63 DW Dutch Warmblood 93 Hol Holsteiner* 4 Alt Altai 34 Buc Buckskin 64 EB East Bulgarian 94 Huc Hucul 5 ACD American Cream Draft 35 Bud Budyonny 65 Egy Egyptian 95 HW Hungarian Warmblood 6 ACW American Creme and White 36 By Byelorussian Harness 66 EP Eriskay Pony 96 Ice Icelandic* 7 AWP American Walking Pony 37 Cam Camargue* 67 EN Estonian Native 97 Io Iomud 8 And Andalusian* 38 Camp Campolina 68 ExP Exmoor Pony 98 ID Irish Draught 9 Anv Andravida 39 Can Canadian 69 Fae Faeroes Pony 99 Jin Jinzhou 10 A-K Anglo-Kabarda 40 Car Carthusian 70 Fa Falabella* 100 Jut Jutland 11 Ap Appaloosa* 41 Cas Caspian 71 FP Fell Pony* 101 Kab Kabarda 12 Arp Araappaloosa 42 Cay Cayuse 72 Fin Finnhorse* 102 Kar Karabair 13 A Arabian / Arab* 43 Ch Cheju 73 Fl Fleuve 103 Kara Karabakh 14 Ard Ardennes 44 CC Chilean Corralero 74 Fo Fouta 104 Kaz Kazakh 15 AC Argentine Criollo 45 CP Chincoteague Pony 75 Fr Frederiksborg 105 KPB Kerry Bog Pony 16 Ast Asturian 46 CB Cleveland Bay 76 Fb Freiberger* 106 KM Kiger Mustang 17 AB Australian Brumby 47 Cly Clydesdale* 77 FS French Saddlebred 107 KP Kirdi Pony 18 ASH Australian Stock Horse 48 CN Cob Normand* 78 FT French Trotter 108 KF Kisber Felver 19 Az Azteca -

AUSTRALIAN LIPIZZANER REGISTRY INC. Registry for Australia, New Zealand and Malaysia “Striving for Breeding Excellence” Issue 15 August 2017
AUSTRALIAN LIPIZZANER REGISTRY INC. Registry for Australia, New Zealand and Malaysia “Striving for Breeding Excellence” Issue 15 August 2017 AUSTRALIAN LIPIZZANER REGISTRY INC. ALR Newsletter Issue 15 August 2017 Hello Lipizzaner enthusiasts! Our organisation continues to grow and a huge welcome is extended to all our new members. We look forward to supporting you in breeding, information, horse registration or performance; whichever discipline you should choose. Thank you so much for your support of this wonderful breed! The Australian Lipizzaner Registry Inc. Annual General meetings will be brought forward in the future, due to show and other commitments from members during the warmer months of the year. The meeting this year was held at Phoenix Park Stud in SA. We were shown many Lipizzaner horses, stallions, colts, fillies, mares and geldings, and the AGM was attended by some of our newest members. A great day was had by all. In Contents: Young stock are now “coming of age” and ready to get out and about. To assist with this task we are blessed with continued visits from some talented The Murgese Breed international riders aiming to pass on some of their wonderful knowledge. Asia Pacific Out and About Hungarian horsewoman, Edit Kappel is due to visit Australia again this International Out and About November and she will be running a series of clinics for both dressage and liberty work. Young horses in work For Sale Contact the ALR Header photo: Mares Montebella and Gidrane (left) grazing at Centaur Connecton in Queensland left with the foal Galatea out of Gidrane foreground. -

Mitochondrial DNA Lineages of Italian Giara and Sarcidano Horses
Mitochondrial DNA lineages of Italian Giara and Sarcidano horses L. Morelli1†, A. Useli1, D. Sanna1, M. Barbato1,2, D. Contu3, M. Pala4,5, M. Cancedda6 and P. Francalacci1 1Dipartimento di Scienze della Natura e del Territorio, Unità di Zoologia Archeozoologia e Genetica, Università di Sassari, Sassari, Italy 2Cardiff School of Biosciences, Cardiff University, Cardiff, UK 3Laboratorio di Immunogenetica, Ospedale Microcitemico, Cagliari, Italy 4Dipartimento di Biologia e Biotecnologie “L. Spallanzani” Università di Pavia, Pavia, Italy 5School of Applied Sciences, University of Huddersfield, Queensgate, Huddersfield, UK 6Dipartimento di Medicina Veterinaria, Università di Sassari, Sassari, Italy †This paper is dedicated to the memory of Laura Morelli who prematurely passed away before the publication. Corresponding author: A. Useli E-mail: [email protected] Genet. Mol. Res. 13 (4): 8241-8257 (2014) Received December 18, 2012 Accepted June 14, 2013 Published October 20, 2014 DOI http://dx.doi.org/10.4238/2014.October.20.1 ABSTRACT. Giara and Sarcidano are 2 of the 15 extant native Italian horse breeds with limited dispersal capability that originated from a larger number of individuals. The 2 breeds live in two distinct isolated locations on the island of Sardinia. To determine the genetic structure and evolutionary history of these 2 Sardinian breeds, the first hypervariable segment of the mitochondrial DNA (mtDNA) was sequenced and analyzed in 40 Giara and Sarcidano horses and compared with publicly available mtDNA data from 43 Old World breeds. Four Genetics and Molecular Research 13 (4): 8241-8257 (2014) ©FUNPEC-RP www.funpecrp.com.br different analyses, including genetic distance, analysis of molecular variance, haplotype sharing, and clustering methods, were used to study the genetic relationships between the Sardinian and other horse breeds. -

20091112 Organismi Passaporto Equidi
Ministero delle politiche agricole alimentari e forestali Dipartimento delle Politiche competitive del mondo rurale e della qualità Ex Direzione Generale dello Sviluppo Rurale, delle Infrastrutture e dei Servizi Ufficio SVIRIS X - Produzioni Animali - Dirigente: Francesco Scala Tel. 06 46655098-46655096 - 06 484459 Fax. 06 46655132 e-mail: [email protected] web: www.politicheagricole.it ELENCO ORGANISMI EMITTENTI PASSAPORTO EQUIDI REG. (CE) N. 504/2008 ART. 4 COMMA 5 LIST OF AGENCIES RELEASING EQUINE PASSPORT REG. (CE) N.504/2008 ART.4, COMMA 5 Codice UELN Libro genealogico/ Razze Contatti Associazione/Organizzazione UELN code Registro anagrafico Races Contacts Associations/Organizations Stud book/population register AIA-Associazione Italiana 380001 Registro Anagrafico Equine: Dr. Giancarlo Allevatori razze equine ed Cavallino della Giara Carchedi Via Tomassetti, 9 asinine a limitata Cavallino di Monterufoli 00161 ROMA diffusione Cavallo del Catria Cavallo del Delta +39 06 854511 Cavallo del Ventasso Cavallo Pentro +39 06 85451322 Cavallo Sarcidano Napoletano @ [email protected] Norico-Pinzgauer Persano www www.aia.it Pony di Esperia Sanfratellano Salernitano Tolfetano Asinine: Asino dell’Amiata Asino dell’Asinara Asino di Martina Franca Asino Ragusano Asino Romagnolo Asino Pantesco Asino Sardo AIA-Associazione Italiana 380001 Libro genealogico Murgese Dr. Giancarlo Allevatori cavallo Murgese Carchedi Via Tomassetti, 9 00161 ROMA +39 06 854511 +39 06 85451322 @ [email protected] www www.aia.it ANACRHAI-Associazione 380002 Libro -

A.N.A.R.E.A.I
A.N.A.R.E.A.I. ASSOCIAZIONE NAZIONALE ALLEVATORI DELLE RAZZE EQUINE ED ASININE ITALIANE Via XXIV Maggio n° 44/45 – 00187 - Roma – C.F. e P.IVA 15689641007 Cod. AUA (a cura di ANAREAI) DOMANDA ADESIONE A SOCIO (da spedire in ORIGINALE) ______________________________ Il sottoscritto ________________________________________________ nato il ______/______/______ a ____________________________________ Residente a _____________________________________ _____________________________________________________ Prov.___________________________ In Via ________________________________________ C.F. ___________________________________ In qualità di : Proprietario Conduttore Legale rappresentante dell’azienda: □ Ragione Sociale : Spazio per il timbro aziendale ___________________________________________ Sede Legale:_________________________________ ___________________________________________ ___________________________________________ Allegare copia CCIAA P.Iva_______________________________________ C. Fiscale______________________________________ Ricevimento documenti fiscali indicare eventuale Codice Univoco _____________________________ □ Altro soggetto / Privato : C. Fiscale_________________________________________ e-mail*______________________________________ PEC *___________________________________ tel.*____________________________ cell.* ____________________________fax. ________________ Allevamento sito in Via/Loc* _____________________________________________ CAP* _________ Comune* _______________________________________________________ Prov.*_______________ -

Piroplasmosis Equina En Áreas Endémicas Como La Península Itálica E Ibérica
UNIVERSIDAD COMPLUTENSE DE MADRID FACULTAD DE VETERINARIA Departamento de Sanidad Animal TESIS DOCTORAL Situación epidemiológica y clínica de la piroplasmosis equina en áreas endémicas como la Península Itálica e Ibérica Epidemiological and clinical situation of equine piroplasmosis in endemic areas such as Italian and Iberian peninsulas / MEMORIA PARA OPTAR AL GRADO DE DOCTOR PRESENTADA POR Leticia Elisa Bartolomé del Pino Directores Aránzazu Meana Mañes Gian Luca Autorino Madrid, 2017 ISBN: 978-84-09-05010-9 © Leticia Elisa Bartolomé del Pino, 2017 UNIVERSIDAD COMPLUTENSE DE MADRID FACULTAD DE VETERINARIA DEPARTAMENTO DE SANIDAD ANIMAL SITUACIÓN EPIDEMIOLÓGICA Y CLÍNICA DE LA PIROPLASMOSIS EQUINA EN ÁREAS ENDÉMICAS COMO LAS PENÍNSULAS ITÁLICA E IBÉRICA EPIDEMIOLOGICAL AND CLINICAL SITUATION OF EQUINE PIROPLASMOSIS IN ENDEMIC AREAS SUCH AS ITALIAN AND IBERIAN PENINSULAS TESIS DOCTORAL LETICIA ELISA BARTOLOMÉ DEL PINO MADRID, 2017 1 UNIVERSIDAD COMPLUTENSE DE MADRID FACULTAD DE VETERINARIA DEPARTAMENTO DE SANIDAD ANIMAL SITUACIÓN EPIDEMIOLÓGICA Y CLÍNICA DE LA PIROPLASMOSIS EQUINA EN ÁREAS ENDÉMICAS COMO LAS PENÍNSULAS ITÁLICA E IBÉRICA EPIDEMIOLOGICAL AND CLINICAL SITUATION OF EQUINE PIROPLASMOSIS IN ENDEMIC AREAS SUCH AS ITALIAN AND IBERIAN PENINSULAS MEMORIA PARA OPTAR AL GRADO DE DOCTOR PRESENTADA POR LETICIA ELISA BARTOLOMÉ DEL PINO DIRECTORES ARÁNZAZU MEANA MAÑES GIAN LUCA AUTORINO MADRID, 2017 Leticia E. Bartolomé del Pino, 2017 3 UNIVERSIDAD COMPLUTENSE DE MADRID FACULTAD DE VETERINARIA DEPARTAMENTO DE SANIDAD ANIMAL SITUACIÓN -

The Myostatin Gene: an Overview of Mechanisms of Action and Its Relevance to Livestock Animals
The Myostatin gene: an overview of mechanisms of action and its relevance to livestock animals Article Accepted Version Aiello, D., Patel, K. and Lasagna, E. (2018) The Myostatin gene: an overview of mechanisms of action and its relevance to livestock animals. Animal Genetics, 49 (6). pp. 505-519. ISSN 1365-2052 doi: https://doi.org/10.1111/age.12696 Available at http://centaur.reading.ac.uk/77388/ It is advisable to refer to the publisher’s version if you intend to cite from the work. See Guidance on citing . To link to this article DOI: http://dx.doi.org/10.1111/age.12696 Publisher: Wiley All outputs in CentAUR are protected by Intellectual Property Rights law, including copyright law. Copyright and IPR is retained by the creators or other copyright holders. Terms and conditions for use of this material are defined in the End User Agreement . www.reading.ac.uk/centaur CentAUR Central Archive at the University of Reading Reading’s research outputs online 1 Review: The Myostatin gene: an overview of mechanisms of action and its 2 relevance to livestock animals 3 D. Aiello 1, K. Patel 2 and E. Lasagna 1 4 5 1 Dipartimento di Scienze Agrarie, Alimentari e Ambientali, Università degli Studi di 6 Perugia, Borgo XX Giugno 74, 06121, Perugia, Italy 7 2 School of Biological Sciences, University of Reading, Berkshire, RG6 6UB, United 8 Kingdom 9 10 Corresponding author: Emiliano Lasagna. Fax: +39 075 5857122. Tel: +39 075 11 5857102. E-mail address: [email protected] 12 13 1 14 15 Summary 16 Myostatin, also known as Growth Differentiation Factor 8, a member of the 17 Transforming Growth Factor-beta (TGF-β) super-family is a negative regulator of 18 muscle development. -

Pharmacovigilance of Veterinary Medicinal Products
a. Reporter Categories Page 1 of 112 Reporter Categories GL42 A.3.1.1. and A.3.2.1. VICH Code VICH TERM VICH DEFINITION C82470 VETERINARIAN Individuals qualified to practice veterinary medicine. C82468 ANIMAL OWNER The owner of the animal or an agent acting on the behalf of the owner. C25741 PHYSICIAN Individuals qualified to practice medicine. C16960 PATIENT The individual(s) (animal or human) exposed to the VMP OTHER HEALTH CARE Health care professional other than specified in list. C53289 PROFESSIONAL C17998 UNKNOWN Not known, not observed, not recorded, or refused b. RA Identifier Codes Page 2 of 112 RA (Regulatory Authorities) Identifier Codes VICH RA Mail/Zip ISO 3166, 3 Character RA Name Street Address City State/County Country Identifier Code Code Country Code 7500 Standish United Food and Drug Administration, Center for USFDACVM Place (HFV-199), Rockville Maryland 20855 States of USA Veterinary Medicine Room 403 America United States Department of Agriculture Animal 1920 Dayton United APHISCVB and Plant Health Inspection Service, Center for Avenue P.O. Box Ames Iowa 50010 States of USA Veterinary Biologic 844 America AGES PharmMed Austrian Medicines and AUTAGESA Schnirchgasse 9 Vienna NA 1030 Austria AUT Medical Devices Agency Eurostation II Federal Agency For Medicines And Health BELFAMHP Victor Hortaplein, Brussel NA 1060 Belgium BEL Products 40 bus 10 7, Shose Bankya BGRIVETP Institute For Control Of Vet Med Prods Sofia NA 1331 Bulgaria BGR Str. CYPVETSE Veterinary Services 1411 Nicosia Nicosia NA 1411 Cyprus CYP Czech CZEUSKVB -

Country Report Italy 2011
European Regional Focal Point for Animal Genetic Resources (ERFP) 21 st April 2011 ERFP Country report 2010 – 2011 COUNTRY: ITALY reported by: Giovanni Bittante Strategic Priority Area 1: Characterization, Inventory and Monitoring of Trends and Associated Risks The inventory of Italian animal genetic resources and the monitoring of trends and associated risks has been undertaken and summarized il the paper "Italian animal genetic resources in the Domestic Animal Diversity Information Systm of FAO" (Giovanni Bittante, 2011, Italian Journal of Animal Science,vol 10:e29, Annex 2). Several research activities on this topic have been carried out by Universities and Research Institutions. Among them see the important activity of ConSDABI (Annex 3). Strategic Priority Area 2: Sustainable Use and Development Beyond the systematic control of animals of Italian populations (herd books, pedigree registries, milk recording, type evaluation, etc.) made by the different associations of breeders, as outlined in Annex 2, several projects dealing with sustainable use, products valorisation and development has been carried out by national and local governments, agencies, breeders associations and consortia. Strategic Priority Area 3: Conservation (please give details for the most relevant institutions for national genebanks / cryopreservation in the table in Annex 1) In situ conservation activities are going on for almost all the Italian AnGR, while the project of a national virtual cryo-bank is not yet fully established, despite the intense work of the -

Horse Breeds - Volume 3
Horse Breeds - Volume 3 A Wikipedia Compilation by Michael A. Linton Contents Articles Latvian horse 1 Lipizzan 3 Lithuanian Heavy Draught 11 Lokai 12 Losino horse 13 Lusitano 14 Malopolski 19 Mallorquín 21 Mangalarga 23 Mangalarga Marchador 24 Maremmano 28 Marismeño 30 Marwari horse 31 Mecklenburger 35 Međimurje horse 39 Menorquín horse 41 Mérens horse 43 Messara horse 51 Miniature horse 52 Misaki horse 57 Missouri Fox Trotter 59 Monchino 62 Mongolian horse 63 Monterufolino 65 Morab 66 Morgan horse 70 Moyle horse 76 Murakoz horse 77 Murgese 78 Mustang horse 80 Namib Desert Horse 86 Nangchen horse 91 National Show Horse 92 Nez Perce Horse 94 Nivernais horse 96 Nokota horse 97 Nonius horse 101 Nordlandshest/Lyngshest 104 Noriker horse 106 Norman Cob 109 Coldblood trotter 114 North Swedish Horse 116 Novokirghiz 118 Oberlander horse 119 Oldenburg horse 120 Orlov Trotter 125 Ostfriesen and Alt-Oldenburger 129 Pampa horse 134 Paso Fino 135 Pentro horse 140 Percheron 141 Persano horse 148 Peruvian Paso 149 Pintabian 154 Pleven horse 156 Poitevin horse 157 Posavac horse 164 Pryor Mountain Mustang 166 Przewalski's horse 175 Purosangue Orientale 183 Qatgani 185 Quarab 186 Racking horse 188 Retuerta horse 189 Rhenish-German Cold-Blood 190 Rhinelander horse 191 Riwoche horse 192 Rocky Mountain Horse 195 Romanian Sporthorse 197 Russian Don 199 Russian Heavy Draft 201 Russian Trotter 203 References Article Sources and Contributors 204 Image Sources, Licenses and Contributors 208 Article Licenses License 212 Latvian horse 1 Latvian horse Latvian Alternative names Latvian Harness Horse Latvian Carriage Latvian Coach Latvian Draft Latvian Riding Horse Country of origin Latvia Horse (Equus ferus caballus) The Latvian horse comes from Latvia and is split into three types: the common harness horse, a lighter riding horse and a heavier draft type. -

Report on the State of Animal Genetic Resources in Italy
Report on the State of Animal Genetic Resources in Italy English (Compact version) July 2005 Report on the State of Animal Genetic Resouces in Italy INTRODUCTION The authorization for the preparation of this Report on the State of Animal Genetic Resources in Italy has been extended in March 2001 following a communication from the Director General of the Food and Agriculture Organization of the United Nations – FAO. The Ministry of Agricultural and Forestry Policies (MiPAF) has assigned the task of preparing this First Report to ConSDABI (Consorzio per la Sperimentazione, Divulgazione e Applicazione di Biotecniche Innovative) under the direction of Prof. Donato Matassino. This Report, in synthesis, describes the current situation of animal genetic resources for food and agriculture and identifies the needs to implement their monitoring, conservation and utilization, their potential contribution to agriculture, nutrition and to sustainable and multifunctional rural development; it also analyzes the current capacity of the country for the management of these resources and describes the priorities that may be useful for international cooperation. 1 Report on the State of Animal Genetic Resouces in Italy CHAPTER 1 THE STATE OF BIODIVERSITY IN THE ANIMAL SECTOR IN ITALY 1.1. Italian agriculture, animal production systems and relative biodiversity 1.1.1. The country Italy has borders with France (west), Switzerland and Austria (north), Slovenia (east), and in its interior is found the Vatican State and San Marino. The country has 20 regions with 103 provinces and 8.101 municipalities (comuni). In 2003 it had 57, 3 million inhabitants on an area of 301.336 km2, and a mean density of 190 inhabitants / km2.