Tese, E Em Particular
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

View Details
INDEX CHAPTER NUMBER CHAPTER NAME PAGE Extraction of Fungal Chitosan and its Chapter-1 1-17 Advanced Application Isolation and Separation of Phenolics Chapter-2 using HPLC Tool: A Consolidate Survey 18-48 from the Plant System Advances in Microbial Genomics in Chapter-3 49-80 the Post-Genomics Era Advances in Biotechnology in the Chapter-4 81-94 Post Genomics era Plant Growth Promotion by Endophytic Chapter-5 Actinobacteria Associated with 95-107 Medicinal Plants Viability of Probiotics in Dairy Products: A Chapter-6 Review Focusing on Yogurt, Ice 108-132 Cream, and Cheese Published in: Dec 2018 Online Edition available at: http://openaccessebooks.com/ Reprints request: [email protected] Copyright: @ Corresponding Author Advances in Biotechnology Chapter 1 Extraction of Fungal Chitosan and its Advanced Application Sahira Nsayef Muslim1; Israa MS AL-Kadmy1*; Alaa Naseer Mohammed Ali1; Ahmed Sahi Dwaish2; Saba Saadoon Khazaal1; Sraa Nsayef Muslim3; Sarah Naji Aziz1 1Branch of Biotechnology, Department of Biology, College of Science, AL-Mustansiryiah University, Baghdad-Iraq 2Branch of Fungi and Plant Science, Department of Biology, College of Science, AL-Mustansiryiah University, Baghdad-Iraq 3Department of Geophysics, College of remote sensing and geophysics, AL-Karkh University for sci- ence, Baghdad-Iraq *Correspondense to: Israa MS AL-Kadmy, Department of Biology, College of Science, AL-Mustansiryiah University, Baghdad-Iraq. Email: [email protected] 1. Definition and Chemical Structure Biopolymer is a term commonly used for polymers which are synthesized by living organisms [1]. Biopolymers originate from natural sources and are biologically renewable, biodegradable and biocompatible. Chitin and chitosan are the biopolymers that have received much research interests due to their numerous potential applications in agriculture, food in- dustry, biomedicine, paper making and textile industry. -
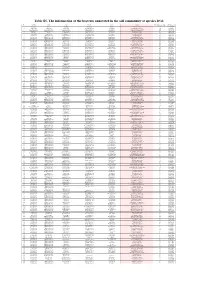
Table S5. the Information of the Bacteria Annotated in the Soil Community at Species Level
Table S5. The information of the bacteria annotated in the soil community at species level No. Phylum Class Order Family Genus Species The number of contigs Abundance(%) 1 Firmicutes Bacilli Bacillales Bacillaceae Bacillus Bacillus cereus 1749 5.145782459 2 Bacteroidetes Cytophagia Cytophagales Hymenobacteraceae Hymenobacter Hymenobacter sedentarius 1538 4.52499338 3 Gemmatimonadetes Gemmatimonadetes Gemmatimonadales Gemmatimonadaceae Gemmatirosa Gemmatirosa kalamazoonesis 1020 3.000970902 4 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas indica 797 2.344876284 5 Firmicutes Bacilli Lactobacillales Streptococcaceae Lactococcus Lactococcus piscium 542 1.594633558 6 Actinobacteria Thermoleophilia Solirubrobacterales Conexibacteraceae Conexibacter Conexibacter woesei 471 1.385742446 7 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas taxi 430 1.265115184 8 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas wittichii 388 1.141545794 9 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas sp. FARSPH 298 0.876754244 10 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sorangium cellulosum 260 0.764953367 11 Proteobacteria Deltaproteobacteria Myxococcales Polyangiaceae Sorangium Sphingomonas sp. Cra20 260 0.764953367 12 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas panacis 252 0.741416341 -

Genome Mining of Biosynthetic and Chemotherapeutic Gene Clusters in Streptomyces Bacteria Kaitlyn C
www.nature.com/scientificreports OPEN Genome mining of biosynthetic and chemotherapeutic gene clusters in Streptomyces bacteria Kaitlyn C. Belknap1,2, Cooper J. Park1,2, Brian M. Barth1 & Cheryl P. Andam 1* Streptomyces bacteria are known for their prolifc production of secondary metabolites, many of which have been widely used in human medicine, agriculture and animal health. To guide the efective prioritization of specifc biosynthetic gene clusters (BGCs) for drug development and targeting the most prolifc producer strains, knowledge about phylogenetic relationships of Streptomyces species, genome- wide diversity and distribution patterns of BGCs is critical. We used genomic and phylogenetic methods to elucidate the diversity of major classes of BGCs in 1,110 publicly available Streptomyces genomes. Genome mining of Streptomyces reveals high diversity of BGCs and variable distribution patterns in the Streptomyces phylogeny, even among very closely related strains. The most common BGCs are non-ribosomal peptide synthetases, type 1 polyketide synthases, terpenes, and lantipeptides. We also found that numerous Streptomyces species harbor BGCs known to encode antitumor compounds. We observed that strains that are considered the same species can vary tremendously in the BGCs they carry, suggesting that strain-level genome sequencing can uncover high levels of BGC diversity and potentially useful derivatives of any one compound. These fndings suggest that a strain-level strategy for exploring secondary metabolites for clinical use provides an alternative or complementary approach to discovering novel pharmaceutical compounds from microbes. Members of the bacterial genus Streptomyces (phylum Actinobacteria) are best known as major bacterial produc- ers of antibiotics and other useful compounds commonly used in human medicine, animal health and agricul- ture1,2. -

Bioinformatic Analysis of Streptomyces to Enable Improved Drug Discovery
University of New Hampshire University of New Hampshire Scholars' Repository Master's Theses and Capstones Student Scholarship Spring 2019 BIOINFORMATIC ANALYSIS OF STREPTOMYCES TO ENABLE IMPROVED DRUG DISCOVERY Kaitlyn Christina Belknap University of New Hampshire, Durham Follow this and additional works at: https://scholars.unh.edu/thesis Recommended Citation Belknap, Kaitlyn Christina, "BIOINFORMATIC ANALYSIS OF STREPTOMYCES TO ENABLE IMPROVED DRUG DISCOVERY" (2019). Master's Theses and Capstones. 1268. https://scholars.unh.edu/thesis/1268 This Thesis is brought to you for free and open access by the Student Scholarship at University of New Hampshire Scholars' Repository. It has been accepted for inclusion in Master's Theses and Capstones by an authorized administrator of University of New Hampshire Scholars' Repository. For more information, please contact [email protected]. BIOINFORMATIC ANALYSIS OF STREPTOMYCES TO ENABLE IMPROVED DRUG DISCOVERY BY KAITLYN C. BELKNAP B.S Medical Microbiology, University of New Hampshire, 2017 THESIS Submitted to the University of New Hampshire in Partial Fulfillment of the Requirements for the Degree of Master of Science in Genetics May, 2019 ii BIOINFORMATIC ANALYSIS OF STREPTOMYCES TO ENABLE IMPROVED DRUG DISCOVERY BY KAITLYN BELKNAP This thesis was examined and approved in partial fulfillment of the requirements for the degree of Master of Science in Genetics by: Thesis Director, Brian Barth, Assistant Professor of Pharmacology Co-Thesis Director, Cheryl Andam, Assistant Professor of Microbial Ecology Krisztina Varga, Assistant Professor of Biochemistry Colin McGill, Associate Professor of Chemistry (University of Alaska Anchorage) On February 8th, 2019 Approval signatures are on file with the University of New Hampshire Graduate School. -

Sphingobium Estronivorans Sp. Nov. and Sphingobium Bisphenolivorans Sp
TAXONOMIC DESCRIPTION Qin et al., Int. J. Syst. Evol. Microbiol. 2020;70:1822–1829 DOI 10.1099/ijsem.0.003978 Sphingobium estronivorans sp. nov. and Sphingobium bisphenolivorans sp. nov., isolated from a wastewater treatment plant Dan Qin1,2, Cong Ma3, Min Lv4 and Chang- Ping Yu1,5,* ABSTRACT Two Gram- stain- negative, aerobic, motile and rod- shaped bacteria, one designated as strain AXBT, capable of degrading estro- gens, and another, YL23T, capable of degrading estrogen and bisphenol A, were isolated from activated sludge in Xiamen City, PR China. The optimum temperature and pH of both strains were 25–35 °C and pH 7.0–8.0. While strain AXBT could tolerate 3 % (w/v) NaCl, YL23T could only grow between 0–1 % (w/v) NaCl. They contained ubiquinone-10 as the major quinone, spermidine as the major polyamine, summed feature 8 (comprising C18:1ω6c and/or C18:1ω7c) as the major fatty acids and diphosphatidyl- glycerol, phosphatidylcholine, phosphatidyldimethylethanolamine, phosphatidylethanolamine, phosphatidylglycerol and sphin- goglycolipid as the major polar lipids. The DNA G+C contents of strains AXBT and YL23T were 63.6 and 63.7 mol%, respectively. Based on the results of 16S rRNA gene sequence analysis, strains AXBT and YL23T belonged to the genus Sphingobium. Strain AXBT was most closely related to Sphingobium chlorophenolicum NBRC 16172T (97.5 %) and Sphingobium chungbukense DJ77T (97.2 %), and strain YL23T was most closely related to S. chlorophenolicum NBRC 16172T (97.4 %) and S. quisquiliarum P25T (97.1 %). Average nucleotide identity values between these two strains and S. chlorophenolicum NBRC 16172T, S. chungbuke- nse DJ77T, Sphingobium chinhatense IP26T, Sphingobium quisquiliarum P25T and Sphingobium japonicum UT26ST were from 80.7 to 85.8 %. -

Downloaded from the NCBI Database on December 2017
G C A T T A C G G C A T genes Article Genomic Analysis of γ-Hexachlorocyclohexane-Degrading Sphingopyxis lindanitolerans WS5A3p Strain in the Context of the Pangenome of Sphingopyxis Michal A. Kaminski 1 , Adam Sobczak 1,2, Andrzej Dziembowski 1,2 and Leszek Lipinski 1,2,* 1 Institute of Biochemistry and Biophysics, Polish Academy of Sciences, Pawinskiego 5a, 02-106 Warsaw, Poland 2 Institute of Genetics and Biotechnology, Faculty of Biology, University of Warsaw, Pawinskiego 5a, 02-106 Warsaw, Poland * Correspondence: [email protected] Received: 29 July 2019; Accepted: 2 September 2019; Published: 6 September 2019 Abstract: Sphingopyxis inhabit diverse environmental niches, including marine, freshwater, oceans, soil and anthropogenic sites. The genus includes 20 phylogenetically distinct, valid species, but only a few with a sequenced genome. In this work, we analyzed the nearly complete genome of the newly described species, Sphingopyxis lindanitolerans, and compared it to the other available Sphingopyxis genomes. The genome included 4.3 Mbp in total and consists of a circular chromosome, and two putative plasmids. Among the identified set of lin genes responsible for γ-hexachlorocyclohexane pesticide degradation, we discovered a gene coding for a new isoform of the LinA protein. The significant potential of this species in the remediation of contaminated soil is also correlated with the fact that its genome encodes a higher number of enzymes potentially involved in aromatic compound degradation than for most other Sphingopyxis strains. Additional analysis of 44 Sphingopyxis representatives provides insights into the pangenome of Sphingopyxis and revealed a core of 734 protein clusters and between four and 1667 unique proteins per genome. -

Sphingobium Cupriresistens Sp. Nov., a Copper- Resistant Bacterium Isolated from Copper Mine Soil, and Emended Description of the Genus Sphingobium
%paper no. ije040865 charlesworth ref: ije040865& New Taxa - Proteobacteria International Journal of Systematic and Evolutionary Microbiology (2013), 63, 000–000 DOI 10.1099/ijs.0.040865-0 Sphingobium cupriresistens sp. nov., a copper- resistant bacterium isolated from copper mine soil, and emended description of the genus Sphingobium Liqiong Li,3 Hongliang Liu,3 Zunji Shi and Gejiao Wang Correspondence State Key Laboratory of Agricultural Microbiology, College of Life Science and Technology, Gejiao Wang Huazhong Agricultural University, Wuhan, 430070, PR China [email protected] or [email protected] A Gram-negative, aerobic, copper-resistant bacterium, designated strain CU4T, was isolated from copper mine soil in Daye, China. Phylogenetic analysis based on 16S rRNA gene sequences showed highest similarity to Sphingobium rhizovicinum CC-FH12-1T (98.4 %), followed by Sphingobium francense Sp+T (97.2 %), Sphingobium japonicum UT26T (97.1 %), Sphingobium abikonense NBRC 16140T (97.0 %), Sphingobium xenophagum DSM 6383T (96.9 %) and Sphingobium yanoikuyae DSM 7462T (95.5 %). The major fatty acids (.5 %) were summed feature 7 (C18 : 1v7c,C18 : 1v9t and/or C18 : 1v12t), summed feature 4 (C16 : 1v7c and/or iso- C15 : 0 2-OH), C16 : 0 and C14 : 0 2-OH, and the predominant quinone was ubiquinone Q-10. Spermidine was the major polyamine component. The major polar lipids were diphosphatidyl- glycerol, phosphatidylethanolamine, phosphatidylglycerol, sphingoglycolipid, phosphatidyldi- methylethanolamine and phosphatidylcholine. The genomic DNA G+C content of strain CU4T was 64.9 mol%. Comparison of DNA–DNA hybridization, phenotypic and chemotaxonomic characteristics between strain CU4T and phylogenetically related strains revealed that the new isolate represents a novel species of the genus Sphingobium, for which the name Sphingobium cupriresistens sp. -

Genome Organization of Sphingobium Indicum B90A: an Archetypal Hexachlorocyclohexane (HCH) Degrading Genotype
Genome Organization of Sphingobium indicum B90A: An Archetypal Hexachlorocyclohexane (HCH) Degrading Genotype Helianthous Vermaa, Abhay Bajaja, Roshan Kumara, Jasvinder Kaura, Shailly Ananda, Namita Nayyara, Akshita Puria,c, Yogendra Singhb, J. P. Khuranac and Rup Lala* *Author for correspondence: Prof. Rup Lal, e-mail: [email protected], aRoom No. 115, Molecular Biology Laboratory, Department of Zoology, University of Delhi, Delhi 110007, India. bRoom No. 112, Bacterial Pathogenesis Laboratory, Department of Zoology, University of Delhi, Delhi 110007, India. cInterdisciplinary Centre for Plant Genomics & Department of Plant Molecular Biology, University of Delhi South Campus, New Delhi, India. Data Deposition: Sequence data deposited in NCBI database under GenBank Accession numbers: CP013070.1, CP013071.1, CP013072.1 and CP013073.1. © The Author(s) 2017. Published by Oxford University Press on behalf of the Society for Molecular Biology and Evolution. This is an Open Access article distributed under the terms of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/4.0/), which permits non-commercial re-use, distribution, and reproduction in any medium, provided the original work is properly cited. For commercial re-use, please contact [email protected] Abstract. Among sphingomonads, Sphingobium indicum B90A is a widely investigated for its ability to degrade a manmade pesticide, γ-hexachlorocyclohexane (γ-HCH) and its isomers (α-, β-, δ- and ε-HCH). In this study, complete genome of strain B90A was constructed using Single Molecule Real Time Sequencing (SMRT) and Illumina platform. The complete genome revealed that strain B90A harbors four replicons: one chromosome (3,654,322 bp) and three plasmids designated as pSRL1 (139,218 bp), pSRL2 (108,430 bp) and pSRL3 (43,761 bp). -

Genomic Insights Into the Evolution of Hybrid Isoprenoid Biosynthetic Gene Clusters in the MAR4 Marine Streptomycete Clade
UC San Diego UC San Diego Previously Published Works Title Genomic insights into the evolution of hybrid isoprenoid biosynthetic gene clusters in the MAR4 marine streptomycete clade. Permalink https://escholarship.org/uc/item/9944f7t4 Journal BMC genomics, 16(1) ISSN 1471-2164 Authors Gallagher, Kelley A Jensen, Paul R Publication Date 2015-11-17 DOI 10.1186/s12864-015-2110-3 Peer reviewed eScholarship.org Powered by the California Digital Library University of California Gallagher and Jensen BMC Genomics (2015) 16:960 DOI 10.1186/s12864-015-2110-3 RESEARCH ARTICLE Open Access Genomic insights into the evolution of hybrid isoprenoid biosynthetic gene clusters in the MAR4 marine streptomycete clade Kelley A. Gallagher and Paul R. Jensen* Abstract Background: Considerable advances have been made in our understanding of the molecular genetics of secondary metabolite biosynthesis. Coupled with increased access to genome sequence data, new insight can be gained into the diversity and distributions of secondary metabolite biosynthetic gene clusters and the evolutionary processes that generate them. Here we examine the distribution of gene clusters predicted to encode the biosynthesis of a structurally diverse class of molecules called hybrid isoprenoids (HIs) in the genus Streptomyces. These compounds are derived from a mixed biosynthetic origin that is characterized by the incorporation of a terpene moiety onto a variety of chemical scaffolds and include many potent antibiotic and cytotoxic agents. Results: One hundred and twenty Streptomyces genomes were searched for HI biosynthetic gene clusters using ABBA prenyltransferases (PTases) as queries. These enzymes are responsible for a key step in HI biosynthesis. The strains included 12 that belong to the ‘MAR4’ clade, a largely marine-derived lineage linked to the production of diverse HI secondary metabolites. -

New Natural Products Identified by Combined Genomics-Metabolomics Profiling of Marine Streptomyces Sp. MP131-18
www.nature.com/scientificreports OPEN New natural products identified by combined genomics-metabolomics profiling of marineStreptomyces sp. Received: 12 June 2016 Accepted: 10 January 2017 MP131-18 Published: 10 February 2017 Constanze Paulus1, Yuriy Rebets1, Bogdan Tokovenko1, Suvd Nadmid1, Larisa P. Terekhova2, Maksym Myronovskyi1, Sergey B. Zotchev3,4, Christian Rückert5,†, Simone Braig6, Stefan Zahler6, Jörn Kalinowski5 & Andriy Luzhetskyy1,7 Marine actinobacteria are drawing more and more attention as a promising source of new natural products. Here we report isolation, genome sequencing and metabolic profiling of new strain Streptomyces sp. MP131-18 isolated from marine sediment sample collected in the Trondheim Fjord, Norway. The 16S rRNA and multilocus phylogenetic analysis showed that MP131-18 belongs to the genus Streptomyces. The genome of MP131-18 isolate was sequenced, and 36 gene clusters involved in the biosynthesis of 18 different types of secondary metabolites were predicted using antiSMASH analysis. The combined genomics-metabolics profiling of the strain led to the identification of several new biologically active compounds. As a result, the family of bisindole pyrroles spiroindimicins was extended with two new members, spiroindimicins E and F. Furthermore, prediction of the biosynthetic pathway for unusual α-pyrone lagunapyrone isolated from MP131-18 resulted in foresight and identification of two new compounds of this family – lagunapyrones D and E. The diversity of identified and predicted compounds from Streptomyces sp. MP131-18 demonstrates that marine-derived actinomycetes are not only a promising source of new natural products, but also represent a valuable pool of genes for combinatorial biosynthesis of secondary metabolites. The discovery of new antibiotics remains one of the most important tasks of modern biotechnology due to the rapid emergence of antibiotic resistance among pathogenic bacteria1. -

Nomenclature of Taxa of the Order Actinomycetales (Schizomycetes) Erwin Francis Lessel Iowa State University
Iowa State University Capstones, Theses and Retrospective Theses and Dissertations Dissertations 1961 Nomenclature of taxa of the order Actinomycetales (Schizomycetes) Erwin Francis Lessel Iowa State University Follow this and additional works at: https://lib.dr.iastate.edu/rtd Part of the Microbiology Commons Recommended Citation Lessel, Erwin Francis, "Nomenclature of taxa of the order Actinomycetales (Schizomycetes) " (1961). Retrospective Theses and Dissertations. 2440. https://lib.dr.iastate.edu/rtd/2440 This Dissertation is brought to you for free and open access by the Iowa State University Capstones, Theses and Dissertations at Iowa State University Digital Repository. It has been accepted for inclusion in Retrospective Theses and Dissertations by an authorized administrator of Iowa State University Digital Repository. For more information, please contact [email protected]. This dissertation has been (J 1-3042 microfilmed exactly as received LESSEL, Jr., iJrxvin Francis, 1U3U- NOMENCLATURE OF TAXA OF THE ORUEIR AC TINO M YC E TA LE S (SCIIIZO M YC ETES). Iowa State University of Science and Technology Ph.D., 1001 Bacteriology University Microfilms, Inc., Ann Arbor, Mic hi g NOMENCLATURE OF TAXA OF THE ORDKR ACTINOMYCETALES (SCHIZOMYCETES) oy Erwin Francis Lessel, Jr. A Dissertation Submitted to the Graduate Faculty in Partial Fulfillment of the Requirements for the Degree of DOCTOR OF PHILOSOPHY Ma-'or Subject: Bacteriology Approved: Signature was redacted for privacy. In Charge of Major Work Signature was redacted for privacy. -

Sphingobium Bisphenolivorans Sp
View metadata, citation and similar papers at core.ac.uk brought to you by CORE provided by Institutional Repository of Yantai Institute of Coastal Zone Research, CAS TAXONOMIC DESCRIPTION Qin et al., Int. J. Syst. Evol. Microbiol. 2020;70:1822–1829 DOI 10.1099/ijsem.0.003978 Sphingobium estronivorans sp. nov. and Sphingobium bisphenolivorans sp. nov., isolated from a wastewater treatment plant Dan Qin1,2, Cong Ma3, Min Lv4 and Chang- Ping Yu1,5,* ABSTRACT Two Gram- stain- negative, aerobic, motile and rod- shaped bacteria, one designated as strain AXBT, capable of degrading estro- gens, and another, YL23T, capable of degrading estrogen and bisphenol A, were isolated from activated sludge in Xiamen City, PR China. The optimum temperature and pH of both strains were 25–35 °C and pH 7.0–8.0. While strain AXBT could tolerate 3 % (w/v) NaCl, YL23T could only grow between 0–1 % (w/v) NaCl. They contained ubiquinone-10 as the major quinone, spermidine as the major polyamine, summed feature 8 (comprising C18:1ω6c and/or C18:1ω7c) as the major fatty acids and diphosphatidyl- glycerol, phosphatidylcholine, phosphatidyldimethylethanolamine, phosphatidylethanolamine, phosphatidylglycerol and sphin- goglycolipid as the major polar lipids. The DNA G+C contents of strains AXBT and YL23T were 63.6 and 63.7 mol%, respectively. Based on the results of 16S rRNA gene sequence analysis, strains AXBT and YL23T belonged to the genus Sphingobium. Strain AXBT was most closely related to Sphingobium chlorophenolicum NBRC 16172T (97.5 %) and Sphingobium chungbukense DJ77T (97.2 %), and strain YL23T was most closely related to S.