Kristinkesslerdissertation.Pdf
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Systematic Detection of Divergent Brain Proteins in Human Evolution and Their Roles in Cognition
bioRxiv preprint doi: https://doi.org/10.1101/658658; this version posted June 3, 2019. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY 4.0 International license. Systematic detection of divergent brain proteins in human evolution and their roles in cognition Guillaume Dumas1,*, Simon Malesys1 and Thomas Bourgeron1 Affiliations: 1 Human Genetics and Cognitive Functions, Institut Pasteur, UMR3571 CNRS, Université de Paris, Paris, (75015) France * Corresponding author: [email protected] 1 bioRxiv preprint doi: https://doi.org/10.1101/658658; this version posted June 3, 2019. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY 4.0 International license. Abstract The human brain differs from that of other primates, but the underlying genetic mechanisms remain unclear. Here we measured the evolutionary pressures acting on all human protein- coding genes (N=17,808) based on their divergence from early hominins such as Neanderthal, and non-human primates. We confirm that genes encoding brain-related proteins are among the most conserved of the human proteome. Conversely, several of the most divergent proteins in humans compared to other primates are associated with brain-associated diseases such as micro/macrocephaly, dyslexia, and autism. We identified specific eXpression profiles of a set of divergent genes in ciliated cells of the cerebellum, that might have contributed to the emergence of fine motor skills and social cognition in humans. -

Integrating Single-Step GWAS and Bipartite Networks Reconstruction Provides Novel Insights Into Yearling Weight and Carcass Traits in Hanwoo Beef Cattle
animals Article Integrating Single-Step GWAS and Bipartite Networks Reconstruction Provides Novel Insights into Yearling Weight and Carcass Traits in Hanwoo Beef Cattle Masoumeh Naserkheil 1 , Abolfazl Bahrami 1 , Deukhwan Lee 2,* and Hossein Mehrban 3 1 Department of Animal Science, University College of Agriculture and Natural Resources, University of Tehran, Karaj 77871-31587, Iran; [email protected] (M.N.); [email protected] (A.B.) 2 Department of Animal Life and Environment Sciences, Hankyong National University, Jungang-ro 327, Anseong-si, Gyeonggi-do 17579, Korea 3 Department of Animal Science, Shahrekord University, Shahrekord 88186-34141, Iran; [email protected] * Correspondence: [email protected]; Tel.: +82-31-670-5091 Received: 25 August 2020; Accepted: 6 October 2020; Published: 9 October 2020 Simple Summary: Hanwoo is an indigenous cattle breed in Korea and popular for meat production owing to its rapid growth and high-quality meat. Its yearling weight and carcass traits (backfat thickness, carcass weight, eye muscle area, and marbling score) are economically important for the selection of young and proven bulls. In recent decades, the advent of high throughput genotyping technologies has made it possible to perform genome-wide association studies (GWAS) for the detection of genomic regions associated with traits of economic interest in different species. In this study, we conducted a weighted single-step genome-wide association study which combines all genotypes, phenotypes and pedigree data in one step (ssGBLUP). It allows for the use of all SNPs simultaneously along with all phenotypes from genotyped and ungenotyped animals. Our results revealed 33 relevant genomic regions related to the traits of interest. -

Molecular Characterization of Acute Myeloid Leukemia by Next Generation Sequencing: Identification of Novel Biomarkers and Targets of Personalized Therapies
Alma Mater Studiorum – Università di Bologna Dipartimento di Medicina Specialistica, Diagnostica e Sperimentale Dottorato di Ricerca in Oncologia, Ematologia e Patologia XXX Ciclo Settore Scientifico Disciplinare: MED/15 Settore Concorsuale:06/D3 Molecular characterization of acute myeloid leukemia by Next Generation Sequencing: identification of novel biomarkers and targets of personalized therapies Presentata da: Antonella Padella Coordinatore Prof. Pier-Luigi Lollini Supervisore: Prof. Giovanni Martinelli Esame finale anno 2018 Abstract Acute myeloid leukemia (AML) is a hematopoietic neoplasm that affects myeloid progenitor cells and it is one of the malignancies best studied by next generation sequencing (NGS), showing a highly heterogeneous genetic background. The aim of the study was to characterize the molecular landscape of 2 subgroups of AML patients carrying either chromosomal number alterations (i.e. aneuploidy) or rare fusion genes. We performed whole exome sequencing and we integrated the mutational data with transcriptomic and copy number analysis. We identified the cell cycle, the protein degradation, response to reactive oxygen species, energy metabolism and biosynthetic process as the pathways mostly targeted by alterations in aneuploid AML. Moreover, we identified a 3-gene expression signature including RAD50, PLK1 and CDC20 that characterize this subgroup. Taking advantage of RNA sequencing we aimed at the discovery of novel and rare gene fusions. We detected 9 rare chimeric transcripts, of which partner genes were transcription factors (ZEB2, BCL11B and MAFK) or tumor suppressors (SAV1 and PUF60) rarely translocated across cancer types. Moreover, we detected cryptic events hiding the loss of NF1 and WT1, two recurrently altered genes in AML. Finally, we explored the oncogenic potential of the ZEB2-BCL11B fusion, which revealed no transforming ability in vitro. -

Cellular and Molecular Signatures in the Disease Tissue of Early
Cellular and Molecular Signatures in the Disease Tissue of Early Rheumatoid Arthritis Stratify Clinical Response to csDMARD-Therapy and Predict Radiographic Progression Frances Humby1,* Myles Lewis1,* Nandhini Ramamoorthi2, Jason Hackney3, Michael Barnes1, Michele Bombardieri1, Francesca Setiadi2, Stephen Kelly1, Fabiola Bene1, Maria di Cicco1, Sudeh Riahi1, Vidalba Rocher-Ros1, Nora Ng1, Ilias Lazorou1, Rebecca E. Hands1, Desiree van der Heijde4, Robert Landewé5, Annette van der Helm-van Mil4, Alberto Cauli6, Iain B. McInnes7, Christopher D. Buckley8, Ernest Choy9, Peter Taylor10, Michael J. Townsend2 & Costantino Pitzalis1 1Centre for Experimental Medicine and Rheumatology, William Harvey Research Institute, Barts and The London School of Medicine and Dentistry, Queen Mary University of London, Charterhouse Square, London EC1M 6BQ, UK. Departments of 2Biomarker Discovery OMNI, 3Bioinformatics and Computational Biology, Genentech Research and Early Development, South San Francisco, California 94080 USA 4Department of Rheumatology, Leiden University Medical Center, The Netherlands 5Department of Clinical Immunology & Rheumatology, Amsterdam Rheumatology & Immunology Center, Amsterdam, The Netherlands 6Rheumatology Unit, Department of Medical Sciences, Policlinico of the University of Cagliari, Cagliari, Italy 7Institute of Infection, Immunity and Inflammation, University of Glasgow, Glasgow G12 8TA, UK 8Rheumatology Research Group, Institute of Inflammation and Ageing (IIA), University of Birmingham, Birmingham B15 2WB, UK 9Institute of -

CSE642 Final Version
Eindhoven University of Technology MASTER Dimensionality reduction of gene expression data Arts, S. Award date: 2018 Link to publication Disclaimer This document contains a student thesis (bachelor's or master's), as authored by a student at Eindhoven University of Technology. Student theses are made available in the TU/e repository upon obtaining the required degree. The grade received is not published on the document as presented in the repository. The required complexity or quality of research of student theses may vary by program, and the required minimum study period may vary in duration. General rights Copyright and moral rights for the publications made accessible in the public portal are retained by the authors and/or other copyright owners and it is a condition of accessing publications that users recognise and abide by the legal requirements associated with these rights. • Users may download and print one copy of any publication from the public portal for the purpose of private study or research. • You may not further distribute the material or use it for any profit-making activity or commercial gain Eindhoven University of Technology MASTER THESIS Dimensionality Reduction of Gene Expression Data Author: S. (Sako) Arts Daily Supervisor: dr. V. (Vlado) Menkovski Graduation Committee: dr. V. (Vlado) Menkovski dr. D.C. (Decebal) Mocanu dr. N. (Nikolay) Yakovets May 16, 2018 v1.0 Abstract The focus of this thesis is dimensionality reduction of gene expression data. I propose and test a framework that deploys linear prediction algorithms resulting in a reduced set of selected genes relevant to a specified case. Abstract In cancer research there is a large need to automate parts of the process of diagnosis, this is mainly to reduce cost, make it faster and more accurate. -
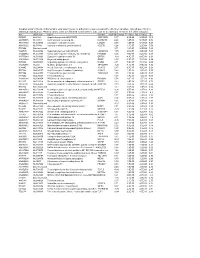
Supplementary File 2A Revised
Supplementary file 2A. Differentially expressed genes in aldosteronomas compared to all other samples, ranked according to statistical significance. Missing values were not allowed in aldosteronomas, but to a maximum of five in the other samples. Acc UGCluster Name Symbol log Fold Change P - Value Adj. P-Value B R99527 Hs.8162 Hypothetical protein MGC39372 MGC39372 2,17 6,3E-09 5,1E-05 10,2 AA398335 Hs.10414 Kelch domain containing 8A KLHDC8A 2,26 1,2E-08 5,1E-05 9,56 AA441933 Hs.519075 Leiomodin 1 (smooth muscle) LMOD1 2,33 1,3E-08 5,1E-05 9,54 AA630120 Hs.78781 Vascular endothelial growth factor B VEGFB 1,24 1,1E-07 2,9E-04 7,59 R07846 Data not found 3,71 1,2E-07 2,9E-04 7,49 W92795 Hs.434386 Hypothetical protein LOC201229 LOC201229 1,55 2,0E-07 4,0E-04 7,03 AA454564 Hs.323396 Family with sequence similarity 54, member B FAM54B 1,25 3,0E-07 5,2E-04 6,65 AA775249 Hs.513633 G protein-coupled receptor 56 GPR56 -1,63 4,3E-07 6,4E-04 6,33 AA012822 Hs.713814 Oxysterol bining protein OSBP 1,35 5,3E-07 7,1E-04 6,14 R45592 Hs.655271 Regulating synaptic membrane exocytosis 2 RIMS2 2,51 5,9E-07 7,1E-04 6,04 AA282936 Hs.240 M-phase phosphoprotein 1 MPHOSPH -1,40 8,1E-07 8,9E-04 5,74 N34945 Hs.234898 Acetyl-Coenzyme A carboxylase beta ACACB 0,87 9,7E-07 9,8E-04 5,58 R07322 Hs.464137 Acyl-Coenzyme A oxidase 1, palmitoyl ACOX1 0,82 1,3E-06 1,2E-03 5,35 R77144 Hs.488835 Transmembrane protein 120A TMEM120A 1,55 1,7E-06 1,4E-03 5,07 H68542 Hs.420009 Transcribed locus 1,07 1,7E-06 1,4E-03 5,06 AA410184 Hs.696454 PBX/knotted 1 homeobox 2 PKNOX2 1,78 2,0E-06 -

Supplementary Table S4. FGA Co-Expressed Gene List in LUAD
Supplementary Table S4. FGA co-expressed gene list in LUAD tumors Symbol R Locus Description FGG 0.919 4q28 fibrinogen gamma chain FGL1 0.635 8p22 fibrinogen-like 1 SLC7A2 0.536 8p22 solute carrier family 7 (cationic amino acid transporter, y+ system), member 2 DUSP4 0.521 8p12-p11 dual specificity phosphatase 4 HAL 0.51 12q22-q24.1histidine ammonia-lyase PDE4D 0.499 5q12 phosphodiesterase 4D, cAMP-specific FURIN 0.497 15q26.1 furin (paired basic amino acid cleaving enzyme) CPS1 0.49 2q35 carbamoyl-phosphate synthase 1, mitochondrial TESC 0.478 12q24.22 tescalcin INHA 0.465 2q35 inhibin, alpha S100P 0.461 4p16 S100 calcium binding protein P VPS37A 0.447 8p22 vacuolar protein sorting 37 homolog A (S. cerevisiae) SLC16A14 0.447 2q36.3 solute carrier family 16, member 14 PPARGC1A 0.443 4p15.1 peroxisome proliferator-activated receptor gamma, coactivator 1 alpha SIK1 0.435 21q22.3 salt-inducible kinase 1 IRS2 0.434 13q34 insulin receptor substrate 2 RND1 0.433 12q12 Rho family GTPase 1 HGD 0.433 3q13.33 homogentisate 1,2-dioxygenase PTP4A1 0.432 6q12 protein tyrosine phosphatase type IVA, member 1 C8orf4 0.428 8p11.2 chromosome 8 open reading frame 4 DDC 0.427 7p12.2 dopa decarboxylase (aromatic L-amino acid decarboxylase) TACC2 0.427 10q26 transforming, acidic coiled-coil containing protein 2 MUC13 0.422 3q21.2 mucin 13, cell surface associated C5 0.412 9q33-q34 complement component 5 NR4A2 0.412 2q22-q23 nuclear receptor subfamily 4, group A, member 2 EYS 0.411 6q12 eyes shut homolog (Drosophila) GPX2 0.406 14q24.1 glutathione peroxidase -

Supplementary Note
1 SUPPLEMENTARY NOTE 2 Table of Contents 3 1 Studies contributing to systolic (SBP) and diastolic blood pressure (DBP) analyses ............ 4 4 2 Consortia and studies providing association results for cardiovascular outcomes ............. 4 5 2.1 CHARGE - Heart Failure Working Group .................................................................................. 4 6 2.2 EchoGen (LM mass and LV weight) .......................................................................................... 4 7 2.3 NEURO-CHARGE (stroke) ........................................................................................................ 4 8 2.4 MetaStroke (stroke) ................................................................................................................ 5 9 2.5 CARDIoGRAMplusC4D (CAD) ................................................................................................... 5 10 2.6 CHARGE CKDgen (CKD, eGFR, microalbuminuria, UACR) ......................................................... 5 11 2.7 KidneyGen (creatinine) ........................................................................................................... 6 12 2.8 CHARGE (cIMT) ....................................................................................................................... 6 13 2.9 CHARGE (mild retinopathy, central retinal artery caliber) ....................................................... 6 14 2.10 SEED (mild retinopathy, central retinal artery caliber) .......................................................... 6 15 -

Scalable Phylogenetic Profiling Using Minhash Uncovers Likely Eukaryotic Sexual Reproduction Genes
bioRxiv preprint doi: https://doi.org/10.1101/852491; this version posted November 22, 2019. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY 4.0 International license. 1 Scalable Phylogenetic Profiling using MinHash Uncovers Likely Eukaryotic Sexual Reproduction Genes 1,2,3,* 1,2,3 4,5 1,2,3,6,7,* David Moi , Laurent Kilchoer , Pablo S. Aguilar and Christophe Dessimoz 1 2 Department of Computational Biology, University of Lausanne, Switzerland; Center for Integrative Genomics, 3 4 University of Lausanne, Switzerland; SIB Swiss Institute of Bioinformatics, Lausanne, Switzerland; Instituto de 5 Investigaciones Biotecnologicas (IIBIO), Universidad Nacional de San Martín Buenos Aires, Argentina; Instituto de 6 Fisiología, Biología Molecular y Neurociencias (IFIBYNE-CONICET), Department of Genetics, Evolution, and 7 Environment, University College London, UK; Department of Computer Science, University College London, UK. *Corresponding authors: [email protected] and [email protected] Abstract Phylogenetic profiling is a computational method to predict genes involved in the same biological process by identifying protein families which tend to be jointly lost or retained across the tree of life. Phylogenetic profiling has customarily been more widely used with prokaryotes than eukaryotes, because the method is thought to require many diverse genomes. There are now many eukaryotic genomes available, but these are considerably larger, and typical phylogenetic profiling methods require quadratic time or worse in the number of genes. We introduce a fast, scalable phylogenetic profiling approach entitled HogProf, which leverages hierarchical orthologous groups for the construction of large profiles and locality-sensitive hashing for efficient retrieval of similar profiles. -

Genome-Wide Transcriptome Analysis of Laminar Tissue During the Early Stages of Experimentally Induced Equine Laminitis
GENOME-WIDE TRANSCRIPTOME ANALYSIS OF LAMINAR TISSUE DURING THE EARLY STAGES OF EXPERIMENTALLY INDUCED EQUINE LAMINITIS A Dissertation by JIXIN WANG Submitted to the Office of Graduate Studies of Texas A&M University in partial fulfillment of the requirements for the degree of DOCTOR OF PHILOSOPHY December 2010 Major Subject: Biomedical Sciences GENOME-WIDE TRANSCRIPTOME ANALYSIS OF LAMINAR TISSUE DURING THE EARLY STAGES OF EXPERIMENTALLY INDUCED EQUINE LAMINITIS A Dissertation by JIXIN WANG Submitted to the Office of Graduate Studies of Texas A&M University in partial fulfillment of the requirements for the degree of DOCTOR OF PHILOSOPHY Approved by: Chair of Committee, Bhanu P. Chowdhary Committee Members, Terje Raudsepp Paul B. Samollow Loren C. Skow Penny K. Riggs Head of Department, Evelyn Tiffany-Castiglioni December 2010 Major Subject: Biomedical Sciences iii ABSTRACT Genome-wide Transcriptome Analysis of Laminar Tissue During the Early Stages of Experimentally Induced Equine Laminitis. (December 2010) Jixin Wang, B.S., Tarim University of Agricultural Reclamation; M.S., South China Agricultural University; M.S., Texas A&M University Chair of Advisory Committee: Dr. Bhanu P. Chowdhary Equine laminitis is a debilitating disease that causes extreme sufferring in afflicted horses and often results in a lifetime of chronic pain. The exact sequence of pathophysiological events culminating in laminitis has not yet been characterized, and this is reflected in the lack of any consistently effective therapeutic strategy. For these reasons, we used a newly developed 21,000 element equine-specific whole-genome oligoarray to perform transcriptomic analysis on laminar tissue from horses with experimentally induced models of laminitis: carbohydrate overload (CHO), hyperinsulinaemia (HI), and oligofructose (OF). -

I FOUR JOINTED BOX ONE, a NOVEL PRO-ANGIOGENIC PROTEIN IN
FOUR JOINTED BOX ONE, A NOVEL PRO-ANGIOGENIC PROTEIN IN COLORECTAL CARCINOMA. BY Nicole Theresa Al-Greene Dissertation Submitted to the Faculty of the Graduate School of Vanderbilt University in partial fulfillment of the requirements for the degree of DOCTOR OF PHILOSOPHY In Cell and Developmental Biology. December, 2013 Nashville Tennessee Approved: R. Daniel Beauchamp Susan Wente James Goldenring Albert Reynolds i DEDICATION To my parents, Karen and John, who have helped me in every way possible, every single day. ii ACKNOWLEDGMENTS. Funding for this work was supported by grants DK052334, CA069457, The GI Cancer SPORE, GM088822, the VICC, the Clinical and Translational Science Award (NCRR/NIH UL1RR024975), the DDRC (P30DK058404), and the Cooperative Human Tissue Network (UO1CA094664) and U01CA094664. I was lucky enough to be allowed to perform research as an undergraduate in the lab of Ken Belanger. I will be forever grateful for that opportunity that sparked my love of research. Equally important was my time as a technician in Len Zon’s lab where I confirmed the fact that I needed to go graduate school and earn my degree. During my time at Vanderbilt I have been helped by so many individuals, and the collaborative nature of everyone I have met with is truly an amazing aspect of the research community here. I am especially thankful for all the technical help and insightful conversations I have had with Natasha Deane, Anna Means, Claudia Andl, Tanner Freeman, Connie Weaver, Keeli Lewis, Jalal Hamaamen, Jenny Zi, John Neff, Christian Kis, Andries Zjistra, Trennis Palmer, Joseph Roland, and Lynn LaPierre. -

Novel Gene Discovery in Primary Ciliary Dyskinesia
Novel Gene Discovery in Primary Ciliary Dyskinesia Mahmoud Raafat Fassad Genetics and Genomic Medicine Programme Great Ormond Street Institute of Child Health University College London A thesis submitted in conformity with the requirements for the degree of Doctor of Philosophy University College London 1 Declaration I, Mahmoud Raafat Fassad, confirm that the work presented in this thesis is my own. Where information has been derived from other sources, I confirm that this has been indicated in the thesis. 2 Abstract Primary Ciliary Dyskinesia (PCD) is one of the ‘ciliopathies’, genetic disorders affecting either cilia structure or function. PCD is a rare recessive disease caused by defective motile cilia. Affected individuals manifest with neonatal respiratory distress, chronic wet cough, upper respiratory tract problems, progressive lung disease resulting in bronchiectasis, laterality problems including heart defects and adult infertility. Early diagnosis and management are essential for better respiratory disease prognosis. PCD is a highly genetically heterogeneous disorder with causal mutations identified in 36 genes that account for the disease in about 70% of PCD cases, suggesting that additional genes remain to be discovered. Targeted next generation sequencing was used for genetic screening of a cohort of patients with confirmed or suggestive PCD diagnosis. The use of multi-gene panel sequencing yielded a high diagnostic output (> 70%) with mutations identified in known PCD genes. Over half of these mutations were novel alleles, expanding the mutation spectrum in PCD genes. The inclusion of patients from various ethnic backgrounds revealed a striking impact of ethnicity on the composition of disease alleles uncovering a significant genetic stratification of PCD in different populations.