Improved Classification and Identification of Acetic Acid Bacteria Based on Molecular Techniques
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Asaia Bogorensis Gen. Nov., Sp. Nov., an Unusual Acetic Acid Bacterium in the Α-Proteobacteria
International Journal of Systematic and Evolutionary Microbiology (2000) 50, 823–829 Printed in Great Britain Asaia bogorensis gen. nov., sp. nov., an unusual acetic acid bacterium in the α-Proteobacteria Yuzo Yamada,1 Kazushige Katsura,1 Hiroko Kawasaki,2 Yantyati Widyastuti,3 Susono Saono,3 Tatsuji Seki,2 Tai Uchimura1 and Kazuo Komagata1 Author for correspondence: Yuzo Yamada. Tel\Fax: j81 54 635 2316. 1 Laboratory of General and Eight Gram-negative, aerobic, rod-shaped and peritrichously flagellated strains Applied Microbiology, were isolated from flowers of the orchid tree (Bauhinia purpurea) and of Department of Applied Biology and Chemistry, plumbago (Plumbago auriculata), and from fermented glutinous rice, all Faculty of Applied collected in Indonesia. The enrichment culture approach for acetic acid bacteria Bioscience, Tokyo was employed, involving use of sorbitol medium at pH 35. All isolates grew University of Agriculture, Sakuragaoka 1-1-1, well at pH 30 and 30 SC. They did not oxidize ethanol to acetic acid except for Setagaya-ku, one strain that oxidized ethanol weakly, and 035% acetic acid inhibited their Tokoyo 156-8502, Japan growth completely. However, they oxidized acetate and lactate to carbon 2 The International Center dioxide and water. The isolates grew well on mannitol agar and on glutamate for Biotechnology, Osaka agar, and assimilated ammonium sulfate for growth on vitamin-free glucose University, Yamadaoka 2-1, Suita, Osaka 565-0871, medium. The isolates produced acid from D-glucose, D-fructose, L-sorbose, Japan dulcitol and glycerol. The quinone system was Q-10. DNA base composition 3 Research and ranged from 593to610 mol% GMC. -
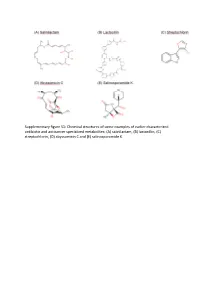
Chemical Structures of Some Examples of Earlier Characterized Antibiotic and Anticancer Specialized
Supplementary figure S1: Chemical structures of some examples of earlier characterized antibiotic and anticancer specialized metabolites: (A) salinilactam, (B) lactocillin, (C) streptochlorin, (D) abyssomicin C and (E) salinosporamide K. Figure S2. Heat map representing hierarchical classification of the SMGCs detected in all the metagenomes in the dataset. Table S1: The sampling locations of each of the sites in the dataset. Sample Sample Bio-project Site depth accession accession Samples Latitude Longitude Site description (m) number in SRA number in SRA AT0050m01B1-4C1 SRS598124 PRJNA193416 Atlantis II water column 50, 200, Water column AT0200m01C1-4D1 SRS598125 21°36'19.0" 38°12'09.0 700 and above the brine N "E (ATII 50, ATII 200, 1500 pool water layers AT0700m01C1-3D1 SRS598128 ATII 700, ATII 1500) AT1500m01B1-3C1 SRS598129 ATBRUCL SRS1029632 PRJNA193416 Atlantis II brine 21°36'19.0" 38°12'09.0 1996– Brine pool water ATBRLCL1-3 SRS1029579 (ATII UCL, ATII INF, N "E 2025 layers ATII LCL) ATBRINP SRS481323 PRJNA219363 ATIID-1a SRS1120041 PRJNA299097 ATIID-1b SRS1120130 ATIID-2 SRS1120133 2168 + Sea sediments Atlantis II - sediments 21°36'19.0" 38°12'09.0 ~3.5 core underlying ATII ATIID-3 SRS1120134 (ATII SDM) N "E length brine pool ATIID-4 SRS1120135 ATIID-5 SRS1120142 ATIID-6 SRS1120143 Discovery Deep brine DDBRINP SRS481325 PRJNA219363 21°17'11.0" 38°17'14.0 2026– Brine pool water N "E 2042 layers (DD INF, DD BR) DDBRINE DD-1 SRS1120158 PRJNA299097 DD-2 SRS1120203 DD-3 SRS1120205 Discovery Deep 2180 + Sea sediments sediments 21°17'11.0" -

The Lichens' Microbiota, Still a Mystery?
fmicb-12-623839 March 24, 2021 Time: 15:25 # 1 REVIEW published: 30 March 2021 doi: 10.3389/fmicb.2021.623839 The Lichens’ Microbiota, Still a Mystery? Maria Grimm1*, Martin Grube2, Ulf Schiefelbein3, Daniela Zühlke1, Jörg Bernhardt1 and Katharina Riedel1 1 Institute of Microbiology, University Greifswald, Greifswald, Germany, 2 Institute of Plant Sciences, Karl-Franzens-University Graz, Graz, Austria, 3 Botanical Garden, University of Rostock, Rostock, Germany Lichens represent self-supporting symbioses, which occur in a wide range of terrestrial habitats and which contribute significantly to mineral cycling and energy flow at a global scale. Lichens usually grow much slower than higher plants. Nevertheless, lichens can contribute substantially to biomass production. This review focuses on the lichen symbiosis in general and especially on the model species Lobaria pulmonaria L. Hoffm., which is a large foliose lichen that occurs worldwide on tree trunks in undisturbed forests with long ecological continuity. In comparison to many other lichens, L. pulmonaria is less tolerant to desiccation and highly sensitive to air pollution. The name- giving mycobiont (belonging to the Ascomycota), provides a protective layer covering a layer of the green-algal photobiont (Dictyochloropsis reticulata) and interspersed cyanobacterial cell clusters (Nostoc spec.). Recently performed metaproteome analyses Edited by: confirm the partition of functions in lichen partnerships. The ample functional diversity Nathalie Connil, Université de Rouen, France of the mycobiont contrasts the predominant function of the photobiont in production Reviewed by: (and secretion) of energy-rich carbohydrates, and the cyanobiont’s contribution by Dirk Benndorf, nitrogen fixation. In addition, high throughput and state-of-the-art metagenomics and Otto von Guericke University community fingerprinting, metatranscriptomics, and MS-based metaproteomics identify Magdeburg, Germany Guilherme Lanzi Sassaki, the bacterial community present on L. -

Developing a Genetic Manipulation System for the Antarctic Archaeon, Halorubrum Lacusprofundi: Investigating Acetamidase Gene Function
www.nature.com/scientificreports OPEN Developing a genetic manipulation system for the Antarctic archaeon, Halorubrum lacusprofundi: Received: 27 May 2016 Accepted: 16 September 2016 investigating acetamidase gene Published: 06 October 2016 function Y. Liao1, T. J. Williams1, J. C. Walsh2,3, M. Ji1, A. Poljak4, P. M. G. Curmi2, I. G. Duggin3 & R. Cavicchioli1 No systems have been reported for genetic manipulation of cold-adapted Archaea. Halorubrum lacusprofundi is an important member of Deep Lake, Antarctica (~10% of the population), and is amendable to laboratory cultivation. Here we report the development of a shuttle-vector and targeted gene-knockout system for this species. To investigate the function of acetamidase/formamidase genes, a class of genes not experimentally studied in Archaea, the acetamidase gene, amd3, was disrupted. The wild-type grew on acetamide as a sole source of carbon and nitrogen, but the mutant did not. Acetamidase/formamidase genes were found to form three distinct clades within a broad distribution of Archaea and Bacteria. Genes were present within lineages characterized by aerobic growth in low nutrient environments (e.g. haloarchaea, Starkeya) but absent from lineages containing anaerobes or facultative anaerobes (e.g. methanogens, Epsilonproteobacteria) or parasites of animals and plants (e.g. Chlamydiae). While acetamide is not a well characterized natural substrate, the build-up of plastic pollutants in the environment provides a potential source of introduced acetamide. In view of the extent and pattern of distribution of acetamidase/formamidase sequences within Archaea and Bacteria, we speculate that acetamide from plastics may promote the selection of amd/fmd genes in an increasing number of environmental microorganisms. -

Supplementary Information for Microbial Electrochemical Systems Outperform Fixed-Bed Biofilters for Cleaning-Up Urban Wastewater
Electronic Supplementary Material (ESI) for Environmental Science: Water Research & Technology. This journal is © The Royal Society of Chemistry 2016 Supplementary information for Microbial Electrochemical Systems outperform fixed-bed biofilters for cleaning-up urban wastewater AUTHORS: Arantxa Aguirre-Sierraa, Tristano Bacchetti De Gregorisb, Antonio Berná, Juan José Salasc, Carlos Aragónc, Abraham Esteve-Núñezab* Fig.1S Total nitrogen (A), ammonia (B) and nitrate (C) influent and effluent average values of the coke and the gravel biofilters. Error bars represent 95% confidence interval. Fig. 2S Influent and effluent COD (A) and BOD5 (B) average values of the hybrid biofilter and the hybrid polarized biofilter. Error bars represent 95% confidence interval. Fig. 3S Redox potential measured in the coke and the gravel biofilters Fig. 4S Rarefaction curves calculated for each sample based on the OTU computations. Fig. 5S Correspondence analysis biplot of classes’ distribution from pyrosequencing analysis. Fig. 6S. Relative abundance of classes of the category ‘other’ at class level. Table 1S Influent pre-treated wastewater and effluents characteristics. Averages ± SD HRT (d) 4.0 3.4 1.7 0.8 0.5 Influent COD (mg L-1) 246 ± 114 330 ± 107 457 ± 92 318 ± 143 393 ± 101 -1 BOD5 (mg L ) 136 ± 86 235 ± 36 268 ± 81 176 ± 127 213 ± 112 TN (mg L-1) 45.0 ± 17.4 60.6 ± 7.5 57.7 ± 3.9 43.7 ± 16.5 54.8 ± 10.1 -1 NH4-N (mg L ) 32.7 ± 18.7 51.6 ± 6.5 49.0 ± 2.3 36.6 ± 15.9 47.0 ± 8.8 -1 NO3-N (mg L ) 2.3 ± 3.6 1.0 ± 1.6 0.8 ± 0.6 1.5 ± 2.0 0.9 ± 0.6 TP (mg -

Deep Divergence and Rapid Evolutionary Rates in Gut-Associated Acetobacteraceae of Ants Bryan P
Brown and Wernegreen BMC Microbiology (2016) 16:140 DOI 10.1186/s12866-016-0721-8 RESEARCH ARTICLE Open Access Deep divergence and rapid evolutionary rates in gut-associated Acetobacteraceae of ants Bryan P. Brown1,2 and Jennifer J. Wernegreen1,2* Abstract Background: Symbiotic associations between gut microbiota and their animal hosts shape the evolutionary trajectories of both partners. The genomic consequences of these relationships are significantly influenced by a variety of factors, including niche localization, interaction potential, and symbiont transmission mode. In eusocial insect hosts, socially transmitted gut microbiota may represent an intermediate point between free living or environmentally acquired bacteria and those with strict host association and maternal transmission. Results: We characterized the bacterial communities associated with an abundant ant species, Camponotus chromaiodes. While many bacteria had sporadic distributions, some taxa were abundant and persistent within and across ant colonies. Specially, two Acetobacteraceae operational taxonomic units (OTUs; referred to as AAB1 and AAB2) were abundant and widespread across host samples. Dissection experiments confirmed that AAB1 and AAB2 occur in C. chromaiodes gut tracts. We explored the distribution and evolution of these Acetobacteraceae OTUs in more depth. We found that Camponotus hosts representing different species and geographical regions possess close relatives of the Acetobacteraceae OTUs detected in C. chromaiodes. Phylogenetic analysis revealed that AAB1 and AAB2 join other ant associates in a monophyletic clade. This clade consists of Acetobacteraceae from three ant tribes, including a third, basal lineage associated with Attine ants. This ant-specific AAB clade exhibits a significant acceleration of substitution rates at the 16S rDNA gene and elevated AT content. -

Acetobacter Sacchari Sp. Nov., for a Plant Growth-Promoting Acetic Acid Bacterium Isolated in Vietnam
Annals of Microbiology (2019) 69:1155–11631163 https://doi.org/10.1007/s13213-019-01497-0 ORIGINAL ARTICLE Acetobacter sacchari sp. nov., for a plant growth-promoting acetic acid bacterium isolated in Vietnam Huong Thi Lan Vu1,2 & Pattaraporn Yukphan3 & Van Thi Thu Bui1 & Piyanat Charoenyingcharoen3 & Sukunphat Malimas4 & Linh Khanh Nguyen1 & Yuki Muramatsu5 & Naoto Tanaka6 & Somboon Tanasupawat7 & Binh Thanh Le2 & Yasuyoshi Nakagawa5 & Yuzo Yamada3,8,9 Received: 21 January 2019 /Accepted: 7 July 2019 /Published online: 18 July 2019 # Università degli studi di Milano 2019 Abstract Purpose Two bacterial strains, designated as isolates VTH-Ai14T and VTH-Ai15, that have plant growth-promoting ability were isolated during the study on acetic acid bacteria diversity in Vietnam. The phylogenetic analysis based on 16S rRNA gene sequences showed that the two isolates were located closely to Acetobacter nitrogenifigens RG1T but formed an independent cluster. Methods The phylogenetic analysis based on 16S rRNA gene and three housekeeping genes’ (dnaK, groEL, and rpoB) sequences were analyzed. The genomic DNA of the two isolates, VTH-Ai14T and VTH-Ai15, Acetobacter nitrogenifigens RG1T, the closest phylogenetic species, and Acetobacter aceti NBRC 14818T were hybridized and calculated the %similarity. Then, phenotypic and chemotaxonomic characteristics were determined for species’ description using the conventional method. Results The 16S rRNA gene and concatenated of the three housekeeping genes phylogenetic analysis suggests that the two isolates were constituted in a species separated from Acetobacter nitrogenifigens, Acetobacter aceti,andAcetobacter sicerae. The two isolates VTH-Ai14T and VTH-Ai15 showed 99.65% and 98.65% similarity of 16S rRNA gene when compared with Acetobacter nitrogenifigens and Acetobacter aceti and they were so different from Acetobacter nitrogenifigens RG1T with 56.99 ± 3.6 and 68.15 ± 1.8% in DNA-DNA hybridization, when isolates VTH-Ai14T and VTH-Ai15 were respectively labeled. -

And Acetobacter Pornorurn Spm Nov., Two New Species Isolated from Industrial Vinegar Fermentations
International Journal of Systematic Bacteriology (1 998),48, 93 5-940 Printed in Great Britain Description of Acetobacter oboediens Spm nov, and Acetobacter pornorurn Spm nov., two new species isolated from industrial vinegar fermentations Stephan J. Sokollek, Christian HerteI and Walter P. Hammes Author for correspondence: Walter P. Hammes. Tel: +49 71 1 459 2305. Fax: +49 71 1 459 4255. e-mail: [email protected] lnstitut fur Two strains of Acetobacter sp., LTH 2460Tand LTH 2458T, have been isolated Lebensmitteltechnologie, from running red wine and cider vinegar fermentations, respectively. Universitat Hohenheim, GarbenstraBe 28, D-70599 Taxonomic characteristics of the isolates were investigated. Comparative Stuttg a rt, Germany analysis of the 165 rRNA sequences revealed > 99% similarity between strain LTH 2460Tand the type strains of the related species Acetobacter europaeus and Acetobacter xylinus and between strain LTH 2458Tand Acetobacter pasteurianus. On the other hand, low levels of DNA relatedness (< 34%) were determined in DNA-DNA similarity studies. This relatedness below the species level was consistent with specific physiological characteristics permitting clear identification of these strains within established species of acetic acid bacteria. Based on these results, the names Acetobacter oboediens sp. nov. and Acetobacterpomorum sp. nov. are proposed for strains LTH 2460Tand LTH 2458T, respectively. The phylogenetic positions of the new species are reflected by a 165 rRNA-based tree. Furthermore, a 165 rRNA-targeted oligonucleotide probe specific for A. oboediens was constructed. Keywords: Acetobacter oboediens sp. nov., Acetobacter pomorum sp. nov., vinegar fermentation, acetic acid bacteria INTRODUCTION isolated from vinegar fermentations are not suitable for preservation (Sievers & Teuber, 1995). -

Influence of Lab Adapted Natural Diet, Genotype, and Microbiota On
INFLUENCE OF LAB ADAPTED NATURAL DIET, GENOTYPE, AND MICROBIOTA ON DROSOPHILA MELANOGASTER LARVAE by ANDREI BOMBIN LAURA K. REED, COMMITTEE CHAIR JULIE B. OLSON JOHN H. YODER STANISLAVA CHTARBANOVA-RUDLOFF CASEY D. MORROW A DISSERTATION Submitted in partial fulfillment of the requirements for the degree of Doctor of Philosophy in the Department of Biological Sciences in the Graduate School of The University of Alabama TUSCALOOSA, ALABAMA 2020 Copyright Andrei Bombin 2020 ALL RIGHTS RESERED ABSTRACT Obesity is an increasing pandemic and is caused by multiple factors including genotype, psychological stress, and gut microbiota. Our project investigated the effects produced by microbiota community, acquired from the environment and horizontal transfer, on traits related to obesity. The study applied a novel approach of raising Drosophila melanogaster, from ten wild-derived genetic lines on naturally fermented peaches, preserving genuine microbial conditions. Larvae raised on the natural and standard lab diets were significantly different in every tested phenotype. Frozen peach food provided nutritional conditions similar to the natural ones and preserved key microbial taxa necessary for survival and development. On the peach diet, the presence of parental microbiota increased the weight and development rate. Larvae raised on each tested diet formed microbial communities distinct from each other. In addition, we evaluated the change in microbial communities and larvae phenotypes due to the high fat and high sugar diet modifications. We observed that presence of symbiotic microbiota often mitigated the effect that harmful dietary modifications produced on larvae and was crucial for Drosophila survival on high sugar peach diets. Although genotype of the host was the most influential factor shaping the microbiota community, several dominant microbial taxa were consistently associated with nutritional modifications across lab and peach diets. -

Isolation and Characterization of Acetobacter Aceti from Rotten Papaya
J. Bangladesh Agril. Univ. 13(2): 299–306, 2015 ISSN 1810-3030 Isolation and characterization of Acetobacter aceti from rotten papaya J. Kowser*, M. G. Aziz and M. B. Uddin Department of Food Technology and Rural Industries, Bangladesh Agricultural University, Mymensingh-2202, Bangladesh, *Email: [email protected] Abstract The present study was concerned with the isolation and characterization of Acetobacter aceti from rotten papaya for vinegar production. The samples were inoculated in sterilized GYC standard media and then incubated at 30°C for 48 hours. Successive subculture was performed to screen out the strains. In Gram’s staining, the morphology of the isolated bacteria exhibited pink, small rod shaped single, pair and chain in arrangement, in the hanging drops technique, all the isolates revealed motile. Biochemical tests were performed by fermentation of five basic sugars by producing both acid and gas bubbles in Durham tube. All of the isolates were Indole, Voges-Proskauer (VP) and Oxidase negative, Methyl Red (MR) and Catalase positive. The growth rate of isolated strain was optimized by weighing dry cell and turbidity at 600 nm at different concentrations of dextrose (1%, 5% and 10%). Ten (10) percent dextrose solution showed rapid growth and higher cell mass than 5% and 1% solution respectively. Acidity of the media gradually increased from 0.102% to 2.18% from day 0 to day 7 and pH of the media decreased from 6.8 to 5.5 during the period. This protocol was successful for enriching Acetobacter aceti, which was essential for vinegar production. Keywords: Rotten Papaya, Acetobacter aceti, GYC culture media, Morphological and Biochemical assessment Introduction Acetic acid bacteria (AAB) is a group of gram-negative, aerobic and motile rods that carry out incomplete oxidation of alcohol and sugars, leading to the accumulation of organic acids as end products. -

Advances in Applied Microbiology, Voume 49 (Advances in Applied
ADVANCES IN Applied Microbiology VOLUME 49 ThisPageIntentionallyLeftBlank ADVANCES IN Applied Microbiology Edited by ALLEN I. LASKIN JOAN W. BENNETT Somerset, New Jersey New Orleans, Louisiana GEOFFREY M. GADD Dundee, United Kingdom VOLUME 49 San Diego New York Boston London Sydney Tokyo Toronto This book is printed on acid-free paper. ∞ Copyright C 2001 by ACADEMIC PRESS All Rights Reserved. No part of this publication may be reproduced or transmitted in any form or by any means, electronic or mechanical, including photocopy, recording, or any information storage and retrieval system, without permission in writing from the Publisher. The appearance of the code at the bottom of the first page of a chapter in this book indicates the Publisher’s consent that copies of the chapter may be made for personal or internal use of specific clients. This consent is given on the condition, however, that the copier pay the stated per copy fee through the Copyright Clearance Center, Inc. (222 Rosewood Drive, Danvers, Massachusetts 01923), for copying beyond that permitted by Sections 107 or 108 of the U.S. Copyright Law. This consent does not extend to other kinds of copying, such as copying for general distribution, for advertising or promotional purposes, for creating new collective works, or for resale. Copy fees for pre-2000 chapters are as shown on the title pages. If no fee code appears on the title page, the copy fee is the same as for current chapters. 0065-2164/01 $35.00 Academic Press A division of Harcourt, Inc. 525 B Street, Suite 1900, San Diego, California 92101-4495, USA http://www.academicpress.com Academic Press Harcourt Place, 32 Jamestown Road, London NW1 7BY, UK http://www.academicpress.com International Standard Serial Number: 0065-2164 International Standard Book Number: 0-12-002649-X PRINTED IN THE UNITED STATES OF AMERICA 010203040506MM987654321 CONTENTS Microbial Transformations of Explosives SUSAN J. -

Acetobacter Fabarum Genes Influencing Drosophila Melanogaster Phenotypes Kylie Makay White Brigham Young University
Brigham Young University BYU ScholarsArchive All Theses and Dissertations 2017-12-01 Acetobacter fabarum Genes Influencing Drosophila melanogaster Phenotypes Kylie MaKay White Brigham Young University Follow this and additional works at: https://scholarsarchive.byu.edu/etd Part of the Microbiology Commons BYU ScholarsArchive Citation White, Kylie MaKay, "Acetobacter fabarum Genes Influencing Drosophila melanogaster Phenotypes" (2017). All Theses and Dissertations. 6613. https://scholarsarchive.byu.edu/etd/6613 This Thesis is brought to you for free and open access by BYU ScholarsArchive. It has been accepted for inclusion in All Theses and Dissertations by an authorized administrator of BYU ScholarsArchive. For more information, please contact [email protected], [email protected]. Acetobacter fabarum Genes Influencing Drosophila melanogaster Phenotypes Kylie Makay White A thesis submitted to the faculty of Brigham Young University in partial fulfillment of the requirements for the degree of Master of Science John M. Chaston, Chair Joel Griffitts Laura Bridgewater Department of Microbiology and Molecular Biology Brigham Young University Copyright © 2017 Kylie Makay White All Rights Reserved ABSTRACT Acetobacter fabarum Genes Influencing Drosophila melanogaster Phenotypes Kylie Makay White Department of Microbiology and Molecular Biology, BYU Master of Science Research in our lab has predicted hundreds of bacterial genes that influence nine different traits in the fruit fly, Drosophila melanogaster. As a practical alternative to creating site-directed mutants for each of the predicted genes, we created an arrayed transposon insertion library using a strain of Acetobacter fabarum DsW_054 isolated from fruit flies. Creation of the Acetobacter fabarum DsW_054 gene knock-out library was done through random transposon insertion, combinatorial mapping and Illumina sequencing.