Sanjay Kumar Gupta
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Large-Scale Analysis of Genome and Transcriptome Alterations in Multiple Tumors Unveils Novel Cancer-Relevant Splicing Networks
Downloaded from genome.cshlp.org on September 28, 2021 - Published by Cold Spring Harbor Laboratory Press Research Large-scale analysis of genome and transcriptome alterations in multiple tumors unveils novel cancer-relevant splicing networks Endre Sebestyén,1,5 Babita Singh,1,5 Belén Miñana,1,2 Amadís Pagès,1 Francesca Mateo,3 Miguel Angel Pujana,3 Juan Valcárcel,1,2,4 and Eduardo Eyras1,4 1Universitat Pompeu Fabra, E08003 Barcelona, Spain; 2Centre for Genomic Regulation, E08003 Barcelona, Spain; 3Program Against Cancer Therapeutic Resistance (ProCURE), Catalan Institute of Oncology (ICO), Bellvitge Institute for Biomedical Research (IDIBELL), E08908 L’Hospitalet del Llobregat, Spain; 4Catalan Institution for Research and Advanced Studies, E08010 Barcelona, Spain Alternative splicing is regulated by multiple RNA-binding proteins and influences the expression of most eukaryotic genes. However, the role of this process in human disease, and particularly in cancer, is only starting to be unveiled. We system- atically analyzed mutation, copy number, and gene expression patterns of 1348 RNA-binding protein (RBP) genes in 11 solid tumor types, together with alternative splicing changes in these tumors and the enrichment of binding motifs in the alter- natively spliced sequences. Our comprehensive study reveals widespread alterations in the expression of RBP genes, as well as novel mutations and copy number variations in association with multiple alternative splicing changes in cancer drivers and oncogenic pathways. Remarkably, the altered splicing patterns in several tumor types recapitulate those of undifferen- tiated cells. These patterns are predicted to be mainly controlled by MBNL1 and involve multiple cancer drivers, including the mitotic gene NUMA1. We show that NUMA1 alternative splicing induces enhanced cell proliferation and centrosome am- plification in nontumorigenic mammary epithelial cells. -

The Zinc-Finger Protein CNBP Is Required for Forebrain Formation In
Development 130, 1367-1379 1367 © 2003 The Company of Biologists Ltd doi:10.1242/dev.00349 The zinc-finger protein CNBP is required for forebrain formation in the mouse Wei Chen1,2, Yuqiong Liang1, Wenjie Deng1, Ken Shimizu1, Amir M. Ashique1,2, En Li3 and Yi-Ping Li1,2,* 1Department of Cytokine Biology, The Forsyth Institute, Boston, MA 02115, USA 2Harvard-Forsyth Department of Oral Biology, Harvard School of Dental Medicine, Boston, MA 02115, USA 3Cardiovascular Research Center, Massachusetts General Hospital, Department of Medicine, Harvard Medical School, Charlestown, MA 02129, USA *Author for correspondence (e-mail: [email protected]) Accepted 19 December 2002 SUMMARY Mouse mutants have allowed us to gain significant insight (AME), headfolds and forebrain. In Cnbp–/– embryos, the into axis development. However, much remains to be visceral endoderm remains in the distal tip of the conceptus learned about the cellular and molecular basis of early and the ADE fails to form, whereas the node and notochord forebrain patterning. We describe a lethal mutation mouse form normally. A substantial reduction in cell proliferation strain generated using promoter-trap mutagenesis. The was observed in the anterior regions of Cnbp–/– embryos at mutants exhibit severe forebrain truncation in homozygous gastrulation and neural-fold stages. In these regions, Myc mouse embryos and various craniofacial defects in expression was absent, indicating CNBP targets Myc in heterozygotes. We show that the defects are caused by rostral head formation. Our findings demonstrate that disruption of the gene encoding cellular nucleic acid Cnbp is essential for the forebrain induction and binding protein (CNBP); Cnbp transgenic mice were able specification. -

Table S1 the Four Gene Sets Derived from Gene Expression Profiles of Escs and Differentiated Cells
Table S1 The four gene sets derived from gene expression profiles of ESCs and differentiated cells Uniform High Uniform Low ES Up ES Down EntrezID GeneSymbol EntrezID GeneSymbol EntrezID GeneSymbol EntrezID GeneSymbol 269261 Rpl12 11354 Abpa 68239 Krt42 15132 Hbb-bh1 67891 Rpl4 11537 Cfd 26380 Esrrb 15126 Hba-x 55949 Eef1b2 11698 Ambn 73703 Dppa2 15111 Hand2 18148 Npm1 11730 Ang3 67374 Jam2 65255 Asb4 67427 Rps20 11731 Ang2 22702 Zfp42 17292 Mesp1 15481 Hspa8 11807 Apoa2 58865 Tdh 19737 Rgs5 100041686 LOC100041686 11814 Apoc3 26388 Ifi202b 225518 Prdm6 11983 Atpif1 11945 Atp4b 11614 Nr0b1 20378 Frzb 19241 Tmsb4x 12007 Azgp1 76815 Calcoco2 12767 Cxcr4 20116 Rps8 12044 Bcl2a1a 219132 D14Ertd668e 103889 Hoxb2 20103 Rps5 12047 Bcl2a1d 381411 Gm1967 17701 Msx1 14694 Gnb2l1 12049 Bcl2l10 20899 Stra8 23796 Aplnr 19941 Rpl26 12096 Bglap1 78625 1700061G19Rik 12627 Cfc1 12070 Ngfrap1 12097 Bglap2 21816 Tgm1 12622 Cer1 19989 Rpl7 12267 C3ar1 67405 Nts 21385 Tbx2 19896 Rpl10a 12279 C9 435337 EG435337 56720 Tdo2 20044 Rps14 12391 Cav3 545913 Zscan4d 16869 Lhx1 19175 Psmb6 12409 Cbr2 244448 Triml1 22253 Unc5c 22627 Ywhae 12477 Ctla4 69134 2200001I15Rik 14174 Fgf3 19951 Rpl32 12523 Cd84 66065 Hsd17b14 16542 Kdr 66152 1110020P15Rik 12524 Cd86 81879 Tcfcp2l1 15122 Hba-a1 66489 Rpl35 12640 Cga 17907 Mylpf 15414 Hoxb6 15519 Hsp90aa1 12642 Ch25h 26424 Nr5a2 210530 Leprel1 66483 Rpl36al 12655 Chi3l3 83560 Tex14 12338 Capn6 27370 Rps26 12796 Camp 17450 Morc1 20671 Sox17 66576 Uqcrh 12869 Cox8b 79455 Pdcl2 20613 Snai1 22154 Tubb5 12959 Cryba4 231821 Centa1 17897 -

PDF Output of CLIC (Clustering by Inferred Co-Expression)
PDF Output of CLIC (clustering by inferred co-expression) Dataset: Num of genes in input gene set: 7 Total number of genes: 16493 CLIC PDF output has three sections: 1) Overview of Co-Expression Modules (CEMs) Heatmap shows pairwise correlations between all genes in the input query gene set. Red lines shows the partition of input genes into CEMs, ordered by CEM strength. Each row shows one gene, and the brightness of squares indicates its correlations with other genes. Gene symbols are shown at left side and on the top of the heatmap. 2) Details of each CEM and its expansion CEM+ Top panel shows the posterior selection probability (dataset weights) for top GEO series datasets. Bottom panel shows the CEM genes (blue rows) as well as expanded CEM+ genes (green rows). Each column is one GEO series dataset, sorted by their posterior probability of being selected. The brightness of squares indicates the gene's correlations with CEM genes in the corresponding dataset. CEM+ includes genes that co-express with CEM genes in high-weight datasets, measured by LLR score. 3) Details of each GEO series dataset and its expression profile: Top panel shows the detailed information (e.g. title, summary) for the GEO series dataset. Bottom panel shows the background distribution and the expression profile for CEM genes in this dataset. Overview of Co-Expression Modules (CEMs) with Dataset Weighting Scale of average Pearson correlations Num of Genes in Query Geneset: 7. Num of CEMs: 1. 0.0 0.2 0.4 0.6 0.8 1.0 Ctr9 Rtf1 Paf1 Cdc73 Leo1 Wdr61 Pex2 Ctr9 Rtf1 -

Protein Identities in Evs Isolated from U87-MG GBM Cells As Determined by NG LC-MS/MS
Protein identities in EVs isolated from U87-MG GBM cells as determined by NG LC-MS/MS. No. Accession Description Σ Coverage Σ# Proteins Σ# Unique Peptides Σ# Peptides Σ# PSMs # AAs MW [kDa] calc. pI 1 A8MS94 Putative golgin subfamily A member 2-like protein 5 OS=Homo sapiens PE=5 SV=2 - [GG2L5_HUMAN] 100 1 1 7 88 110 12,03704523 5,681152344 2 P60660 Myosin light polypeptide 6 OS=Homo sapiens GN=MYL6 PE=1 SV=2 - [MYL6_HUMAN] 100 3 5 17 173 151 16,91913397 4,652832031 3 Q6ZYL4 General transcription factor IIH subunit 5 OS=Homo sapiens GN=GTF2H5 PE=1 SV=1 - [TF2H5_HUMAN] 98,59 1 1 4 13 71 8,048185945 4,652832031 4 P60709 Actin, cytoplasmic 1 OS=Homo sapiens GN=ACTB PE=1 SV=1 - [ACTB_HUMAN] 97,6 5 5 35 917 375 41,70973209 5,478027344 5 P13489 Ribonuclease inhibitor OS=Homo sapiens GN=RNH1 PE=1 SV=2 - [RINI_HUMAN] 96,75 1 12 37 173 461 49,94108966 4,817871094 6 P09382 Galectin-1 OS=Homo sapiens GN=LGALS1 PE=1 SV=2 - [LEG1_HUMAN] 96,3 1 7 14 283 135 14,70620005 5,503417969 7 P60174 Triosephosphate isomerase OS=Homo sapiens GN=TPI1 PE=1 SV=3 - [TPIS_HUMAN] 95,1 3 16 25 375 286 30,77169764 5,922363281 8 P04406 Glyceraldehyde-3-phosphate dehydrogenase OS=Homo sapiens GN=GAPDH PE=1 SV=3 - [G3P_HUMAN] 94,63 2 13 31 509 335 36,03039959 8,455566406 9 Q15185 Prostaglandin E synthase 3 OS=Homo sapiens GN=PTGES3 PE=1 SV=1 - [TEBP_HUMAN] 93,13 1 5 12 74 160 18,68541938 4,538574219 10 P09417 Dihydropteridine reductase OS=Homo sapiens GN=QDPR PE=1 SV=2 - [DHPR_HUMAN] 93,03 1 1 17 69 244 25,77302971 7,371582031 11 P01911 HLA class II histocompatibility antigen, -

Supplemental Materials ZNF281 Enhances Cardiac Reprogramming
Supplemental Materials ZNF281 enhances cardiac reprogramming by modulating cardiac and inflammatory gene expression Huanyu Zhou, Maria Gabriela Morales, Hisayuki Hashimoto, Matthew E. Dickson, Kunhua Song, Wenduo Ye, Min S. Kim, Hanspeter Niederstrasser, Zhaoning Wang, Beibei Chen, Bruce A. Posner, Rhonda Bassel-Duby and Eric N. Olson Supplemental Table 1; related to Figure 1. Supplemental Table 2; related to Figure 1. Supplemental Table 3; related to the “quantitative mRNA measurement” in Materials and Methods section. Supplemental Table 4; related to the “ChIP-seq, gene ontology and pathway analysis” and “RNA-seq” and gene ontology analysis” in Materials and Methods section. Supplemental Figure S1; related to Figure 1. Supplemental Figure S2; related to Figure 2. Supplemental Figure S3; related to Figure 3. Supplemental Figure S4; related to Figure 4. Supplemental Figure S5; related to Figure 6. Supplemental Table S1. Genes included in human retroviral ORF cDNA library. Gene Gene Gene Gene Gene Gene Gene Gene Symbol Symbol Symbol Symbol Symbol Symbol Symbol Symbol AATF BMP8A CEBPE CTNNB1 ESR2 GDF3 HOXA5 IL17D ADIPOQ BRPF1 CEBPG CUX1 ESRRA GDF6 HOXA6 IL17F ADNP BRPF3 CERS1 CX3CL1 ETS1 GIN1 HOXA7 IL18 AEBP1 BUD31 CERS2 CXCL10 ETS2 GLIS3 HOXB1 IL19 AFF4 C17ORF77 CERS4 CXCL11 ETV3 GMEB1 HOXB13 IL1A AHR C1QTNF4 CFL2 CXCL12 ETV7 GPBP1 HOXB5 IL1B AIMP1 C21ORF66 CHIA CXCL13 FAM3B GPER HOXB6 IL1F3 ALS2CR8 CBFA2T2 CIR1 CXCL14 FAM3D GPI HOXB7 IL1F5 ALX1 CBFA2T3 CITED1 CXCL16 FASLG GREM1 HOXB9 IL1F6 ARGFX CBFB CITED2 CXCL3 FBLN1 GREM2 HOXC4 IL1F7 -
Supplemental Tables4.Pdf
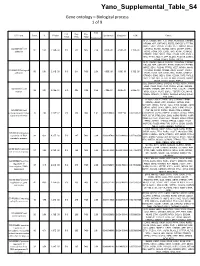
Yano_Supplemental_Table_S4 Gene ontology – Biological process 1 of 9 Fold List Pop Pop GO Term Count % PValue Bonferroni Benjamini FDR Genes Total Hits Total Enrichment DLC1, CADM1, NELL2, CLSTN1, PCDHGA8, CTNNB1, NRCAM, APP, CNTNAP2, FERT2, RAPGEF1, PTPRM, MPDZ, SDK1, PCDH9, PTPRS, VEZT, NRXN1, MYH9, GO:0007155~cell CTNNA2, NCAM1, NCAM2, DDR1, LSAMP, CNTN1, 50 5.61 2.14E-08 510 311 7436 2.34 4.50E-05 4.50E-05 3.70E-05 adhesion ROR2, VCAN, DST, LIMS1, TNC, ASTN1, CTNND2, CTNND1, CDH2, NEO1, CDH4, CD24A, FAT3, PVRL3, TRO, TTYH1, MLLT4, LPP, NLGN1, PCDH19, LAMA1, ITGA9, CDH13, CDON, PSPC1 DLC1, CADM1, NELL2, CLSTN1, PCDHGA8, CTNNB1, NRCAM, APP, CNTNAP2, FERT2, RAPGEF1, PTPRM, MPDZ, SDK1, PCDH9, PTPRS, VEZT, NRXN1, MYH9, GO:0022610~biological CTNNA2, NCAM1, NCAM2, DDR1, LSAMP, CNTN1, 50 5.61 2.14E-08 510 311 7436 2.34 4.50E-05 4.50E-05 3.70E-05 adhesion ROR2, VCAN, DST, LIMS1, TNC, ASTN1, CTNND2, CTNND1, CDH2, NEO1, CDH4, CD24A, FAT3, PVRL3, TRO, TTYH1, MLLT4, LPP, NLGN1, PCDH19, LAMA1, ITGA9, CDH13, CDON, PSPC1 DCC, ENAH, PLXNA2, CAPZA2, ATP5B, ASTN1, PAX6, ZEB2, CDH2, CDH4, GLI3, CD24A, EPHB1, NRCAM, GO:0006928~cell CTTNBP2, EDNRB, APP, PTK2, ETV1, CLASP2, STRBP, 36 4.04 3.46E-07 510 205 7436 2.56 7.28E-04 3.64E-04 5.98E-04 motion NRG1, DCLK1, PLAT, SGPL1, TGFBR1, EVL, MYH9, YWHAE, NCKAP1, CTNNA2, SEMA6A, EPHA4, NDEL1, FYN, LRP6 PLXNA2, ADCY5, PAX6, GLI3, CTNNB1, LPHN2, EDNRB, LPHN3, APP, CSNK2A1, GPR45, NRG1, RAPGEF1, WWOX, SGPL1, TLE4, SPEN, NCAM1, DDR1, GRB10, GRM3, GNAQ, HIPK1, GNB1, HIPK2, PYGO1, GO:0007166~cell RNF138, ROR2, CNTN1, -

Immunoprecipitation and Mass Spectrometry Defines an Extensive
BRES : 44759 Model7 pp: À 1221ðcol:fig: : NILÞ brain research ] ( ]]]]) ]]]– ]]] Available online at www.sciencedirect.com 121 122 123 124 125 126 www.elsevier.com/locate/brainres 127 128 129 Review 130 131 fi 132 Immunoprecipitation and mass spectrometry de nes 133 – 134 an extensive RBM45 protein protein interaction 135 Q2 136 network 137 138 a a,b a a c 139 Yang Li , Mahlon Collins , Jiyan An , Rachel Geiser , Tony Tegeler , c c c a,b,n 140 Q1 Kristine Tsantilas , Krystine Garcia , Patrick Pirrotte , Robert Bowser 141 aDivisions of Neurology and Neurobiology, Barrow Neurological Institute, St. Joseph's Hospital and Medical Center, 142 Phoenix, AZ 85013, USA 143 bUniversity of Pittsburgh School of Medicine, Pittsburgh, PA 15261, USA 144 cCenter for Proteomics, TGen (Translational Genomics Research Institute), Phoenix, AZ 85004, USA 145 146 147 article info abstract 148 149 Article history: The pathological accumulation of RNA-binding proteins (RBPs) within inclusion bodies is a 150 Received 30 January 2016 hallmark of amyotrophic lateral sclerosis (ALS) and frontotemporal lobar degeneration 151 Received in revised form (FTLD). RBP aggregation results in both toxic gain and loss of normal function. Determining 152 25 February 2016 the protein binding partners and normal functions of disease-associated RBPs is necessary 153 Accepted 28 February 2016 to fully understand molecular mechanisms of RBPs in disease. Herein, we characterized 154 the protein–protein interactions (PPIs) of RBM45, a RBP that localizes to inclusions in ALS/ 155 – fi Keywords: FTLD. Using immunoprecipitation coupled to mass spectrometry (IP MS), we identi ed 132 156 fi RBM45 proteins that speci cally interact with RBM45 within HEK293 cells. -

Comparative Interactomics Analysis of Different ALS-Associated Proteins
Acta Neuropathol (2016) 132:175–196 DOI 10.1007/s00401-016-1575-8 ORIGINAL PAPER Comparative interactomics analysis of different ALS‑associated proteins identifies converging molecular pathways Anna M. Blokhuis1 · Max Koppers1,2 · Ewout J. N. Groen1,2,9 · Dianne M. A. van den Heuvel1 · Stefano Dini Modigliani4 · Jasper J. Anink5,6 · Katsumi Fumoto1,10 · Femke van Diggelen1 · Anne Snelting1 · Peter Sodaar2 · Bert M. Verheijen1,2 · Jeroen A. A. Demmers7 · Jan H. Veldink2 · Eleonora Aronica5,6 · Irene Bozzoni3 · Jeroen den Hertog8 · Leonard H. van den Berg2 · R. Jeroen Pasterkamp1 Received: 22 January 2016 / Revised: 14 April 2016 / Accepted: 15 April 2016 / Published online: 10 May 2016 © The Author(s) 2016. This article is published with open access at Springerlink.com Abstract Amyotrophic lateral sclerosis (ALS) is a dev- OPTN and UBQLN2, in which mutations caused loss or astating neurological disease with no effective treatment gain of protein interactions. Several of the identified inter- available. An increasing number of genetic causes of ALS actomes showed a high degree of overlap: shared binding are being identified, but how these genetic defects lead to partners of ATXN2, FUS and TDP-43 had roles in RNA motor neuron degeneration and to which extent they affect metabolism; OPTN- and UBQLN2-interacting proteins common cellular pathways remains incompletely under- were related to protein degradation and protein transport, stood. To address these questions, we performed an inter- and C9orf72 interactors function in mitochondria. To con- actomic analysis to identify binding partners of wild-type firm that this overlap is important for ALS pathogenesis, (WT) and ALS-associated mutant versions of ATXN2, we studied fragile X mental retardation protein (FMRP), C9orf72, FUS, OPTN, TDP-43 and UBQLN2 in neuronal one of the common interactors of ATXN2, FUS and TDP- cells. -

1 Title 1 Loss of PABPC1 Is Compensated by Elevated PABPC4
bioRxiv preprint doi: https://doi.org/10.1101/2021.02.07.430165; this version posted February 15, 2021. The copyright holder for this preprint (which was not certified by peer review) is the author/funder. All rights reserved. No reuse allowed without permission. 1 1 Title 2 Loss of PABPC1 is compensated by elevated PABPC4 and correlates with transcriptome 3 changes 4 5 Jingwei Xie1, 2, Xiaoyu Wei1, Yu Chen1 6 7 1 Department of Biochemistry and Groupe de recherche axé sur la structure des 8 protéines, McGill University, Montreal, Quebec H3G 0B1, Canada 9 10 2 To whom correspondence should be addressed: Dept. of Biochemistry, McGill 11 University, Montreal, QC H3G 0B1, Canada. E-mail: [email protected]. 12 13 14 15 Abstract 16 Cytoplasmic poly(A) binding protein (PABP) is an essential translation factor that binds to 17 the 3' tail of mRNAs to promote translation and regulate mRNA stability. PABPC1 is the 18 most abundant of several PABP isoforms that exist in mammals. Here, we used the 19 CRISPR/Cas genome editing system to shift the isoform composition in HEK293 cells. 20 Disruption of PABPC1 elevated PABPC4 levels. Transcriptome analysis revealed that the 21 shift in the dominant PABP isoform was correlated with changes in key transcriptional 22 regulators. This study provides insight into understanding the role of PABP isoforms in 23 development and differentiation. 24 Keywords 25 PABPC1, PABPC4, c-Myc 26 bioRxiv preprint doi: https://doi.org/10.1101/2021.02.07.430165; this version posted February 15, 2021. The copyright holder for this preprint (which was not certified by peer review) is the author/funder. -

PABPC4 293T Cell Transient Overexpression Lysate(Denatured)
Produktinformation Diagnostik & molekulare Diagnostik Laborgeräte & Service Zellkultur & Verbrauchsmaterial Forschungsprodukte & Biochemikalien Weitere Information auf den folgenden Seiten! See the following pages for more information! Lieferung & Zahlungsart Lieferung: frei Haus Bestellung auf Rechnung SZABO-SCANDIC Lieferung: € 10,- HandelsgmbH & Co KG Erstbestellung Vorauskassa Quellenstraße 110, A-1100 Wien T. +43(0)1 489 3961-0 Zuschläge F. +43(0)1 489 3961-7 [email protected] • Mindermengenzuschlag www.szabo-scandic.com • Trockeneiszuschlag • Gefahrgutzuschlag linkedin.com/company/szaboscandic • Expressversand facebook.com/szaboscandic PABPC4 293T Cell Transient Overexpression Lysate(Denatured) Catalog # : H00008761-T01 規格 : [ 100 uL ] List All Specification Application Image Transfected 293T Western Blot Cell Line: Plasmid: pCMV-PABPC4 full-length Host: Human Theoretical MW 72.71 (kDa): Quality Control Transient overexpression cell lysate was tested with Anti-PABPC4 Testing: antibody (H00008761-B01) by Western Blots. SDS-PAGE Gel PABPC4 transfected lysate. Western Blot Lane 1: PABPC4 transfected lysate ( 72.71 KDa) Lane 2: Non-transfected lysate. Storage Buffer: 1X Sample Buffer (50 mM Tris-HCl, 2% SDS, 10% glycerol, 300 mM 2- mercaptoethanol, 0.01% Bromophenol blue) Storage Store at -80°C. Aliquot to avoid repeated freezing and thawing. Instruction: MSDS: Download Applications Page 1 of 2 2016/5/22 Western Blot Gene Information Entrez GeneID: 8761 GeneBank BC071591.1 Accession#: Protein AAH71591.1 Accession#: Gene Name: PABPC4 Gene Alias: APP-1,APP1,FLJ43938,PABP4,iPABP Gene poly(A) binding protein, cytoplasmic 4 (inducible form) Description: Omim ID: 603407 Gene Ontology: Hyperlink Gene Summary: Poly(A)-binding proteins (PABPs) bind to the poly(A) tail present at the 3-prime ends of most eukaryotic mRNAs. -

Supplementary Table S4. FGA Co-Expressed Gene List in LUAD
Supplementary Table S4. FGA co-expressed gene list in LUAD tumors Symbol R Locus Description FGG 0.919 4q28 fibrinogen gamma chain FGL1 0.635 8p22 fibrinogen-like 1 SLC7A2 0.536 8p22 solute carrier family 7 (cationic amino acid transporter, y+ system), member 2 DUSP4 0.521 8p12-p11 dual specificity phosphatase 4 HAL 0.51 12q22-q24.1histidine ammonia-lyase PDE4D 0.499 5q12 phosphodiesterase 4D, cAMP-specific FURIN 0.497 15q26.1 furin (paired basic amino acid cleaving enzyme) CPS1 0.49 2q35 carbamoyl-phosphate synthase 1, mitochondrial TESC 0.478 12q24.22 tescalcin INHA 0.465 2q35 inhibin, alpha S100P 0.461 4p16 S100 calcium binding protein P VPS37A 0.447 8p22 vacuolar protein sorting 37 homolog A (S. cerevisiae) SLC16A14 0.447 2q36.3 solute carrier family 16, member 14 PPARGC1A 0.443 4p15.1 peroxisome proliferator-activated receptor gamma, coactivator 1 alpha SIK1 0.435 21q22.3 salt-inducible kinase 1 IRS2 0.434 13q34 insulin receptor substrate 2 RND1 0.433 12q12 Rho family GTPase 1 HGD 0.433 3q13.33 homogentisate 1,2-dioxygenase PTP4A1 0.432 6q12 protein tyrosine phosphatase type IVA, member 1 C8orf4 0.428 8p11.2 chromosome 8 open reading frame 4 DDC 0.427 7p12.2 dopa decarboxylase (aromatic L-amino acid decarboxylase) TACC2 0.427 10q26 transforming, acidic coiled-coil containing protein 2 MUC13 0.422 3q21.2 mucin 13, cell surface associated C5 0.412 9q33-q34 complement component 5 NR4A2 0.412 2q22-q23 nuclear receptor subfamily 4, group A, member 2 EYS 0.411 6q12 eyes shut homolog (Drosophila) GPX2 0.406 14q24.1 glutathione peroxidase