Pmtv, Pomovirus)
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Wo 2007/056463 A2
(12) INTERNATIONAL APPLICATION PUBLISHED UNDER THE PATENT COOPERATION TREATY (PCT) (19) World Intellectual Property Organization International Bureau (10) International Publication Number (43) International Publication Date PCT 18 May 2007 (18.05.2007) WO 2007/056463 A2 (51) International Patent Classification: (74) Agents: WILLIAMS, Kathleen et al.; Edwards Angell C12Q 1/70 (2006.01) C12Q 1/68 (2006.01) Palmer & Dodge LLP, P.O. Box 55874, Boston, MA 02205 (US). (21) International Application Number: (81) Designated States (unless otherwise indicated, for every PCT/US2006/043502 kind of national protection available): AE, AG, AL, AM, AT,AU, AZ, BA, BB, BG, BR, BW, BY, BZ, CA, CH, CN, (22) International Filing Date: CO, CR, CU, CZ, DE, DK, DM, DZ, EC, EE, EG, ES, FI, 9 November 2006 (09.1 1.2006) GB, GD, GE, GH, GM, GT, HN, HR, HU, ID, IL, IN, IS, JP, KE, KG, KM, KN, KP, KR, KZ, LA, LC, LK, LR, LS, (25) Filing Language: English LT, LU, LV,LY,MA, MD, MG, MK, MN, MW, MX, MY, MZ, NA, NG, NI, NO, NZ, OM, PG, PH, PL, PT, RO, RS, (26) Publication Language: English RU, SC, SD, SE, SG, SK, SL, SM, SV, SY, TJ, TM, TN, TR, TT, TZ, UA, UG, US, UZ, VC, VN, ZA, ZM, ZW (30) Priority Data: (84) Designated States (unless otherwise indicated, for every 60/735,085 9 November 2005 (09. 11.2005) US kind of regional protection available): ARIPO (BW, GH, GM, KE, LS, MW, MZ, NA, SD, SL, SZ, TZ, UG, ZM, (71) Applicant (for all designated States except US): ZW), Eurasian (AM, AZ, BY, KG, KZ, MD, RU, TJ, TM), PRIMERA BIOSYSTEMS, INC. -

ICTV Code Assigned: 2011.001Ag Officers)
This form should be used for all taxonomic proposals. Please complete all those modules that are applicable (and then delete the unwanted sections). For guidance, see the notes written in blue and the separate document “Help with completing a taxonomic proposal” Please try to keep related proposals within a single document; you can copy the modules to create more than one genus within a new family, for example. MODULE 1: TITLE, AUTHORS, etc (to be completed by ICTV Code assigned: 2011.001aG officers) Short title: Change existing virus species names to non-Latinized binomials (e.g. 6 new species in the genus Zetavirus) Modules attached 1 2 3 4 5 (modules 1 and 9 are required) 6 7 8 9 Author(s) with e-mail address(es) of the proposer: Van Regenmortel Marc, [email protected] Burke Donald, [email protected] Calisher Charles, [email protected] Dietzgen Ralf, [email protected] Fauquet Claude, [email protected] Ghabrial Said, [email protected] Jahrling Peter, [email protected] Johnson Karl, [email protected] Holbrook Michael, [email protected] Horzinek Marian, [email protected] Keil Guenther, [email protected] Kuhn Jens, [email protected] Mahy Brian, [email protected] Martelli Giovanni, [email protected] Pringle Craig, [email protected] Rybicki Ed, [email protected] Skern Tim, [email protected] Tesh Robert, [email protected] Wahl-Jensen Victoria, [email protected] Walker Peter, [email protected] Weaver Scott, [email protected] List the ICTV study group(s) that have seen this proposal: A list of study groups and contacts is provided at http://www.ictvonline.org/subcommittees.asp . -

Overlapping Genes Produce Proteins with Unusual Sequence Properties And
JVI Accepts, published online ahead of print on 29 July 2009 J. Virol. doi:10.1128/JVI.00595-09 Copyright © 2009, American Society for Microbiology and/or the Listed Authors/Institutions. All Rights Reserved. 1 Overlapping genes produce proteins with unusual sequence properties and 2 offer insight into de novo protein creation 3 4 Corinne Rancurel1, Mahvash Khosravi2, Keith A. Dunker2, Pedro R. Romero2*, and David Karlin3* 5 6 1 Architecture et Fonction des Macromolécules Biologiques, Case 932, Campus de Luminy, 13288 Marseille 7 Cedex 9, France 8 2 Center for Computational Biology and Bioinformatics, 410 West 10th Street, Suite 5000, Indiana University 9 - Purdue University, Indianapolis, IN 46202-5122, USA, Downloaded from 10 3 25, rue de Casssis, 13008 Marseille, FRANCE 11 12 * Corresponding authors: [email protected], [email protected] 13 14 Running Title : Overlapping proteins have unusual sequence properties http://jvi.asm.org/ 15 Keywords 16 de novo gene creation; de novo protein creation; novel proteins; new proteins; orphan proteins; orphan 17 genes; ORFans; overlapping genes; overlapping reading frames; overprinting; unstructured proteins; 18 disordered proteins; intrinsic disorder; structural disorder; disorder prediction; profile-profile comparison; on April 22, 2020 by guest 19 PFAM; viral genomics; viral bioinformatics; viral structural genomics. 20 21 Abbreviations 22 Only abbreviations used in the main text are listed here; others are given in the figure captions. 23 30K, conserved domain of the 30K family of movement proteins; aa, amino acid; dsRNA, double-stranded 24 RNA; GT, guanylyltransferase; MP, movement protein; MT, methyltransferase; N, nucleoprotein; NSs, non 25 structural protein of orthobunyaviruses; ORF, open reading frame; PDB, protein databank (database of 26 protein structures); PFAM, Protein families (database of families of protein sequences); ssRNA, single- 27 stranded RNA; TGB, triple gene block; RE, relative (compositional) entropy; tm, transmembrane segment. -

Small Hydrophobic Viral Proteins Involved in Intercellular Movement of Diverse Plant Virus Genomes Sergey Y
AIMS Microbiology, 6(3): 305–329. DOI: 10.3934/microbiol.2020019 Received: 23 July 2020 Accepted: 13 September 2020 Published: 21 September 2020 http://www.aimspress.com/journal/microbiology Review Small hydrophobic viral proteins involved in intercellular movement of diverse plant virus genomes Sergey Y. Morozov1,2,* and Andrey G. Solovyev1,2,3 1 A. N. Belozersky Institute of Physico-Chemical Biology, Moscow State University, Moscow, Russia 2 Department of Virology, Biological Faculty, Moscow State University, Moscow, Russia 3 Institute of Molecular Medicine, Sechenov First Moscow State Medical University, Moscow, Russia * Correspondence: E-mail: [email protected]; Tel: +74959393198. Abstract: Most plant viruses code for movement proteins (MPs) targeting plasmodesmata to enable cell-to-cell and systemic spread in infected plants. Small membrane-embedded MPs have been first identified in two viral transport gene modules, triple gene block (TGB) coding for an RNA-binding helicase TGB1 and two small hydrophobic proteins TGB2 and TGB3 and double gene block (DGB) encoding two small polypeptides representing an RNA-binding protein and a membrane protein. These findings indicated that movement gene modules composed of two or more cistrons may encode the nucleic acid-binding protein and at least one membrane-bound movement protein. The same rule was revealed for small DNA-containing plant viruses, namely, viruses belonging to genus Mastrevirus (family Geminiviridae) and the family Nanoviridae. In multi-component transport modules the nucleic acid-binding MP can be viral capsid protein(s), as in RNA-containing viruses of the families Closteroviridae and Potyviridae. However, membrane proteins are always found among MPs of these multicomponent viral transport systems. -

Type D Primate Retroviruses: a Review1
[CANCER RESEARCH 38, 3123-3139, October 1978] Type D Primate Retroviruses: A Review1 Donald Fine and Gerald Schochetman Viral Oncology Program, Frederick Cancer Research Center. Frederick, Maryland 21701 Abstract The prototype virus of the type D retroviruses is the techniques. The retrovirus associated with the induction of Mason-Pfizer monkey virus (MPMV). MPMV was originally mammary cancer in mice, MMTV,2 is morphologically dis isolated from a breast carcinoma of a female rhesus tinct from the type C viruses and is the prototype of the type monkey (an Old World monkey). MPMV is of obvious B virus group (30). A retrovirus group of primate origin, importance in that it is the only retrovirus thus far isolated which has properties similar to the type C and type B from a mammary tumor of a primate and has been shown viruses but is morphologically distinct from them, has been to have transforming potential for primate cells in vitro. designated genus Oncornavirus D (30). The prototype virus Subsequent to the isolation of MPMV, viruses morpholog of this genus is the MPMV. MPMV was originally isolated ically and ¡mmunologicallyindistinguishable from MPMV from a mammary carcinoma of a female rhesus monkey (an have been isolated from normal placenta and lactating Old World monkey) (19, 60). Because mammary carcinomas mammary glands of other rhesus monkeys in captivity. represent the most prevalent form of neoplasia in humans Recently, viruses morphologically resembling MPMV have and, furthermore, because the incidence of mammary tu been isolated from a langur monkey (another Old World mors in subhuman primates is rare (61, 62, 70, 78), MPMV monkey) and from squirrel monkeys (a New World mon is of obvious potential importance because it is the only key). -
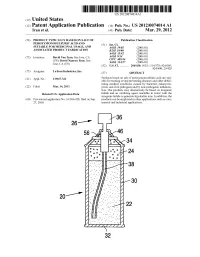
Us 2012/0074.014 A1 2 .. 1
US 2012O074O14A1 (19) United States (12) Patent Application Publication (10) Pub. No.: US 2012/0074.014 A1 Tran et al. (43) Pub. Date: Mar. 29, 2012 (54) PRODUCT TYPICALLY BASED ON SALT OF Publication Classification PEROXYMONOSULFURIC ACID AND (51) Int. Cl SUITABLE FOR MEDICINAL USAGE, AND iBio/02 (2006.01) ASSOCATED PRODUCT FABRICATION B23P 19/00 (2006.01) A633/42 (2006.01) (75) Inventors: David Van Tran, San Jose, CA A6IR 9/14 (2006.01) (US); David Nguyen Tran, San CD7C 409/244 (2006.01) Jose, CA (US) A6II 3/327 (2006.01) s (52) U.S. Cl. ............. 206/438: 562/1; 514/578; 424/605; 424/400; 29/428 (73) Assignee: LuTran Industries, Inc. (57) ABSTRACT 21) Appl. No.: 13AO47,742 Products based on salt of peroxyperoxVmonosulfuric acid are suit (21) Appl. No 9 able for treating or/and preventing diseases and other debili tating medical conditions caused by bacterial, eukaryotic, (22) Filed: Mar. 14, 2011 prion, and viral pathogens and by non-pathogenic inflamma tion. The products may alternatively be based on inorganic Related U.S. Application Data halide and an oxidizing agent reactable in water with the inorganic halide to generate hypohalite ions. In addition, the (60) Provisional application No. 61/386,928, filed on Sep. products can be employed in other applications such as com 27, 2010. mercial and industrial applications. 5T. 2 .. 2. 1.4. J., Patent Application Publication Mar. 29, 2012 Sheet 1 of 2 US 2012/0074014 A1 cro Q-D 22 Fig. 2a Fig.2b Patent Application Publication Mar. 29, 2012 Sheet 2 of 2 US 2012/0074014 A1 90 92 US 2012/0074014 A1 Mar. -

Evidence to Support Safe Return to Clinical Practice by Oral Health Professionals in Canada During the COVID-19 Pandemic: a Repo
Evidence to support safe return to clinical practice by oral health professionals in Canada during the COVID-19 pandemic: A report prepared for the Office of the Chief Dental Officer of Canada. November 2020 update This evidence synthesis was prepared for the Office of the Chief Dental Officer, based on a comprehensive review under contract by the following: Paul Allison, Faculty of Dentistry, McGill University Raphael Freitas de Souza, Faculty of Dentistry, McGill University Lilian Aboud, Faculty of Dentistry, McGill University Martin Morris, Library, McGill University November 30th, 2020 1 Contents Page Introduction 3 Project goal and specific objectives 3 Methods used to identify and include relevant literature 4 Report structure 5 Summary of update report 5 Report results a) Which patients are at greater risk of the consequences of COVID-19 and so 7 consideration should be given to delaying elective in-person oral health care? b) What are the signs and symptoms of COVID-19 that oral health professionals 9 should screen for prior to providing in-person health care? c) What evidence exists to support patient scheduling, waiting and other non- treatment management measures for in-person oral health care? 10 d) What evidence exists to support the use of various forms of personal protective equipment (PPE) while providing in-person oral health care? 13 e) What evidence exists to support the decontamination and re-use of PPE? 15 f) What evidence exists concerning the provision of aerosol-generating 16 procedures (AGP) as part of in-person -

Dissecting the Molecular Mechanism of the Subcellular Localization And
www.nature.com/scientificreports OPEN Dissecting the Molecular Mechanism of the Subcellular Localization and Cell-to-cell Received: 7 April 2017 Accepted: 9 August 2017 Movement of the Sugarcane mosaic Published: xx xx xxxx virus P3N-PIPO Guangyuan Cheng1, Meng Dong1, Qian Xu1, Lei Peng1, Zongtao Yang1, Taiyun Wei2 & Jingsheng Xu1 The coding sequence of P3N-PIPO was cloned by fusion PCR from Sugarcane mosaic virus (SCMV), a main causal agent of sugarcane (Saccharum spp. hybrid) mosaic disease. SCMV P3N-PIPO preferentially localized to the plasma membrane (PM) compared with the plasmodesmata (PD), as demonstrated by transient expression and plasmolysis assays in the leaf epidermal cells of Nicotiana benthamiana. The subcellular localization of the P3N-PIPO mutants P3N-PIPOT1 and P3N-PIPOT2 with 29 and 63 amino acids deleted from the C-terminus of PIPO, respectively, revealed that the 19 amino acids at the N-terminus of PIPO contributed to the PD localization. Interaction assays showed that the 63 amino acids at the C-terminus of PIPO determined the P3N-PIPO interaction with PM-associated Ca2+-binding protein 1, ScPCaP1, which was isolated from the SCMV-susceptible sugarcane cultivar Badila. Like wild- type P3N-PIPO, P3N-PIPOT1 and P3N-PIPOT2 could translocate to neighbouring cells and recruit the SCMV cylindrical inclusion protein to the PM. Thus, interactions with ScPCaP1 may contribute to, but not determine, SCMV Pm3N-PIPO’s localization to the PM or PD. These results also imply the existence of truncated P3N-PIPO in nature. Te movement of a virus from an infected cell to an adjacent cell through the plasmodesmata (PD) is an impor- tant step in establishing a systemic infection in a host1, 2. -

Armillaria Root Rot Fungi Host Single-Stranded RNA Viruses
www.nature.com/scientificreports OPEN Armillaria root rot fungi host single‑stranded RNA viruses Riikka Linnakoski1,5, Suvi Sutela1,5, Martin P. A. Coetzee2, Tuan A. Duong2, Igor N. Pavlov3,4, Yulia A. Litovka3,4, Jarkko Hantula1, Brenda D. Wingfeld2 & Eeva J. Vainio1* Species of Armillaria are distributed globally and include some of the most important pathogens of forest and ornamental trees. Some of them form large long‑living clones that are considered as one of the largest organisms on earth and are capable of long‑range spore‑mediated transfer as well as vegetative spread by drought‑resistant hyphal cords called rhizomorphs. However, the virus community infecting these species has remained unknown. In this study we used dsRNA screening and high‑throughput sequencing to search for possible virus infections in a collection of Armillaria isolates representing three diferent species: Armillaria mellea from South Africa, A. borealis from Finland and Russia (Siberia) and A. cepistipes from Finland. Our analysis revealed the presence of both negative‑ sense RNA viruses and positive‑sense RNA viruses, while no dsRNA viruses were detected. The viruses included putative new members of virus families Mymonaviridae, Botourmiaviridae and Virgaviridae and members of a recently discovered virus group tentatively named “ambiviruses” with ambisense bicistronic genomic organization. We demonstrated that Armillaria isolates can be cured of viruses by thermal treatment, which enables the examination of virus efects on host growth and phenotype using isogenic virus‑infected and virus‑free strains. Te fungal genus Armillaria (Fr.) Staude includes more than 40 described species1. Tey are mainly known as notorious plant pathogens of managed natural forests and plantations of non-native tree species that infect hundreds of diferent plants, including economically important conifers (e.g. -

RNA/RNA Interactions Involved in the Regulation of Benyviridae Viral Cicle Mattia Dall’Ara
RNA/RNA interactions involved in the regulation of Benyviridae viral cicle Mattia Dall’Ara To cite this version: Mattia Dall’Ara. RNA/RNA interactions involved in the regulation of Benyviridae viral cicle. Vegetal Biology. Université de Strasbourg; Università degli studi (Bologne, Italie), 2018. English. NNT : 2018STRAJ019. tel-02003448 HAL Id: tel-02003448 https://tel.archives-ouvertes.fr/tel-02003448 Submitted on 1 Feb 2019 HAL is a multi-disciplinary open access L’archive ouverte pluridisciplinaire HAL, est archive for the deposit and dissemination of sci- destinée au dépôt et à la diffusion de documents entific research documents, whether they are pub- scientifiques de niveau recherche, publiés ou non, lished or not. The documents may come from émanant des établissements d’enseignement et de teaching and research institutions in France or recherche français ou étrangers, des laboratoires abroad, or from public or private research centers. publics ou privés. Université de Strasbourg et Università di Bologna Thèse en co-tutelle Présentée à la FACULTÉ DES SCIENCES DE LA VIE En vue de l'obtention du titre de DOCTEUR DE L'UNIVERSITÉ DE STRASBOURG Discipline : Sciences de la Vie et de la Santé, Spécialité : Aspects moléculaires et cellulaires de la biologie Par Mattia Dall’Ara RNA/RNA interactions involved in the regulation of Benyviridae viral cicle Soutenue publiquement le 18 mai 2018 devant la Commission d'Examen : Dr. Stéphane BLANC Rapporteur Externe Dr. Renato BRANDIMARTI Rapporteur Externe Dr. Roland MARQUET Examinateur Interne Dr. Mirco IOTTI Examinateur Interne Pr. David GILMER Directeur de Thèse Dr. Claudio RATTI Co-directeur de Thèse DISTAL Dipartimento di Scienze e Tecnologie Agro-Alimentari Bologne Italie Acknowledgments First of all I want to thank my tutors David and Claudio. -

Multipartite Viruses: Organization, Emergence and Evolution
UNIVERSIDAD AUTÓNOMA DE MADRID FACULTAD DE CIENCIAS DEPARTAMENTO DE BIOLOGÍA MOLECULAR MULTIPARTITE VIRUSES: ORGANIZATION, EMERGENCE AND EVOLUTION TESIS DOCTORAL Adriana Lucía Sanz García Madrid, 2019 MULTIPARTITE VIRUSES Organization, emergence and evolution TESIS DOCTORAL Memoria presentada por Adriana Luc´ıa Sanz Garc´ıa Licenciada en Bioqu´ımica por la Universidad Autonoma´ de Madrid Supervisada por Dra. Susanna Manrubia Cuevas Centro Nacional de Biotecnolog´ıa (CSIC) Memoria presentada para optar al grado de Doctor en Biociencias Moleculares Facultad de Ciencias Departamento de Biolog´ıa Molecular Universidad Autonoma´ de Madrid Madrid, 2019 Tesis doctoral Multipartite viruses: Organization, emergence and evolution, 2019, Madrid, Espana. Memoria presentada por Adriana Luc´ıa-Sanz, licenciada en Bioqumica´ y con un master´ en Biof´ısica en la Universidad Autonoma´ de Madrid para optar al grado de doctor en Biociencias Moleculares del departamento de Biolog´ıa Molecular en la facultad de Ciencias de la Universidad Autonoma´ de Madrid Supervisora de tesis: Dr. Susanna Manrubia Cuevas. Investigadora Cient´ıfica en el Centro Nacional de Biotecnolog´ıa (CSIC), C/ Darwin 3, 28049 Madrid, Espana. to the reader CONTENTS Acknowledgments xi Resumen xiii Abstract xv Introduction xvii I.1 What is a virus? xvii I.2 What is a multipartite virus? xix I.3 The multipartite lifecycle xx I.4 Overview of this thesis xxv PART I OBJECTIVES PART II METHODOLOGY 0.5 Database management for constructing the multipartite and segmented datasets 3 0.6 Analytical -

The Evolution of Genome Compression and Genomic Novelty in RNA Viruses
Downloaded from genome.cshlp.org on October 3, 2021 - Published by Cold Spring Harbor Laboratory Press Letter The evolution of genome compression and genomic novelty in RNA viruses Robert Belshaw,1,3 Oliver G. Pybus,1 and Andrew Rambaut2 1Department of Zoology, University of Oxford, Oxford OX1 3PS, United Kingdom; 2Institute of Evolutionary Biology, University of Edinburgh, Edinburgh EH9 3JT, United Kingdom The genomes of RNA viruses are characterized by their extremely small size and extremely high mutation rates (typically 10 kb and 10−4/base/replication cycle, respectively), traits that are thought to be causally linked. One aspect of their small size is the genome compression caused by the use of overlapping genes (where some nucleotides code for two genes). Using a comparative analysis of all known RNA viral species, we show that viruses with larger genomes tend to have less gene overlap. We provide a numerical model to show how a high mutation rate could lead to gene overlap, and we discuss the factors that might explain the observed relationship between gene overlap and genome size. We also propose a model for the evolution of gene overlap based on the co-opting of previously unused ORFs, which gives rise to two types of overlap: (1) the creation of novel genes inside older genes, predominantly via +1 frameshifts, and (2) the incremental increase in overlap between originally contiguous genes, with no frameshift preference. Both types of overlap are viewed as the creation of genomic novelty under pressure for genome compression. Simulations based on our model generate the empirical size distributions of overlaps and explain the observed frameshift preferences.