G Protein-Coupled Receptor Systems As Crucial Regulators of DNA Damage
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Genome-Wide Analysis of 5-Hmc in the Peripheral Blood of Systemic Lupus Erythematosus Patients Using an Hmedip-Chip
INTERNATIONAL JOURNAL OF MOLECULAR MEDICINE 35: 1467-1479, 2015 Genome-wide analysis of 5-hmC in the peripheral blood of systemic lupus erythematosus patients using an hMeDIP-chip WEIGUO SUI1*, QIUPEI TAN1*, MING YANG1, QIANG YAN1, HUA LIN1, MINGLIN OU1, WEN XUE1, JIEJING CHEN1, TONGXIANG ZOU1, HUANYUN JING1, LI GUO1, CUIHUI CAO1, YUFENG SUN1, ZHENZHEN CUI1 and YONG DAI2 1Guangxi Key Laboratory of Metabolic Diseases Research, Central Laboratory of Guilin 181st Hospital, Guilin, Guangxi 541002; 2Clinical Medical Research Center, the Second Clinical Medical College of Jinan University (Shenzhen People's Hospital), Shenzhen, Guangdong 518020, P.R. China Received July 9, 2014; Accepted February 27, 2015 DOI: 10.3892/ijmm.2015.2149 Abstract. Systemic lupus erythematosus (SLE) is a chronic, Introduction potentially fatal systemic autoimmune disease characterized by the production of autoantibodies against a wide range Systemic lupus erythematosus (SLE) is a typical systemic auto- of self-antigens. To investigate the role of the 5-hmC DNA immune disease, involving diffuse connective tissues (1) and modification with regard to the onset of SLE, we compared is characterized by immune inflammation. SLE has a complex the levels 5-hmC between SLE patients and normal controls. pathogenesis (2), involving genetic, immunologic and envi- Whole blood was obtained from patients, and genomic DNA ronmental factors. Thus, it may result in damage to multiple was extracted. Using the hMeDIP-chip analysis and valida- tissues and organs, especially the kidneys (3). SLE arises from tion by quantitative RT-PCR (RT-qPCR), we identified the a combination of heritable and environmental influences. differentially hydroxymethylated regions that are associated Epigenetics, the study of changes in gene expression with SLE. -

Genetic Variation Across the Human Olfactory Receptor Repertoire Alters Odor Perception
bioRxiv preprint doi: https://doi.org/10.1101/212431; this version posted November 1, 2017. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY 4.0 International license. Genetic variation across the human olfactory receptor repertoire alters odor perception Casey Trimmer1,*, Andreas Keller2, Nicolle R. Murphy1, Lindsey L. Snyder1, Jason R. Willer3, Maira Nagai4,5, Nicholas Katsanis3, Leslie B. Vosshall2,6,7, Hiroaki Matsunami4,8, and Joel D. Mainland1,9 1Monell Chemical Senses Center, Philadelphia, Pennsylvania, USA 2Laboratory of Neurogenetics and Behavior, The Rockefeller University, New York, New York, USA 3Center for Human Disease Modeling, Duke University Medical Center, Durham, North Carolina, USA 4Department of Molecular Genetics and Microbiology, Duke University Medical Center, Durham, North Carolina, USA 5Department of Biochemistry, University of Sao Paulo, Sao Paulo, Brazil 6Howard Hughes Medical Institute, New York, New York, USA 7Kavli Neural Systems Institute, New York, New York, USA 8Department of Neurobiology and Duke Institute for Brain Sciences, Duke University Medical Center, Durham, North Carolina, USA 9Department of Neuroscience, University of Pennsylvania School of Medicine, Philadelphia, Pennsylvania, USA *[email protected] ABSTRACT The human olfactory receptor repertoire is characterized by an abundance of genetic variation that affects receptor response, but the perceptual effects of this variation are unclear. To address this issue, we sequenced the OR repertoire in 332 individuals and examined the relationship between genetic variation and 276 olfactory phenotypes, including the perceived intensity and pleasantness of 68 odorants at two concentrations, detection thresholds of three odorants, and general olfactory acuity. -

How Shelterin Protects Mammalian Telomeres 303 ANRV361-GE42-15 ARI 3 October 2008 10:10 (See 3' 5' TRF2 S Mplex
ANRV361-GE42-15 ARI 3 October 2008 10:10 ANNUAL How Shelterin Protects REVIEWS Further Click here for quick links to Annual Reviews content online, Mammalian Telomeres including: • Other articles in this volume 1 • Top cited articles Wilhelm Palm and Titia de Lange • Top downloaded articles • Our comprehensive search Laboratory for Cell Biology and Genetics, The Rockefeller University, New York, NY 10021; email: [email protected] 1Current address: Max Planck Institute of Molecular Cell Biology and Genetics, 01307 Dresden, Germany Annu. Rev. Genet. 2008. 42:301–34 Key Words First published online as a Review in Advance on ATM, ATR, cancer, NHEJ, HR August 4, 2008 by Rockefeller University on 10/13/09. For personal use only. The Annual Review of Genetics is online at Abstract genet.annualreviews.org The genomes of prokaryotes and eukaryotic organelles are usually cir- This article’s doi: cular as are most plasmids and viral genomes. In contrast, the nuclear Annu. Rev. Genet. 2008.42:301-334. Downloaded from arjournals.annualreviews.org 10.1146/annurev.genet.41.110306.130350 genomes of eukaryotes are organized on linear chromosomes, which Copyright c 2008 by Annual Reviews. require mechanisms to protect and replicate DNA ends. Eukaryotes All rights reserved navigate these problemswith the advent of telomeres, protective nucle- 0066-4197/08/1201-0301$20.00 oprotein complexes at the ends of linear chromosomes, and telomerase, the enzyme that maintains the DNA in these structures. Mammalian telomeres contain a specific protein complex, shelterin, that functions to protect chromosome ends from all aspects of the DNA damage re- sponse and regulates telomere maintenance by telomerase. -

Pan-Cancer Analysis of Alternative Lengthening of Telomere Activity
cancers Article Pan-Cancer Analysis of Alternative Lengthening of Telomere Activity Ji-Yong Sung 1,2, Hee-Woong Lim 3, Je-Gun Joung 1,* and Woong-Yang Park 1,2,4,* 1 Samsung Genome Institute, Samsung Medical Center, Seoul 06351, Korea; [email protected] 2 Department of Health Science and Technology, Samsung Advanced Institute of Health Science and Technology, Sungkyunkwan University, Seoul 06351, Korea 3 Division of Biomedical Informatics, Cincinnati Children’s Hospital Medical Center, Cincinnati, OH 45229, USA; [email protected] 4 Department of Molecular Cell Biology, School of Medicine, Sungkyunkwan University, Seoul 06351, Korea * Correspondence: [email protected] (J.-G.J.); [email protected] (W.-Y.P.); Tel.: +82-2-3410-1706 (J.-G.J.); +82-2-3410-6128 (W.-Y.P.); Fax: +82-2-2148-9819 (W.-Y.P.) Received: 30 June 2020; Accepted: 5 August 2020; Published: 7 August 2020 Abstract: Alternative lengthening of telomeres (ALT) is a telomerase-independent mechanism that extends telomeres in cancer cells. It influences tumorigenesis and patient survival. Despite the clinical significance of ALT in tumors, the manner in which ALT is activated and influences prognostic outcomes in distinct cancer types is unclear. In this work, we profiled distinct telomere maintenance mechanisms (TMMs) using 8953 transcriptomes of 31 different cancer types from The Cancer Genome Atlas (TCGA). Our results demonstrated that approximately 29% of cancer types display high ALT activity with low telomerase activity in the telomere-lengthening group. Among the distinct ALT mechanisms, homologous recombination was frequently observed in sarcoma, adrenocortical carcinoma, and kidney chromophobe. Five cancer types showed a significant difference in survival in the presence of high ALT activity. -

Genome-Wide Prediction of Small Molecule Binding to Remote
bioRxiv preprint doi: https://doi.org/10.1101/2020.08.04.236729; this version posted August 5, 2020. The copyright holder for this preprint (which was not certified by peer review) is the author/funder. All rights reserved. No reuse allowed without permission. 1 Genome-wide Prediction of Small Molecule Binding 2 to Remote Orphan Proteins Using Distilled Sequence 3 Alignment Embedding 1 2 3 4 4 Tian Cai , Hansaim Lim , Kyra Alyssa Abbu , Yue Qiu , 5,6 1,2,3,4,7,* 5 Ruth Nussinov , and Lei Xie 1 6 Ph.D. Program in Computer Science, The Graduate Center, The City University of New York, New York, 10016, USA 2 7 Ph.D. Program in Biochemistry, The Graduate Center, The City University of New York, New York, 10016, USA 3 8 Department of Computer Science, Hunter College, The City University of New York, New York, 10065, USA 4 9 Ph.D. Program in Biology, The Graduate Center, The City University of New York, New York, 10016, USA 5 10 Computational Structural Biology Section, Basic Science Program, Frederick National Laboratory for Cancer Research, 11 Frederick, MD 21702, USA 6 12 Department of Human Molecular Genetics and Biochemistry, Sackler School of Medicine, Tel Aviv University, Tel 13 Aviv, Israel 7 14 Helen and Robert Appel Alzheimer’s Disease Research Institute, Feil Family Brain & Mind Research Institute, Weill 15 Cornell Medicine, Cornell University, New York, 10021, USA * 16 [email protected] 17 July 27, 2020 1 bioRxiv preprint doi: https://doi.org/10.1101/2020.08.04.236729; this version posted August 5, 2020. -

Edinburgh Research Explorer
Edinburgh Research Explorer International Union of Basic and Clinical Pharmacology. LXXXVIII. G protein-coupled receptor list Citation for published version: Davenport, AP, Alexander, SPH, Sharman, JL, Pawson, AJ, Benson, HE, Monaghan, AE, Liew, WC, Mpamhanga, CP, Bonner, TI, Neubig, RR, Pin, JP, Spedding, M & Harmar, AJ 2013, 'International Union of Basic and Clinical Pharmacology. LXXXVIII. G protein-coupled receptor list: recommendations for new pairings with cognate ligands', Pharmacological reviews, vol. 65, no. 3, pp. 967-86. https://doi.org/10.1124/pr.112.007179 Digital Object Identifier (DOI): 10.1124/pr.112.007179 Link: Link to publication record in Edinburgh Research Explorer Document Version: Publisher's PDF, also known as Version of record Published In: Pharmacological reviews Publisher Rights Statement: U.S. Government work not protected by U.S. copyright General rights Copyright for the publications made accessible via the Edinburgh Research Explorer is retained by the author(s) and / or other copyright owners and it is a condition of accessing these publications that users recognise and abide by the legal requirements associated with these rights. Take down policy The University of Edinburgh has made every reasonable effort to ensure that Edinburgh Research Explorer content complies with UK legislation. If you believe that the public display of this file breaches copyright please contact [email protected] providing details, and we will remove access to the work immediately and investigate your claim. Download date: 02. Oct. 2021 1521-0081/65/3/967–986$25.00 http://dx.doi.org/10.1124/pr.112.007179 PHARMACOLOGICAL REVIEWS Pharmacol Rev 65:967–986, July 2013 U.S. -

A Computational Approach for Defining a Signature of Β-Cell Golgi Stress in Diabetes Mellitus
Page 1 of 781 Diabetes A Computational Approach for Defining a Signature of β-Cell Golgi Stress in Diabetes Mellitus Robert N. Bone1,6,7, Olufunmilola Oyebamiji2, Sayali Talware2, Sharmila Selvaraj2, Preethi Krishnan3,6, Farooq Syed1,6,7, Huanmei Wu2, Carmella Evans-Molina 1,3,4,5,6,7,8* Departments of 1Pediatrics, 3Medicine, 4Anatomy, Cell Biology & Physiology, 5Biochemistry & Molecular Biology, the 6Center for Diabetes & Metabolic Diseases, and the 7Herman B. Wells Center for Pediatric Research, Indiana University School of Medicine, Indianapolis, IN 46202; 2Department of BioHealth Informatics, Indiana University-Purdue University Indianapolis, Indianapolis, IN, 46202; 8Roudebush VA Medical Center, Indianapolis, IN 46202. *Corresponding Author(s): Carmella Evans-Molina, MD, PhD ([email protected]) Indiana University School of Medicine, 635 Barnhill Drive, MS 2031A, Indianapolis, IN 46202, Telephone: (317) 274-4145, Fax (317) 274-4107 Running Title: Golgi Stress Response in Diabetes Word Count: 4358 Number of Figures: 6 Keywords: Golgi apparatus stress, Islets, β cell, Type 1 diabetes, Type 2 diabetes 1 Diabetes Publish Ahead of Print, published online August 20, 2020 Diabetes Page 2 of 781 ABSTRACT The Golgi apparatus (GA) is an important site of insulin processing and granule maturation, but whether GA organelle dysfunction and GA stress are present in the diabetic β-cell has not been tested. We utilized an informatics-based approach to develop a transcriptional signature of β-cell GA stress using existing RNA sequencing and microarray datasets generated using human islets from donors with diabetes and islets where type 1(T1D) and type 2 diabetes (T2D) had been modeled ex vivo. To narrow our results to GA-specific genes, we applied a filter set of 1,030 genes accepted as GA associated. -

Targeting Lysophosphatidic Acid in Cancer: the Issues in Moving from Bench to Bedside
View metadata, citation and similar papers at core.ac.uk brought to you by CORE provided by IUPUIScholarWorks cancers Review Targeting Lysophosphatidic Acid in Cancer: The Issues in Moving from Bench to Bedside Yan Xu Department of Obstetrics and Gynecology, Indiana University School of Medicine, 950 W. Walnut Street R2-E380, Indianapolis, IN 46202, USA; [email protected]; Tel.: +1-317-274-3972 Received: 28 August 2019; Accepted: 8 October 2019; Published: 10 October 2019 Abstract: Since the clear demonstration of lysophosphatidic acid (LPA)’s pathological roles in cancer in the mid-1990s, more than 1000 papers relating LPA to various types of cancer were published. Through these studies, LPA was established as a target for cancer. Although LPA-related inhibitors entered clinical trials for fibrosis, the concept of targeting LPA is yet to be moved to clinical cancer treatment. The major challenges that we are facing in moving LPA application from bench to bedside include the intrinsic and complicated metabolic, functional, and signaling properties of LPA, as well as technical issues, which are discussed in this review. Potential strategies and perspectives to improve the translational progress are suggested. Despite these challenges, we are optimistic that LPA blockage, particularly in combination with other agents, is on the horizon to be incorporated into clinical applications. Keywords: Autotaxin (ATX); ovarian cancer (OC); cancer stem cell (CSC); electrospray ionization tandem mass spectrometry (ESI-MS/MS); G-protein coupled receptor (GPCR); lipid phosphate phosphatase enzymes (LPPs); lysophosphatidic acid (LPA); phospholipase A2 enzymes (PLA2s); nuclear receptor peroxisome proliferator-activated receptor (PPAR); sphingosine-1 phosphate (S1P) 1. -
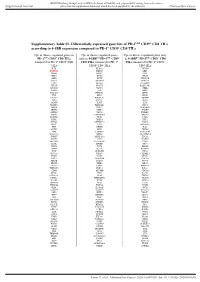
Supplementary Table S5. Differentially Expressed Gene Lists of PD-1High CD39+ CD8 Tils According to 4-1BB Expression Compared to PD-1+ CD39- CD8 Tils
BMJ Publishing Group Limited (BMJ) disclaims all liability and responsibility arising from any reliance Supplemental material placed on this supplemental material which has been supplied by the author(s) J Immunother Cancer Supplementary Table S5. Differentially expressed gene lists of PD-1high CD39+ CD8 TILs according to 4-1BB expression compared to PD-1+ CD39- CD8 TILs Up- or down- regulated genes in Up- or down- regulated genes Up- or down- regulated genes only PD-1high CD39+ CD8 TILs only in 4-1BBneg PD-1high CD39+ in 4-1BBpos PD-1high CD39+ CD8 compared to PD-1+ CD39- CD8 CD8 TILs compared to PD-1+ TILs compared to PD-1+ CD39- TILs CD39- CD8 TILs CD8 TILs IL7R KLRG1 TNFSF4 ENTPD1 DHRS3 LEF1 ITGA5 MKI67 PZP KLF3 RYR2 SIK1B ANK3 LYST PPP1R3B ETV1 ADAM28 H2AC13 CCR7 GFOD1 RASGRP2 ITGAX MAST4 RAD51AP1 MYO1E CLCF1 NEBL S1PR5 VCL MPP7 MS4A6A PHLDB1 GFPT2 TNF RPL3 SPRY4 VCAM1 B4GALT5 TIPARP TNS3 PDCD1 POLQ AKAP5 IL6ST LY9 PLXND1 PLEKHA1 NEU1 DGKH SPRY2 PLEKHG3 IKZF4 MTX3 PARK7 ATP8B4 SYT11 PTGER4 SORL1 RAB11FIP5 BRCA1 MAP4K3 NCR1 CCR4 S1PR1 PDE8A IFIT2 EPHA4 ARHGEF12 PAICS PELI2 LAT2 GPRASP1 TTN RPLP0 IL4I1 AUTS2 RPS3 CDCA3 NHS LONRF2 CDC42EP3 SLCO3A1 RRM2 ADAMTSL4 INPP5F ARHGAP31 ESCO2 ADRB2 CSF1 WDHD1 GOLIM4 CDK5RAP1 CD69 GLUL HJURP SHC4 GNLY TTC9 HELLS DPP4 IL23A PITPNC1 TOX ARHGEF9 EXO1 SLC4A4 CKAP4 CARMIL3 NHSL2 DZIP3 GINS1 FUT8 UBASH3B CDCA5 PDE7B SOGA1 CDC45 NR3C2 TRIB1 KIF14 TRAF5 LIMS1 PPP1R2C TNFRSF9 KLRC2 POLA1 CD80 ATP10D CDCA8 SETD7 IER2 PATL2 CCDC141 CD84 HSPA6 CYB561 MPHOSPH9 CLSPN KLRC1 PTMS SCML4 ZBTB10 CCL3 CA5B PIP5K1B WNT9A CCNH GEM IL18RAP GGH SARDH B3GNT7 C13orf46 SBF2 IKZF3 ZMAT1 TCF7 NECTIN1 H3C7 FOS PAG1 HECA SLC4A10 SLC35G2 PER1 P2RY1 NFKBIA WDR76 PLAUR KDM1A H1-5 TSHZ2 FAM102B HMMR GPR132 CCRL2 PARP8 A2M ST8SIA1 NUF2 IL5RA RBPMS UBE2T USP53 EEF1A1 PLAC8 LGR6 TMEM123 NEK2 SNAP47 PTGIS SH2B3 P2RY8 S100PBP PLEKHA7 CLNK CRIM1 MGAT5 YBX3 TP53INP1 DTL CFH FEZ1 MYB FRMD4B TSPAN5 STIL ITGA2 GOLGA6L10 MYBL2 AHI1 CAND2 GZMB RBPJ PELI1 HSPA1B KCNK5 GOLGA6L9 TICRR TPRG1 UBE2C AURKA Leem G, et al. -

Rabbit Anti-FFAR3/GPR42/FITC Conjugated Antibody-SL16076R
SunLong Biotech Co.,LTD Tel: 0086-571- 56623320 Fax:0086-571- 56623318 E-mail:[email protected] www.sunlongbiotech.com Rabbit Anti-FFAR3/GPR42/FITC Conjugated antibody SL16076R-FITC Product Name: Anti-FFAR3/GPR42/FITC Chinese Name: FITC标记的G protein-coupled receptor42抗体 FFA3R; Ffar3; FFAR3_HUMAN; Free fatty acid receptor 3; G protein coupled Alias: receptor 41; G-protein coupled receptor 41; gpcr41; GPR41; gpr42. Organism Species: Rabbit Clonality: Polyclonal React Species: Human, ICC=1:50-200IF=1:50-200 Applications: not yet tested in other applications. optimal dilutions/concentrations should be determined by the end user. Molecular weight: 39kDa Form: Lyophilized or Liquid Concentration: 1mg/ml immunogen: KLH conjugated synthetic peptide derived from human FFAR3 Lsotype: IgG Purification: affinitywww.sunlongbiotech.com purified by Protein A Storage Buffer: 0.01M TBS(pH7.4) with 1% BSA, 0.03% Proclin300 and 50% Glycerol. Store at -20 °C for one year. Avoid repeated freeze/thaw cycles. The lyophilized antibody is stable at room temperature for at least one month and for greater than a year Storage: when kept at -20°C. When reconstituted in sterile pH 7.4 0.01M PBS or diluent of antibody the antibody is stable for at least two weeks at 2-4 °C. background: G protein-coupled receptors (GPRs), also known as seven transmembrane receptors, heptahelical receptors or 7TM receptors, comprise a superfamily of proteins that play a Product Detail: role in many different stimulus-response pathways. GPRs translate extracellular signals into intracellular signals (a process called G-protein activation) and they respond to a variety of signaling molecules, such as hormones and neurotransmitters. -

G Protein-Coupled Receptors
www.aladdin-e.com Address:800 S Wineville Avenue, Ontario, CA 91761,USA Website:www.aladdin-e.com Email USA: [email protected] Email EU: [email protected] Email Asia Pacific: [email protected] G PROTEIN-COUPLED RECEPTORS Overview: The completion of the Human Genome Project allowed the identification of a large family of proteins with a common motif of seven groups of 20–24 hydrophobic amino acids arranged as a-helices. Approximately 800 of these seven transmembrane (7TM) receptors have been identified of which over 300 are non-olfactory receptors (see Fredriksson et al., 2003; Lagerstrom and Schioth, 2008). Subdivision on the basis of sequence homology allows the definition of rhodopsin, secretin, adhesion, glutamate and Frizzled receptor families. NC-IUPHAR recognizes Classes A, B, and C, which equate to the rhodopsin, secretin, and glutamate receptor families. The nomenclature of 7TM receptors is commonly used interchangeably with G protein-coupled receptors (GPCR), although the former nomenclature recognises signalling of 7TM receptors through pathways not involving G proteins. For example, adiponectin and membrane progestin receptors have some sequence homology to 7TM receptors but signal independently of G proteins and appear to reside in membranes in an inverted fashion compared to conventional GPCR. Additionally, the NPR-C natriuretic peptide receptor (see Page S195) has a single transmembrane domain structure, but appears to couple to G proteins to generate cellular responses. The 300+ non-olfactory GPCR are the targets for the majority of drugs in clinical usage (Overington et al., 2006), although only a minority of these receptors are exploited therapeutically. -

Supplementary Methods
Heterogeneous Contribution of Microdeletions in the Development of Common Generalized and Focal epilepsies. SUPPLEMENTARY METHODS Epilepsy subtype extended description. Genetic Gereralized Epilepsy (GGE): Features unprovoked tonic and/or clonic seizures, originated inconsistently at some focal point within the brain that rapidly generalizes engaging bilateral distributed spikes and waves discharges on the electroencephalogram. This generalization can include cortical and sub cortical structures but not necessarily the entire cortex[1]. GGE is the most common group of epilepsies accounting for 20% of all cases[2]. It is characterized by an age-related onset and a strong familial aggregation and heritability which allows the assumption of a genetic cause. Although genetic associations have been identified, a broad spectrum of causes is acknowledged and remains largely unsolved [3]. Rolandic Epilepsy (RE): Commonly known also as Benign Epilepsy with Centrotemporal Spikes (BECTS), hallmarks early onset diagnosis (mean onset = 7 years old) with brief, focal hemifacial or oropharyngeal sensorimotor seizures alongside speech arrest and secondarily generalized tonic– clonic seizures, which mainly occur during sleep[4]. Rolandic epilepsy features a broad spectrum of less benign related syndromes called atypical Rolandic epilepsy (ARE), including benign partial epilepsy (ABPE), Landau–Kleffner syndrome(LKS) and epileptic encephalopathy with continuous spike-and-waves during sleep (CSWSS)[5]. Together they are the most common childhood epilepsy with a prevalence of 0.2–0.73/1000 (i.e. _1/2500)[6]. Adult Focal Epilepsy (AFE). Focal epilepsy is characterized by sporadic events of seizures originated within a specific brain region and restricted to one hemisphere. Although they can exhibit more than one network of wave discharges on the electroencephalogram, and different degrees of spreading, they feature a consistent site of origin.