Structural-Functional Studies of Mitochondrial Matrix Proteins
Total Page:16
File Type:pdf, Size:1020Kb

Load more
Recommended publications
-

Proteomic Analysis of the Role of the Quality Control Protease LONP1 in Mitochondrial Protein Aggregation
bioRxiv preprint doi: https://doi.org/10.1101/2021.04.12.439502; this version posted April 16, 2021. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. It is made available under aCC-BY-NC-ND 4.0 International license. Proteomic analysis of the role of the quality control protease LONP1 in mitochondrial protein aggregation Karen Pollecker1, Marc Sylvester2 and Wolfgang Voos1,* 1Institute of Biochemistry and Molecular Biology (IBMB), University of Bonn, Faculty of Medicine, Nussallee 11, 53115 Bonn, Germany 2Core facility for mass spectrometry, Institute of Biochemistry and Molecular Biology (IBMB), University of Bonn, Faculty of Medicine, Nussallee 11, 53115 Bonn, Germany *Corresponding author Email: [email protected] Phone: +49-228-732426 Abbreviations: AAA+, ATPases associated with a wide variety of cellular activities; Δψ, mitochondrial membrane potential; gKD, genetic knockdown; HSP, heat shock protein; m, mature form; mt, mitochondrial; p, precursor form; PQC, protein quality control; qMS, quantitative mass spectrometry; ROS, reactive oxygen species; SILAC, stable isotope labeling with amino acids in cell culture; siRNA, small interfering RNA; TIM, preprotein translocase complex of the inner membrane; TMRE, tetramethylrhodamine; TOM, preprotein translocase complex of the outer membrane; UPRmt, mitochondrial unfolded protein response; WT, wild type. bioRxiv preprint doi: https://doi.org/10.1101/2021.04.12.439502; this version posted April 16, 2021. The copyright holder for this preprint (which was not certified by peer review) is the author/funder, who has granted bioRxiv a license to display the preprint in perpetuity. -

TRNT1 Gene Trna Nucleotidyl Transferase 1
TRNT1 gene tRNA nucleotidyl transferase 1 Normal Function The TRNT1 gene provides instructions for making a protein involved in the production ( synthesis) of other proteins. During protein synthesis, a molecule called transfer RNA ( tRNA) helps assemble protein building blocks (amino acids) into a chain that forms the protein. Each tRNA carries a specific amino acid to the growing chain. The TRNT1 protein modifies tRNAs by adding a series of three DNA building blocks (nucleotides), called a CCA trinucleotide, to the molecule. This modification is essential for the correct amino acid to be attached to each tRNA. While most protein synthesis occurs in the fluid surrounding the nucleus (cytoplasm), some proteins are synthesized in cell structures called mitochondria, which are the energy-producing centers in cells. Many mitochondrial proteins form groups (complexes) that carry out the reactions that produce energy. Separate tRNA molecules are used to build proteins in the cytoplasm and mitochondria. The TRNT1 protein attaches the CCA trinucleotide to both cytoplasmic and mitochondrial tRNA molecules. Health Conditions Related to Genetic Changes TRNT1 deficiency More than 20 TRNT1 gene mutations have been found to cause TRNT1 deficiency, a condition with a range of signs and symptoms that affect many body systems. Features can include a blood disorder called sideroblastic anemia, recurrent fevers, a shortage of immune cells called B cells that leads to impairment of the immune system ( immunodeficiency), delayed development of speech and motor skills, and eye abnormalities that cause vision problems. The severity of the condition varies among affected individuals. The TRNT1 gene mutations that cause TRNT1 deficiency lead to a shortage (deficiency) of functional TRNT1 protein. -
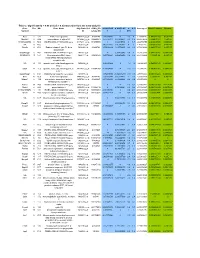
Table 2. Significant
Table 2. Significant (Q < 0.05 and |d | > 0.5) transcripts from the meta-analysis Gene Chr Mb Gene Name Affy ProbeSet cDNA_IDs d HAP/LAP d HAP/LAP d d IS Average d Ztest P values Q-value Symbol ID (study #5) 1 2 STS B2m 2 122 beta-2 microglobulin 1452428_a_at AI848245 1.75334941 4 3.2 4 3.2316485 1.07398E-09 5.69E-08 Man2b1 8 84.4 mannosidase 2, alpha B1 1416340_a_at H4049B01 3.75722111 3.87309653 2.1 1.6 2.84852656 5.32443E-07 1.58E-05 1110032A03Rik 9 50.9 RIKEN cDNA 1110032A03 gene 1417211_a_at H4035E05 4 1.66015788 4 1.7 2.82772795 2.94266E-05 0.000527 NA 9 48.5 --- 1456111_at 3.43701477 1.85785922 4 2 2.8237185 9.97969E-08 3.48E-06 Scn4b 9 45.3 Sodium channel, type IV, beta 1434008_at AI844796 3.79536664 1.63774235 3.3 2.3 2.75319499 1.48057E-08 6.21E-07 polypeptide Gadd45gip1 8 84.1 RIKEN cDNA 2310040G17 gene 1417619_at 4 3.38875643 1.4 2 2.69163229 8.84279E-06 0.0001904 BC056474 15 12.1 Mus musculus cDNA clone 1424117_at H3030A06 3.95752801 2.42838452 1.9 2.2 2.62132809 1.3344E-08 5.66E-07 MGC:67360 IMAGE:6823629, complete cds NA 4 153 guanine nucleotide binding protein, 1454696_at -3.46081884 -4 -1.3 -1.6 -2.6026947 8.58458E-05 0.0012617 beta 1 Gnb1 4 153 guanine nucleotide binding protein, 1417432_a_at H3094D02 -3.13334396 -4 -1.6 -1.7 -2.5946297 1.04542E-05 0.0002202 beta 1 Gadd45gip1 8 84.1 RAD23a homolog (S. -

A Computational Approach for Defining a Signature of Β-Cell Golgi Stress in Diabetes Mellitus
Page 1 of 781 Diabetes A Computational Approach for Defining a Signature of β-Cell Golgi Stress in Diabetes Mellitus Robert N. Bone1,6,7, Olufunmilola Oyebamiji2, Sayali Talware2, Sharmila Selvaraj2, Preethi Krishnan3,6, Farooq Syed1,6,7, Huanmei Wu2, Carmella Evans-Molina 1,3,4,5,6,7,8* Departments of 1Pediatrics, 3Medicine, 4Anatomy, Cell Biology & Physiology, 5Biochemistry & Molecular Biology, the 6Center for Diabetes & Metabolic Diseases, and the 7Herman B. Wells Center for Pediatric Research, Indiana University School of Medicine, Indianapolis, IN 46202; 2Department of BioHealth Informatics, Indiana University-Purdue University Indianapolis, Indianapolis, IN, 46202; 8Roudebush VA Medical Center, Indianapolis, IN 46202. *Corresponding Author(s): Carmella Evans-Molina, MD, PhD ([email protected]) Indiana University School of Medicine, 635 Barnhill Drive, MS 2031A, Indianapolis, IN 46202, Telephone: (317) 274-4145, Fax (317) 274-4107 Running Title: Golgi Stress Response in Diabetes Word Count: 4358 Number of Figures: 6 Keywords: Golgi apparatus stress, Islets, β cell, Type 1 diabetes, Type 2 diabetes 1 Diabetes Publish Ahead of Print, published online August 20, 2020 Diabetes Page 2 of 781 ABSTRACT The Golgi apparatus (GA) is an important site of insulin processing and granule maturation, but whether GA organelle dysfunction and GA stress are present in the diabetic β-cell has not been tested. We utilized an informatics-based approach to develop a transcriptional signature of β-cell GA stress using existing RNA sequencing and microarray datasets generated using human islets from donors with diabetes and islets where type 1(T1D) and type 2 diabetes (T2D) had been modeled ex vivo. To narrow our results to GA-specific genes, we applied a filter set of 1,030 genes accepted as GA associated. -
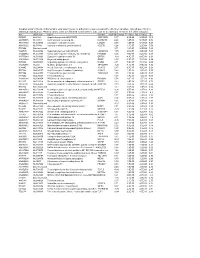
Supplementary File 2A Revised
Supplementary file 2A. Differentially expressed genes in aldosteronomas compared to all other samples, ranked according to statistical significance. Missing values were not allowed in aldosteronomas, but to a maximum of five in the other samples. Acc UGCluster Name Symbol log Fold Change P - Value Adj. P-Value B R99527 Hs.8162 Hypothetical protein MGC39372 MGC39372 2,17 6,3E-09 5,1E-05 10,2 AA398335 Hs.10414 Kelch domain containing 8A KLHDC8A 2,26 1,2E-08 5,1E-05 9,56 AA441933 Hs.519075 Leiomodin 1 (smooth muscle) LMOD1 2,33 1,3E-08 5,1E-05 9,54 AA630120 Hs.78781 Vascular endothelial growth factor B VEGFB 1,24 1,1E-07 2,9E-04 7,59 R07846 Data not found 3,71 1,2E-07 2,9E-04 7,49 W92795 Hs.434386 Hypothetical protein LOC201229 LOC201229 1,55 2,0E-07 4,0E-04 7,03 AA454564 Hs.323396 Family with sequence similarity 54, member B FAM54B 1,25 3,0E-07 5,2E-04 6,65 AA775249 Hs.513633 G protein-coupled receptor 56 GPR56 -1,63 4,3E-07 6,4E-04 6,33 AA012822 Hs.713814 Oxysterol bining protein OSBP 1,35 5,3E-07 7,1E-04 6,14 R45592 Hs.655271 Regulating synaptic membrane exocytosis 2 RIMS2 2,51 5,9E-07 7,1E-04 6,04 AA282936 Hs.240 M-phase phosphoprotein 1 MPHOSPH -1,40 8,1E-07 8,9E-04 5,74 N34945 Hs.234898 Acetyl-Coenzyme A carboxylase beta ACACB 0,87 9,7E-07 9,8E-04 5,58 R07322 Hs.464137 Acyl-Coenzyme A oxidase 1, palmitoyl ACOX1 0,82 1,3E-06 1,2E-03 5,35 R77144 Hs.488835 Transmembrane protein 120A TMEM120A 1,55 1,7E-06 1,4E-03 5,07 H68542 Hs.420009 Transcribed locus 1,07 1,7E-06 1,4E-03 5,06 AA410184 Hs.696454 PBX/knotted 1 homeobox 2 PKNOX2 1,78 2,0E-06 -

Supplementary Table S4. FGA Co-Expressed Gene List in LUAD
Supplementary Table S4. FGA co-expressed gene list in LUAD tumors Symbol R Locus Description FGG 0.919 4q28 fibrinogen gamma chain FGL1 0.635 8p22 fibrinogen-like 1 SLC7A2 0.536 8p22 solute carrier family 7 (cationic amino acid transporter, y+ system), member 2 DUSP4 0.521 8p12-p11 dual specificity phosphatase 4 HAL 0.51 12q22-q24.1histidine ammonia-lyase PDE4D 0.499 5q12 phosphodiesterase 4D, cAMP-specific FURIN 0.497 15q26.1 furin (paired basic amino acid cleaving enzyme) CPS1 0.49 2q35 carbamoyl-phosphate synthase 1, mitochondrial TESC 0.478 12q24.22 tescalcin INHA 0.465 2q35 inhibin, alpha S100P 0.461 4p16 S100 calcium binding protein P VPS37A 0.447 8p22 vacuolar protein sorting 37 homolog A (S. cerevisiae) SLC16A14 0.447 2q36.3 solute carrier family 16, member 14 PPARGC1A 0.443 4p15.1 peroxisome proliferator-activated receptor gamma, coactivator 1 alpha SIK1 0.435 21q22.3 salt-inducible kinase 1 IRS2 0.434 13q34 insulin receptor substrate 2 RND1 0.433 12q12 Rho family GTPase 1 HGD 0.433 3q13.33 homogentisate 1,2-dioxygenase PTP4A1 0.432 6q12 protein tyrosine phosphatase type IVA, member 1 C8orf4 0.428 8p11.2 chromosome 8 open reading frame 4 DDC 0.427 7p12.2 dopa decarboxylase (aromatic L-amino acid decarboxylase) TACC2 0.427 10q26 transforming, acidic coiled-coil containing protein 2 MUC13 0.422 3q21.2 mucin 13, cell surface associated C5 0.412 9q33-q34 complement component 5 NR4A2 0.412 2q22-q23 nuclear receptor subfamily 4, group A, member 2 EYS 0.411 6q12 eyes shut homolog (Drosophila) GPX2 0.406 14q24.1 glutathione peroxidase -

The KMT1A-GATA3-STAT3 Circuit Is a Novel Self-Renewal Signaling of Human Bladder Cancer Stem Cells Zhao Yang1, Luyun He2,3, Kais
The KMT1A-GATA3-STAT3 circuit is a novel self-renewal signaling of human bladder cancer stem cells Zhao Yang1, Luyun He2,3, Kaisu Lin4, Yun Zhang1, Aihua Deng1, Yong Liang1, Chong Li2, 5, & Tingyi Wen1, 6, 1CAS Key Laboratory of Pathogenic Microbiology and Immunology, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China 2Core Facility for Protein Research, Institute of Biophysics, Chinese Academy of Sciences, Beijing 100101, China 3CAS Key Laboratory of Infection and Immunity, Institute of Biophysics, Chinese Academy of Sciences, Beijing 100101, China 4Department of Oncology, the Second Affiliated Hospital of Soochow University, Suzhou 215000, China 5Beijing Jianlan Institute of Medicine, Beijing 100190, China 6Savaid Medical School, University of Chinese Academy of Sciences, Beijing 100049, China Correspondence author: Tingyi Wen, e-mail: [email protected] Chong Li, e-mail: [email protected] Supplementary Figure S1. Isolation of human bladder cancer stem cells. BCMab1 and CD44 were used to isolate bladder cancer stem cells (BCSCs: BCMab1+CD44+) and bladder cancer non-stem cells (BCNSCs: BCMab1-CD44-) from EJ, samples #1 and #2 by flow cytometry. Supplementary Figure S2. Gene ontology analysis of downregulated genes of human BCSCs. (A) Pathway enrichment of 103 downregulated genes in BCSCs. (B) The seven downregulated genes in BCSCs participating in centromeric heterochromatin, mRNA-3’-UTR binding and translation regulator activity signaling pathways were validated by qRT-PCR. Data are presented as mean ± SD. P < 0.05; P < 0.01. Supplementary Figure S3. The expression of KMT1A is higher in human BC than that in peri-tumor tissues. (A) The expression of KMT1A was higher in BC samples than that in peri-tumors as assessed by immunohistochemistry, Scale bar = 50 m. -

Mitochondrial Lon Protease in Human Disease and Aging: Including an Etiologic Classification of Lon-Related Diseases and Disorders
UC Irvine UC Irvine Previously Published Works Title Mitochondrial Lon protease in human disease and aging: Including an etiologic classification of Lon-related diseases and disorders. Permalink https://escholarship.org/uc/item/2pw606qk Authors Bota, Daniela A Davies, Kelvin JA Publication Date 2016-11-01 DOI 10.1016/j.freeradbiomed.2016.06.031 Peer reviewed eScholarship.org Powered by the California Digital Library University of California HHS Public Access Author manuscript Author ManuscriptAuthor Manuscript Author Free Radic Manuscript Author Biol Med. Author Manuscript Author manuscript; available in PMC 2016 December 24. Published in final edited form as: Free Radic Biol Med. 2016 November ; 100: 188–198. doi:10.1016/j.freeradbiomed.2016.06.031. Mitochondrial Lon protease in human disease and aging: Including an etiologic classification of Lon-related diseases and disorders Daniela A. Botaa,* and Kelvin J.A. Daviesb,c a Department of Neurology and Chao Family Comprehensive Cancer Center, UC Irvine School of Medicine, 200 S. Manchester Ave., Suite 206, Orange, CA 92868, USA b Leonard Davis School of Gerontology of the Ethel Percy Andrus Gerontology Center, Los Angeles, CA 90089-0191, USA c Division of Molecular & Computational Biology, Department of Biological Sciences, Dornsife College of Letters, Arts, & Sciences, The University of Southern California, Los Angeles, CA 90089-0191, USA Abstract The Mitochondrial Lon protease, also called LonP1 is a product of the nuclear gene LONP1. Lon is a major regulator of mitochondrial metabolism and response to free radical damage, as well as an essential factor for the maintenance and repair of mitochondrial DNA. Lon is an ATP- stimulated protease that cycles between being bound (at the inner surface of the inner mitochondrial membrane) to the mitochondrial genome, and being released into the mitochondrial matrix where it can degrade matrix proteins. -

Human Induced Pluripotent Stem Cell–Derived Podocytes Mature Into Vascularized Glomeruli Upon Experimental Transplantation
BASIC RESEARCH www.jasn.org Human Induced Pluripotent Stem Cell–Derived Podocytes Mature into Vascularized Glomeruli upon Experimental Transplantation † Sazia Sharmin,* Atsuhiro Taguchi,* Yusuke Kaku,* Yasuhiro Yoshimura,* Tomoko Ohmori,* ‡ † ‡ Tetsushi Sakuma, Masashi Mukoyama, Takashi Yamamoto, Hidetake Kurihara,§ and | Ryuichi Nishinakamura* *Department of Kidney Development, Institute of Molecular Embryology and Genetics, and †Department of Nephrology, Faculty of Life Sciences, Kumamoto University, Kumamoto, Japan; ‡Department of Mathematical and Life Sciences, Graduate School of Science, Hiroshima University, Hiroshima, Japan; §Division of Anatomy, Juntendo University School of Medicine, Tokyo, Japan; and |Japan Science and Technology Agency, CREST, Kumamoto, Japan ABSTRACT Glomerular podocytes express proteins, such as nephrin, that constitute the slit diaphragm, thereby contributing to the filtration process in the kidney. Glomerular development has been analyzed mainly in mice, whereas analysis of human kidney development has been minimal because of limited access to embryonic kidneys. We previously reported the induction of three-dimensional primordial glomeruli from human induced pluripotent stem (iPS) cells. Here, using transcription activator–like effector nuclease-mediated homologous recombination, we generated human iPS cell lines that express green fluorescent protein (GFP) in the NPHS1 locus, which encodes nephrin, and we show that GFP expression facilitated accurate visualization of nephrin-positive podocyte formation in -

Protection of Scaffold Protein Isu from Degradation by the Lon Protease Pim1 As a Component of Fe–S Cluster Biogenesis Regulation
M BoC | ARTICLE Protection of scaffold protein Isu from degradation by the Lon protease Pim1 as a component of Fe–S cluster biogenesis regulation Szymon J. Ciesielskia, Brenda Schilkea, Jaroslaw Marszaleka,b, and Elizabeth A. Craiga,* aDepartment of Biochemistry, University of Wisconsin–Madison, Madison, WI 53706; bIntercollegiate Faculty of Biotechnology, University of Gdansk and Medical University of Gdansk, Gdansk 80307, Poland ABSTRACT Iron–sulfur (Fe–S) clusters, essential protein cofactors, are assembled on the mi- Monitoring Editor tochondrial scaffold protein Isu and then transferred to recipient proteins via a multistep Thomas D. Fox process in which Isu interacts sequentially with multiple protein factors. This pathway is in Cornell University part regulated posttranslationally by modulation of the degradation of Isu, whose abundance Received: Dec 4, 2015 increases >10-fold upon perturbation of the biogenesis process. We tested a model in which Revised: Jan 22, 2016 direct interaction with protein partners protects Isu from degradation by the mitochondrial Accepted: Jan 25, 2016 Lon-type protease. Using purified components, we demonstrated that Isu is indeed a sub- strate of the Lon-type protease and that it is protected from degradation by Nfs1, the sulfur donor for Fe–S cluster assembly, as well as by Jac1, the J-protein Hsp70 cochaperone that functions in cluster transfer from Isu. Nfs1 and Jac1 variants known to be defective in interac- tion with Isu were also defective in protecting Isu from degradation. Furthermore, overpro- duction of Jac1 protected Isu from degradation in vivo, as did Nfs1. Taken together, our re- sults lead to a model of dynamic interplay between a protease and protein factors throughout the Fe–S cluster assembly and transfer process, leading to up-regulation of Isu levels under conditions when Fe–S cluster biogenesis does not meet cellular demands. -

Phosphorylation of Human TFAM in Mitochondria Impairs DNA Binding and Promotes Degradation by the AAA+ Lon Protease
Molecular Cell Article Phosphorylation of Human TFAM in Mitochondria Impairs DNA Binding and Promotes Degradation by the AAA+ Lon Protease Bin Lu,1,4,5 Jae Lee,1,5 Xiaobo Nie,1,4,5 Min Li,1 Yaroslav I. Morozov,2 Sundararajan Venkatesh,1 Daniel F. Bogenhagen,3 Dmitry Temiakov,2 and Carolyn K. Suzuki1,* 1Department of Biochemistry and Molecular Biology, New Jersey Medical School, University of Medicine and Dentistry of New Jersey, Newark, NJ 07103, USA 2Department of Cell Biology, School of Osteopathic Medicine, University of Medicine and Dentistry of New Jersey, Stratford, NJ 08084, USA 3Department of Pharmacological Sciences, State University of New York at Stony Brook, Stony Brook, NY 11794, USA 4Present address: Institute of Biophysics, School of Laboratory Medicine and Life Sciences, Wenzhou Medical College, Wenzhou, Zhejiang 325035, PRC 5These authors contributed equally to this work *Correspondence: [email protected] http://dx.doi.org/10.1016/j.molcel.2012.10.023 SUMMARY contrast, TFAM overproduction in transgenic mice increases mtDNA content (Ekstrand et al., 2004; Larsson et al., 1998) Human mitochondrial transcription factor A (TFAM) and also ameliorates cardiac failure (Ikeuchi et al., 2005), neuro- is a high-mobility group (HMG) protein at the nexus degeneration, and age-dependent deficits in brain function of mitochondrial DNA (mtDNA) replication, transcrip- (Hokari et al., 2010). TFAM is the most abundant component tion, and inheritance. Little is known about the mech- of mitochondrial nucleoids, which are protein complexes anisms underlying its posttranslational regulation. associated with mtDNA that orchestrate genome replication, Here, we demonstrate that TFAM is phosphorylated expression, and inheritance (Bogenhagen et al., 2008, 2012; Kukat et al., 2011). -

Protein Symbol Protein Name Rank Metric Score 4F2 4F2 Cell-Surface
Supplementary Table 2 Supplementary Table 2. Ranked list of proteins present in anti-Sema4D treated macrophage conditioned media obtained in the GSEA analysis of the proteomic data. Proteins are listed according to their rank metric score, which is the score used to position the gene in the ranked list of genes of the GSEA. Values are obtained from comparing Sema4D treated RAW conditioned media versus REST, which includes untreated, IgG treated and anti-Sema4D added RAW conditioned media. GSEA analysis was performed under standard conditions in November 2015. Protein Rank metric symbol Protein name score 4F2 4F2 cell-surface antigen heavy chain 2.5000 PLOD3 Procollagen-lysine,2-oxoglutarate 5-dioxygenase 3 1.4815 ELOB Transcription elongation factor B polypeptide 2 1.4350 ARPC5 Actin-related protein 2/3 complex subunit 5 1.2603 OSTF1 teoclast-stimulating factor 1 1.2500 RL5 60S ribomal protein L5 1.2135 SYK Lysine--tRNA ligase 1.2135 RL10A 60S ribomal protein L10a 1.2135 TXNL1 Thioredoxin-like protein 1 1.1716 LIS1 Platelet-activating factor acetylhydrolase IB subunit alpha 1.1067 A4 Amyloid beta A4 protein 1.0911 H2B1M Histone H2B type 1-M 1.0514 UB2V2 Ubiquitin-conjugating enzyme E2 variant 2 1.0381 PDCD5 Programmed cell death protein 5 1.0373 UCHL3 Ubiquitin carboxyl-terminal hydrolase isozyme L3 1.0061 PLEC Plectin 1.0061 ITPA Inine triphphate pyrophphatase 0.9524 IF5A1 Eukaryotic translation initiation factor 5A-1 0.9314 ARP2 Actin-related protein 2 0.8618 HNRPL Heterogeneous nuclear ribonucleoprotein L 0.8576 DNJA3 DnaJ homolog subfamily