“Investigaç Compostos a “Genetic an of Antimi
Total Page:16
File Type:pdf, Size:1020Kb
Load more
Recommended publications
-

Genomic Insight Into the Host–Endosymbiont Relationship of Endozoicomonas Montiporae CL-33T with Its Coral Host
ORIGINAL RESEARCH published: 08 March 2016 doi: 10.3389/fmicb.2016.00251 Genomic Insight into the Host–Endosymbiont Relationship of Endozoicomonas montiporae CL-33T with its Coral Host Jiun-Yan Ding 1, Jia-Ho Shiu 1, Wen-Ming Chen 2, Yin-Ru Chiang 1 and Sen-Lin Tang 1* 1 Biodiversity Research Center, Academia Sinica, Taipei, Taiwan, 2 Department of Seafood Science, Laboratory of Microbiology, National Kaohsiung Marine University, Kaohsiung, Taiwan The bacterial genus Endozoicomonas was commonly detected in healthy corals in many coral-associated bacteria studies in the past decade. Although, it is likely to be a core member of coral microbiota, little is known about its ecological roles. To decipher potential interactions between bacteria and their coral hosts, we sequenced and investigated the first culturable endozoicomonal bacterium from coral, the E. montiporae CL-33T. Its genome had potential sign of ongoing genome erosion and gene exchange with its Edited by: Rekha Seshadri, host. Testosterone degradation and type III secretion system are commonly present in Department of Energy Joint Genome Endozoicomonas and may have roles to recognize and deliver effectors to their hosts. Institute, USA Moreover, genes of eukaryotic ephrin ligand B2 are present in its genome; presumably, Reviewed by: this bacterium could move into coral cells via endocytosis after binding to coral’s Eph Kathleen M. Morrow, University of New Hampshire, USA receptors. In addition, 7,8-dihydro-8-oxoguanine triphosphatase and isocitrate lyase Jean-Baptiste Raina, are possible type III secretion effectors that might help coral to prevent mitochondrial University of Technology Sydney, Australia dysfunction and promote gluconeogenesis, especially under stress conditions. -

Motiliproteus Sediminis Gen. Nov., Sp. Nov., Isolated from Coastal Sediment
Antonie van Leeuwenhoek (2014) 106:615–621 DOI 10.1007/s10482-014-0232-2 ORIGINAL PAPER Motiliproteus sediminis gen. nov., sp. nov., isolated from coastal sediment Zong-Jie Wang • Zhi-Hong Xie • Chao Wang • Zong-Jun Du • Guan-Jun Chen Received: 3 April 2014 / Accepted: 4 July 2014 / Published online: 20 July 2014 Ó Springer International Publishing Switzerland 2014 Abstract A novel Gram-stain-negative, rod-to- demonstrated that the novel isolate was 93.3 % similar spiral-shaped, oxidase- and catalase- positive and to the type strain of Neptunomonas antarctica, 93.2 % facultatively aerobic bacterium, designated HS6T, was to Neptunomonas japonicum and 93.1 % to Marino- isolated from marine sediment of Yellow Sea, China. bacterium rhizophilum, the closest cultivated rela- It can reduce nitrate to nitrite and grow well in marine tives. The polar lipid profile of the novel strain broth 2216 (MB, Hope Biol-Technology Co., Ltd) consisted of phosphatidylethanolamine, phosphatidyl- with an optimal temperature for growth of 30–33 °C glycerol and some other unknown lipids. Major (range 12–45 °C) and in the presence of 2–3 % (w/v) cellular fatty acids were summed feature 3 (C16:1 NaCl (range 0.5–7 %, w/v). The pH range for growth x7c/iso-C15:0 2-OH), C18:1 x7c and C16:0 and the main was pH 6.2–9.0, with an optimum at 6.5–7.0. Phylo- respiratory quinone was Q-8. The DNA G?C content genetic analysis based on 16S rRNA gene sequences of strain HS6T was 61.2 mol %. Based on the phylogenetic, physiological and biochemical charac- teristics, strain HS6T represents a novel genus and The GenBank accession number for the 16S rRNA gene T species and the name Motiliproteus sediminis gen. -

Oil Type and Temperature Dependent Biodegradation Dynamics
Ribicic et al. BMC Microbiology (2018) 18:83 https://doi.org/10.1186/s12866-018-1221-9 RESEARCH ARTICLE Open Access Oil type and temperature dependent biodegradation dynamics - Combining chemical and microbial community data through multivariate analysis Deni Ribicic1,2* , Kelly Marie McFarlin1, Roman Netzer1, Odd Gunnar Brakstad1, Anika Winkler3, Mimmi Throne-Holst1 and Trond Røvik Størseth1 Abstract Background: This study investigates a comparative multivariate approach for studying the biodegradation of chemically dispersed oil. The rationale for this approach lies in the inherent complexity of the data and challenges associated with comparing multiple experiments with inconsistent sampling points, with respect to inferring correlations and visualizing multiple datasets with numerous variables. We aim to identify novel correlations among microbial community composition, the chemical change of individual petroleum hydrocarbons, oil type and temperature by creating modelled datasets from inconsistent sampling time points. Four different incubation experiments were conducted with freshly collected Norwegian seawater and either Grane and Troll oil dispersed with Corexit 9500. Incubations were conducted at two different temperatures (5 °C and 13 °C) over a period of 64 days. Results: PCA analysis of modelled chemical datasets and calculated half-lives revealed differences in the biodegradation of individual hydrocarbons among temperatures and oil types. At 5 °C, most n-alkanes biodegraded faster in heavy Grane oil compared to light Troll oil. PCA analysis of modelled microbial community datasets reveal differences between temperature and oil type, especially at low temperature. For both oils, Colwelliaceae and Oceanospirillaceae were more prominent in the colder incubation (5 °C) than the warmer (13 °C). Overall, Colwelliaceae, Oceanospirillaceae, Flavobacteriaceae, Rhodobacteraceae, Alteromonadaceae and Piscirickettsiaceae consistently dominated the microbial community at both temperatures and in both oil types. -

Comparative Proteomic Profiling of Newly Acquired, Virulent And
www.nature.com/scientificreports OPEN Comparative proteomic profling of newly acquired, virulent and attenuated Neoparamoeba perurans proteins associated with amoebic gill disease Kerrie Ní Dhufaigh1*, Eugene Dillon2, Natasha Botwright3, Anita Talbot1, Ian O’Connor1, Eugene MacCarthy1 & Orla Slattery4 The causative agent of amoebic gill disease, Neoparamoeba perurans is reported to lose virulence during prolonged in vitro maintenance. In this study, the impact of prolonged culture on N. perurans virulence and its proteome was investigated. Two isolates, attenuated and virulent, had their virulence assessed in an experimental trial using Atlantic salmon smolts and their bacterial community composition was evaluated by 16S rRNA Illumina MiSeq sequencing. Soluble proteins were isolated from three isolates: a newly acquired, virulent and attenuated N. perurans culture. Proteins were analysed using two-dimensional electrophoresis coupled with liquid chromatography tandem mass spectrometry (LC–MS/MS). The challenge trial using naïve smolts confrmed a loss in virulence in the attenuated N. perurans culture. A greater diversity of bacterial communities was found in the microbiome of the virulent isolate in contrast to a reduction in microbial community richness in the attenuated microbiome. A collated proteome database of N. perurans, Amoebozoa and four bacterial genera resulted in 24 proteins diferentially expressed between the three cultures. The present LC–MS/ MS results indicate protein synthesis, oxidative stress and immunomodulation are upregulated in a newly acquired N. perurans culture and future studies may exploit these protein identifcations for therapeutic purposes in infected farmed fsh. Neoparamoeba perurans is an ectoparasitic protozoan responsible for the hyperplastic gill infection of marine cultured fnfsh referred to as amoebic gill disease (AGD)1. -

The Gut Microbiome of the Sea Urchin, Lytechinus Variegatus, from Its Natural Habitat Demonstrates Selective Attributes of Micro
FEMS Microbiology Ecology, 92, 2016, fiw146 doi: 10.1093/femsec/fiw146 Advance Access Publication Date: 1 July 2016 Research Article RESEARCH ARTICLE The gut microbiome of the sea urchin, Lytechinus variegatus, from its natural habitat demonstrates selective attributes of microbial taxa and predictive metabolic profiles Joseph A. Hakim1,†, Hyunmin Koo1,†, Ranjit Kumar2, Elliot J. Lefkowitz2,3, Casey D. Morrow4, Mickie L. Powell1, Stephen A. Watts1,∗ and Asim K. Bej1,∗ 1Department of Biology, University of Alabama at Birmingham, 1300 University Blvd, Birmingham, AL 35294, USA, 2Center for Clinical and Translational Sciences, University of Alabama at Birmingham, Birmingham, AL 35294, USA, 3Department of Microbiology, University of Alabama at Birmingham, Birmingham, AL 35294, USA and 4Department of Cell, Developmental and Integrative Biology, University of Alabama at Birmingham, 1918 University Blvd., Birmingham, AL 35294, USA ∗Corresponding authors: Department of Biology, University of Alabama at Birmingham, 1300 University Blvd, CH464, Birmingham, AL 35294-1170, USA. Tel: +1-(205)-934-8308; Fax: +1-(205)-975-6097; E-mail: [email protected]; [email protected] †These authors contributed equally to this work. One sentence summary: This study describes the distribution of microbiota, and their predicted functional attributes, in the gut ecosystem of sea urchin, Lytechinus variegatus, from its natural habitat of Gulf of Mexico. Editor: Julian Marchesi ABSTRACT In this paper, we describe the microbial composition and their predictive metabolic profile in the sea urchin Lytechinus variegatus gut ecosystem along with samples from its habitat by using NextGen amplicon sequencing and downstream bioinformatics analyses. The microbial communities of the gut tissue revealed a near-exclusive abundance of Campylobacteraceae, whereas the pharynx tissue consisted of Tenericutes, followed by Gamma-, Alpha- and Epsilonproteobacteria at approximately equal capacities. -
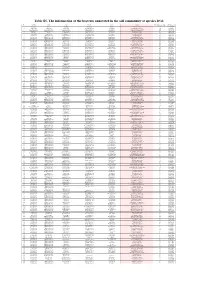
Table S5. the Information of the Bacteria Annotated in the Soil Community at Species Level
Table S5. The information of the bacteria annotated in the soil community at species level No. Phylum Class Order Family Genus Species The number of contigs Abundance(%) 1 Firmicutes Bacilli Bacillales Bacillaceae Bacillus Bacillus cereus 1749 5.145782459 2 Bacteroidetes Cytophagia Cytophagales Hymenobacteraceae Hymenobacter Hymenobacter sedentarius 1538 4.52499338 3 Gemmatimonadetes Gemmatimonadetes Gemmatimonadales Gemmatimonadaceae Gemmatirosa Gemmatirosa kalamazoonesis 1020 3.000970902 4 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas indica 797 2.344876284 5 Firmicutes Bacilli Lactobacillales Streptococcaceae Lactococcus Lactococcus piscium 542 1.594633558 6 Actinobacteria Thermoleophilia Solirubrobacterales Conexibacteraceae Conexibacter Conexibacter woesei 471 1.385742446 7 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas taxi 430 1.265115184 8 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas wittichii 388 1.141545794 9 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas sp. FARSPH 298 0.876754244 10 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sorangium cellulosum 260 0.764953367 11 Proteobacteria Deltaproteobacteria Myxococcales Polyangiaceae Sorangium Sphingomonas sp. Cra20 260 0.764953367 12 Proteobacteria Alphaproteobacteria Sphingomonadales Sphingomonadaceae Sphingomonas Sphingomonas panacis 252 0.741416341 -

Exploring Antibiotic Resistance Genes (Args) Abundance and Distribution on TARA Oceans Samples Through Machine Learning Tools Rafael R
bioRxiv preprint doi: https://doi.org/10.1101/765446. this version posted December 21, 2019. The copyright holder for this preprint (which was not certified by peer review) is the author/funder. It is made available under a CC-BY 4.0 International license. Global ocean resistome revealed: exploring Antibiotic Resistance Genes (ARGs) abundance and distribution on TARA oceans samples through machine learning tools Rafael R. C. Cuadrat1, Maria Sorokina2, Bruno G. Andrade3, Tobias Goris4, Alberto M. R. Dávila5 1 - Molecular Epidemiology Department, German Institute of Human Nutrition Potsdam-Rehbruecke - DIfE 2 - Friedrich-Schiller University, Lessingstrasse 8, 07743 Jena, Germany, ORCID: 0000-0001-9359-7149 3 - Embrapa Southeast Livestock - EMBRAPA 4 - Department of Molecular Toxicology, Research Group Intestinal Microbiology, German Institute of Human Nutrition Potsdam-Rehbruecke - DIfE 5 - Computational and Systems Biology Laboratory, Oswaldo Cruz Institute, FIOCRUZ. Av Brasil 4365, Rio de Janeiro, RJ, Brasil. 21040-900. Abstract The rise of antibiotic resistance (AR) in clinical settings is one of the biggest modern global public health concerns. Therefore, the understanding of AR mechanisms, evolution and global distribution is a priority due to its impact on the treatment course and patient survivability. Besides all efforts in the elucidation of AR mechanisms in clinical strains, little is known about its prevalence and evolution in environmental uncultivable microorganisms. In this study, 293 metagenomic from the TARA Oceans project were used to detect and quantify environmental antibiotic resistance genes (ARGs) using machine learning tools. After extensive manual curation, we show the global ocean ARG abundance, distribution, taxonomy, phylogeny and their potential to be horizontally transferred by plasmids or viruses and their correlation with environmental and geographical parameters. -

87-Fernanda-Cid.Pdf
UNIVERSIDAD DE LA FRONTERA Facultad de Ingeniería y Ciencias Doctorado en Ciencias de Recursos Naturales Characterization of bacterial communities from Deschampsia antarctica phyllosphere and genome sequencing of culturable bacteria with ice recrystallization inhibition activity DOCTORAL THESIS IN FULFILLMENT OF THE REQUERIMENTS FOR THE DEGREE DOCTOR OF SCIENCES IN NATURAL RESOURCES FERNANDA DEL PILAR CID ALDA TEMUCO-CHILE 2018 “Characterization of bacterial communities from Deschampsia antarctica phyllosphere and genome sequencing of culturable bacteria with ice recrystallization inhibition activity” Esta tesis fue realizada bajo la supervisión del Director de tesis, Dr. Milko A. Jorquera Tapia, profesor asociado del Departamento de Ciencias Químicas y Recursos Naturales, Facultad de Ingeniería y Ciencias, Universidad de La Frontera y co-dirigida por el Dr. León A. Bravo Ramírez, profesor titular del Departamento de Ciencias Agronómicas y Recursos Naturales, Facultad de Ciencias Agropecuarias y Forestales, Universidad de La Frontera. Esta tesis ha sido además aprobada por la comisión examinadora. Fernanda del Pilar Cid Alda …………………………………………… ……………………………………. Dr. MILKO JORQUERA T. Dr. Francisco Matus DIRECTOR DEL PROGRAMA DE …………………………………………. DOCTORADO EN CIENCIAS DE Dr. LEON BRAVO R. RECURSOS NATURALES …………………………………………. Dra. MARYSOL ALVEAR Z. ............................................................ Dr. Juan Carlos Parra ………………………………………… DIRECTOR DE POSTGRADO Dra. PAULINA BULL S. UNIVERSIDAD DE LA FRONTERA ………………………………………… Dr. LUIS COLLADO G. ………………………………………… Dra. ANA MUTIS T Dedico esta tesis a mis padres, Lillie Alda Leiva y Fernando Cid Verdugo quienes dedicaron sus vidas a la tarea de enseñar en la Universidad de La Frontera, Temuco-Chile. Abril, 2018 Agradecimientos /Acknowledgements Agradecimientos /Acknowledgements This study was supported by the following research projects: Doctoral Scholarship from Conicyt (no. 21140534) and La Frontera University. -

Degrading Bacteria in Deep‐
View metadata, citation and similar papers at core.ac.uk brought to you by CORE provided by Aberdeen University Research Archive Journal of Applied Microbiology ISSN 1364-5072 ORIGINAL ARTICLE Hydrocarbon-degrading bacteria in deep-water subarctic sediments (Faroe-Shetland Channel) E. Gontikaki1 , L.D. Potts1, J.A. Anderson2 and U. Witte1 1 School of Biological Sciences, University of Aberdeen, Aberdeen, UK 2 Surface Chemistry and Catalysis Group, Materials and Chemical Engineering, School of Engineering, University of Aberdeen, Aberdeen, UK Keywords Abstract clone libraries, Faroe-Shetland Channel, hydrocarbon degradation, isolates, marine Aims: The aim of this study was the baseline description of oil-degrading bacteria, oil spill, Oleispira, sediment. sediment bacteria along a depth transect in the Faroe-Shetland Channel (FSC) and the identification of biomarker taxa for the detection of oil contamination Correspondence in FSC sediments. Evangelia Gontikaki and Ursula Witte, School Methods and Results: Oil-degrading sediment bacteria from 135, 500 and of Biological Sciences, University of Aberdeen, 1000 m were enriched in cultures with crude oil as the sole carbon source (at Aberdeen, UK. ° E-mails: [email protected] and 12, 5 and 0 C respectively). The enriched communities were studied using [email protected] culture-dependent and culture-independent (clone libraries) techniques. Isolated bacterial strains were tested for hydrocarbon degradation capability. 2017/2412: received 8 December 2017, Bacterial isolates included well-known oil-degrading taxa and several that are revised 16 May 2018 and accepted 18 June reported in that capacity for the first time (Sulfitobacter, Ahrensia, Belliella, 2018 Chryseobacterium). The orders Oceanospirillales and Alteromonadales dominated clone libraries in all stations but significant differences occurred at doi:10.1111/jam.14030 genus level particularly between the shallow and the deep, cold-water stations. -

Ecological Drivers of Bacterial Community Assembly in Synthetic Phycospheres
Ecological drivers of bacterial community assembly in synthetic phycospheres He Fua, Mario Uchimiyaa,b, Jeff Gorec, and Mary Ann Morana,1 aDepartment of Marine Sciences, University of Georgia, Athens, GA 30602; bComplex Carbohydrate Research Center, University of Georgia, Athens, GA 30602; and cDepartment of Physics, Massachusetts Institute of Technology, Cambridge, MA 02139 Edited by Edward F. DeLong, University of Hawaii at Manoa, Honolulu, HI, and approved January 6, 2020 (received for review October 3, 2019) In the nutrient-rich region surrounding marine phytoplankton The ecological mechanisms that influence the assembly of cells, heterotrophic bacterioplankton transform a major fraction of phycosphere microbiomes are not well understood, however, in recently fixed carbon through the uptake and catabolism of part because of the micrometer scale at which bacterial commu- phytoplankton metabolites. We sought to understand the rules by nities congregate. It remains unclear whether simple rules exist which marine bacterial communities assemble in these nutrient- that could predict the composition of these communities. enhanced phycospheres, specifically addressing the role of host Phycospheres are short-lived in the ocean, constrained by the resources in driving community coalescence. Synthetic systems with 1- to 2-d average life span of phytoplankton cells (20, 21). The varying combinations of known exometabolites of marine phyto- phycosphere bacterial communities must therefore form and dis- plankton were inoculated with seawater bacterial assemblages, and perse rapidly within a highly dynamic metabolite landscape (14). communities were transferred daily to mimic the average duration We hypothesized a simple rule for assembly in metabolically di- of natural phycospheres. We found that bacterial community verse phycospheres in which communities congregate as the sum assembly was predictable from linear combinations of the taxa of discrete metabolite guilds (22). -

Investigation of the Microbial Communities Associated with the Octocorals Erythropodium
Investigation of the Microbial Communities Associated with the Octocorals Erythropodium caribaeorum and Antillogorgia elisabethae, and Identification of Secondary Metabolites Produced by Octocoral Associated Cultivated Bacteria. By Erin Patricia Barbara McCauley A Thesis Submitted to the Graduate Faculty in Partial Fulfillment of the Requirements for a Degree of • Doctor of Philosophy Department of Biomedical Sciences Faculty of Veterinary Medicine University of Prince Edward Island Charlottetown, P.E.I. April 2017 © 2017, McCauley THESIS/DISSERTATION NON-EXCLUSIVE LICENSE Family Name: McCauley . Given Name, Middle Name (if applicable): Erin Patricia Barbara Full Name of University: University of Prince Edward Island . Faculty, Department, School: Department of Biomedical Sciences, Atlantic Veterinary College Degree for which Date Degree Awarded: , thesis/dissertation was presented: April 3rd, 2017 Doctor of Philosophy Thesis/dissertation Title: Investigation of the Microbial Communities Associated with the Octocorals Erythropodium caribaeorum and Antillogorgia elisabethae, and Identification of Secondary Metabolites Produced by Octocoral Associated Cultivated Bacteria. *Date of Birth. May 4th, 1983 In consideration of my University making my thesis/dissertation available to interested persons, I, :Erin Patricia McCauley hereby grant a non-exclusive, for the full term of copyright protection, license to my University, The University of Prince Edward Island: to archive, preserve, produce, reproduce, publish, communicate, convert into a,riv format, and to make available in print or online by telecommunication to the public for non-commercial purposes; to sub-license to Library and Archives Canada any of the acts mentioned in paragraph (a). I undertake to submit my thesis/dissertation, through my University, to Library and Archives Canada. Any abstract submitted with the . -

Marinomonas Brasiliensis Sp
IJSEM Papers in Press. Published June 18, 2010 as doi:10.1099/ijs.0.024661-0 1 Marinomonas brasilensis sp. nov. isolated from the coral Mussismilia 2 hispida and reclassification of Marinomonas basaltis as a later synonym 3 of Marinomonas communis 4 5 Luciane A. Chimetto1,2,3, Ilse Cleenwerck3, Marcelo Brocchi1, Anne Willems4, 6 Paul De Vos3,4, and Fabiano L. Thompson2* 7 8 1Departament of Genetics, Evolution and Bioagents, Institute of Biology, State University of 9 Campinas (UNICAMP), Brazil 10 11 2Department of Genetics, Institute of Biology, Federal University of Rio de Janeiro (UFRJ), 12 Brazil 13 14 3BCCM/LMG Bacteria Collection, Ghent University, K. L. Ledeganckstraat 35, B-9000 Ghent, 15 Belgium 16 17 4Laboratory of Microbiology, Faculty of Sciences, Ghent University, K. L. Ledeganckstraat 35, B- 18 9000 Ghent, Belgium 19 20 *Corresponding author: Fabiano L. Thompson. Phone/FAX: +55-21-25626567. 21 E-mail: [email protected] 22 23 Running Title: 24 Marinomonas brasilensis sp. nov. 25 Subject Category: New taxa, Proteobacteria 26 27 Footnote: The GenBank/EMBL accession number for the 16S rRNA gene 28 sequence of strain R-40503T is GU929940. 29 30 31 32 33 34 35 36 37 38 39 40 1 41 Abstract 42 A Gram-negative, aerobic bacterium, designated R-40503T was isolated from 43 mucus of the reef builder coral, Mussismilia hispida, located in the São 44 Sebastião Channel, São Paulo, Brazil. Phylogenetic analyses revealed that 45 strain R-40503T belongs to the genus Marinomonas. The 16S rRNA gene 46 sequence similarity of R-40503T was above 97 % with the type strains of 47 Marinomonas vaga, M.